Формула полінуклеотидної вакцини проти дихальних патологій та патологій розмноження свиней
Номер патенту: 91675
Опубліковано: 25.08.2010
Автори: Одонне Жан-Крістоф Франсіс, Рівьєр Мішель, Бушардон Аннабель, Бодю Філіпп
Формула / Реферат
1. Вакцина свиней, що включає плазміду (плазміди), які містять і забезпечують експресію in vivo в клітинах свині-хазяїна послідовності (послідовностей) нуклеїнової кислоти з пестивірусу, що кодує білок (білки), вибрані з групи, що складається з Е1, Е2 і Е1 і Е2, і фармацевтично прийнятний носій.
2. Вакцина за п. 1, в якій плазміда містить послідовність нуклеїнової кислоти, що кодує білок Е1.
3. Вакцина за п. 1, в якій плазміда містить послідовність нуклеїнової кислоти, що кодує білок Е2.
4. Вакцина за п. 1, в якій плазміда містить послідовності нуклеїнової кислоти, що кодує обидва білки Е1 і Е2.
5. Вакцина за п. 1, яка включає першу плазміду, що містить послідовність нуклеїнової кислоти, що кодує білок Е1, і другу плазміду, що містить послідовність нуклеїнової кислоти, що кодує білок Е2.
6. Вакцина за п. 1, в якій експресія послідовності (послідовностей) нуклеїнових кислот знаходиться під контролем промотора, вибраного з групи, що складається з промотора CMV-IE, раннього промотора SV40, пізнього промотора SV40, промотора LTR вірусу саркоми Рауса і промотора гена цитоскелета.
7. Вакцина за п. 6, в якій промотором є промотор CMV-IE.
8. Спосіб вакцинації свиней, що включає: введення вказаній свині вакцини, вибраної з групи, що складається з цільної живої вакцини, цільної інактивованої вакцини, субодиничної вакцини і рекомбінантної вакцини; і далі введення вказаній свині вакцини за п. 1.
9. Спосіб вакцинації свиней, що включає введення вказаній свині вакцини за п. 1.
10. Набір, що включає (і) вакцину за п. 1 і (іі) свинячу вакцину, вибрану з групи, що складається з цільної живої вакцини, цільної інактивованої вакцини, субодиничної вакцини і рекомбінантної вакцини.
Текст
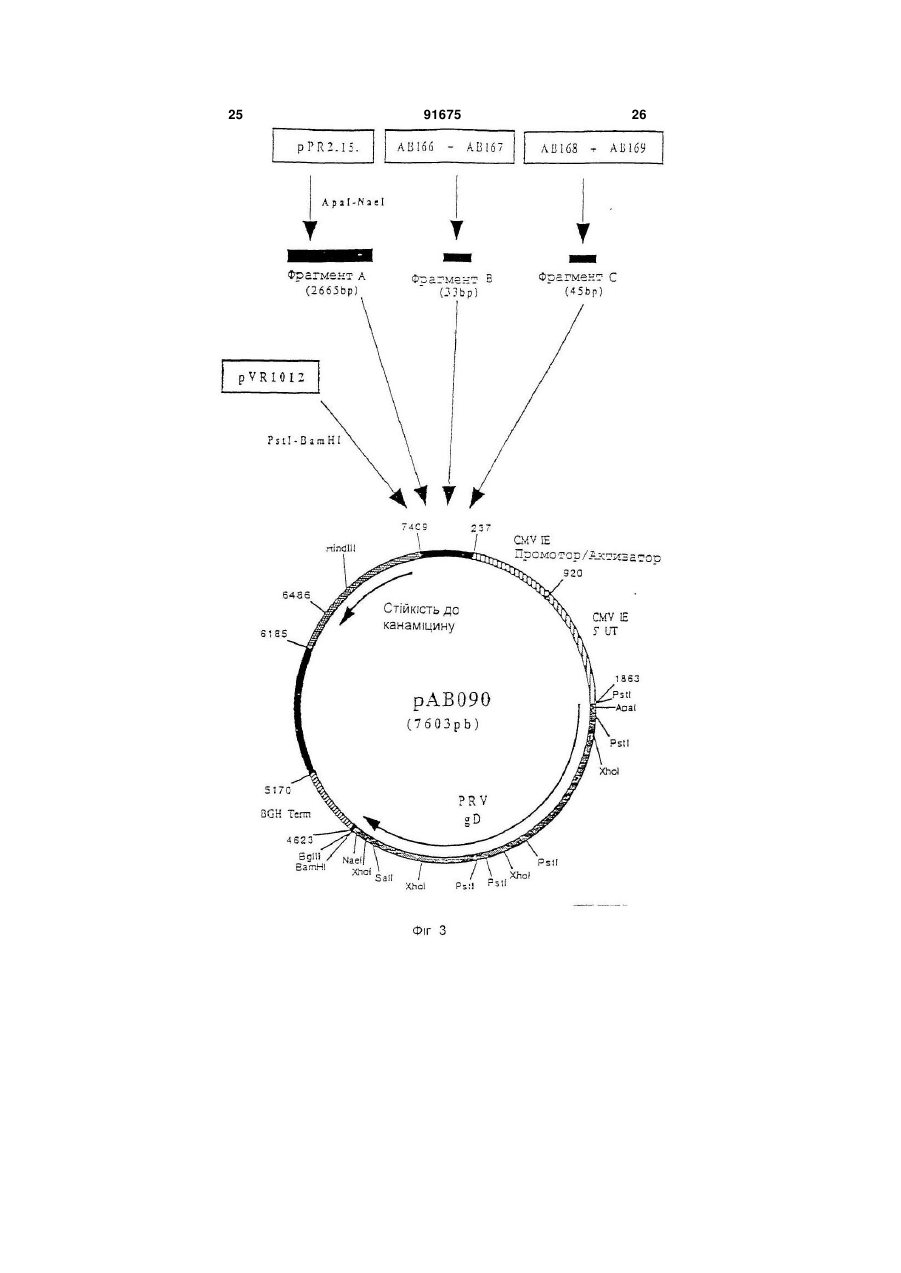
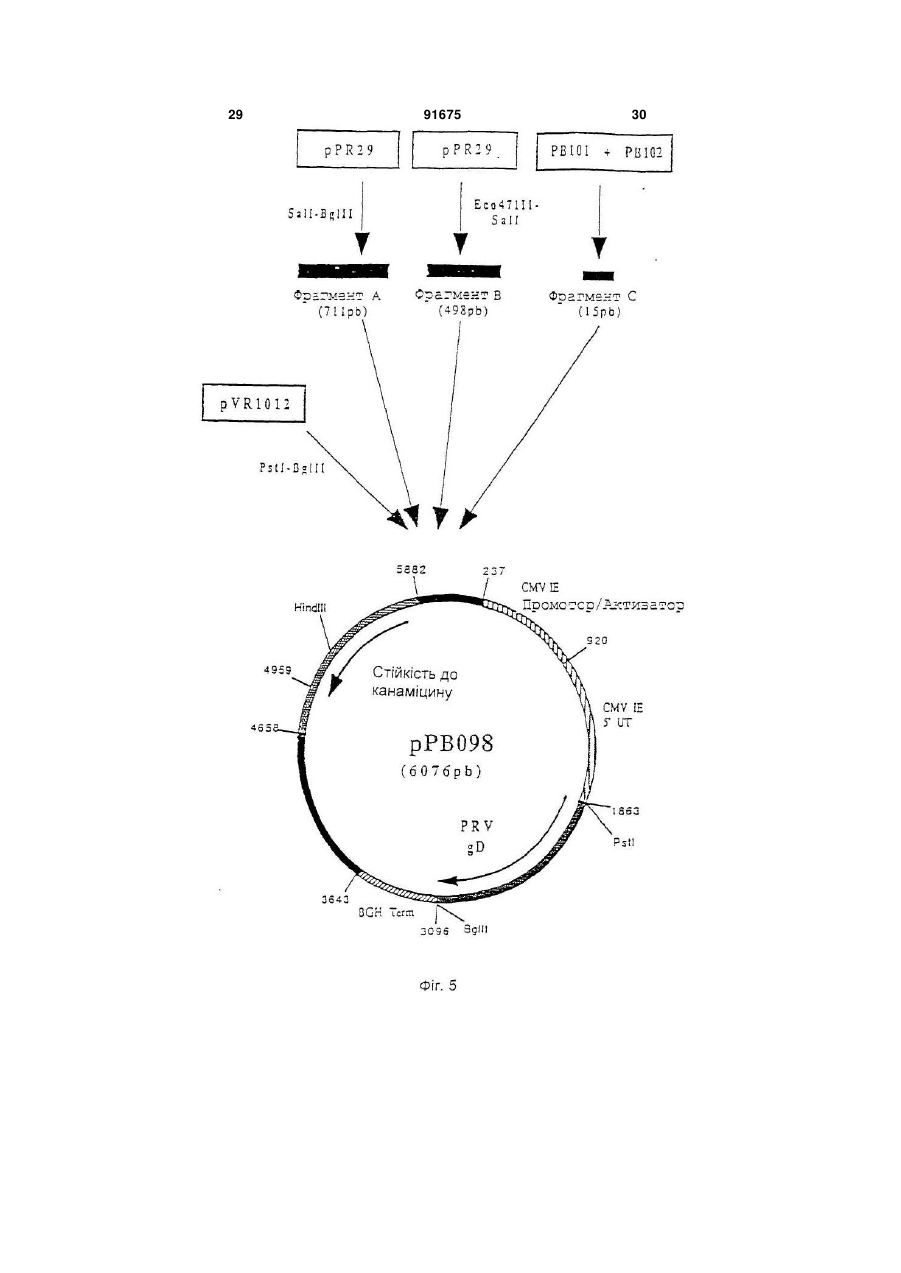
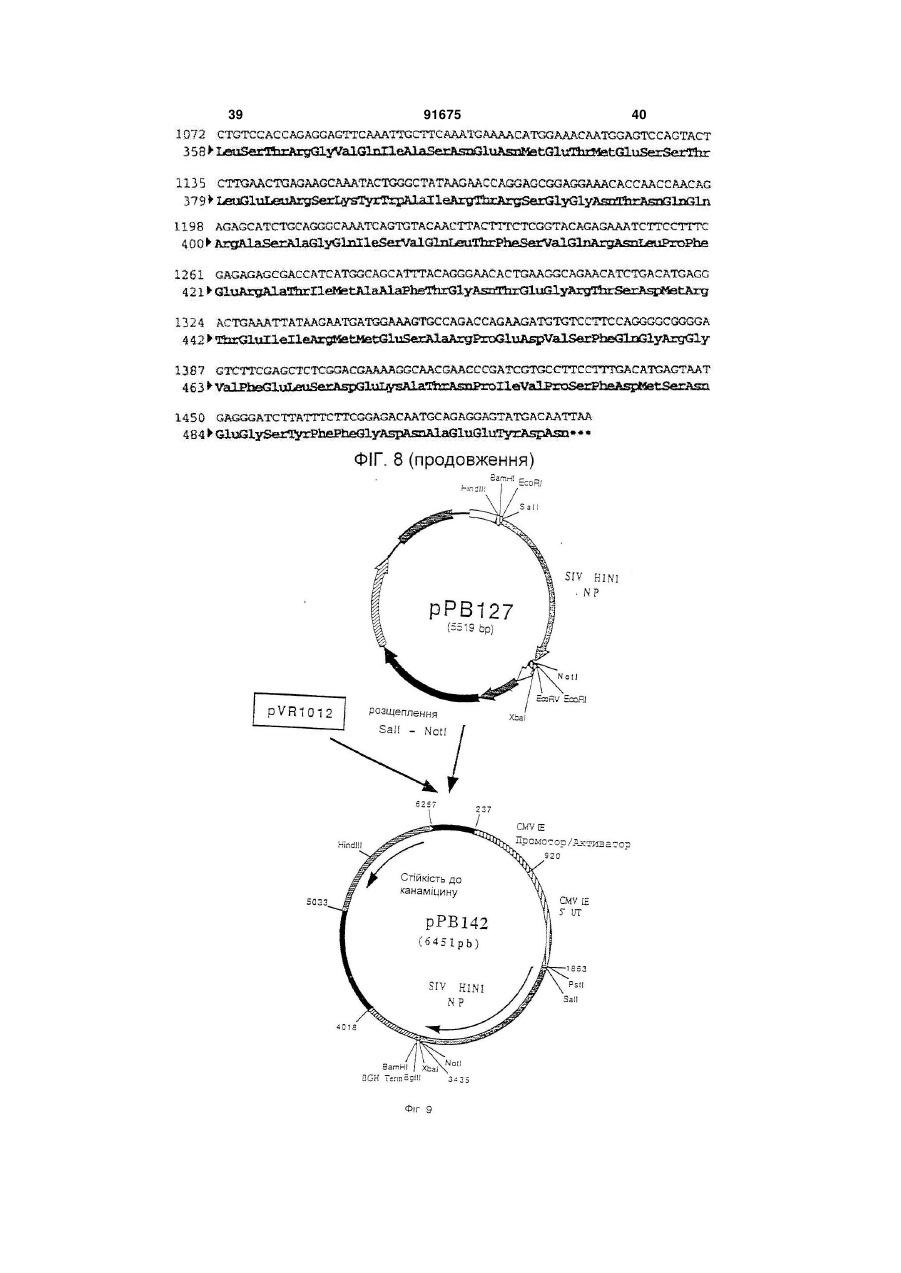
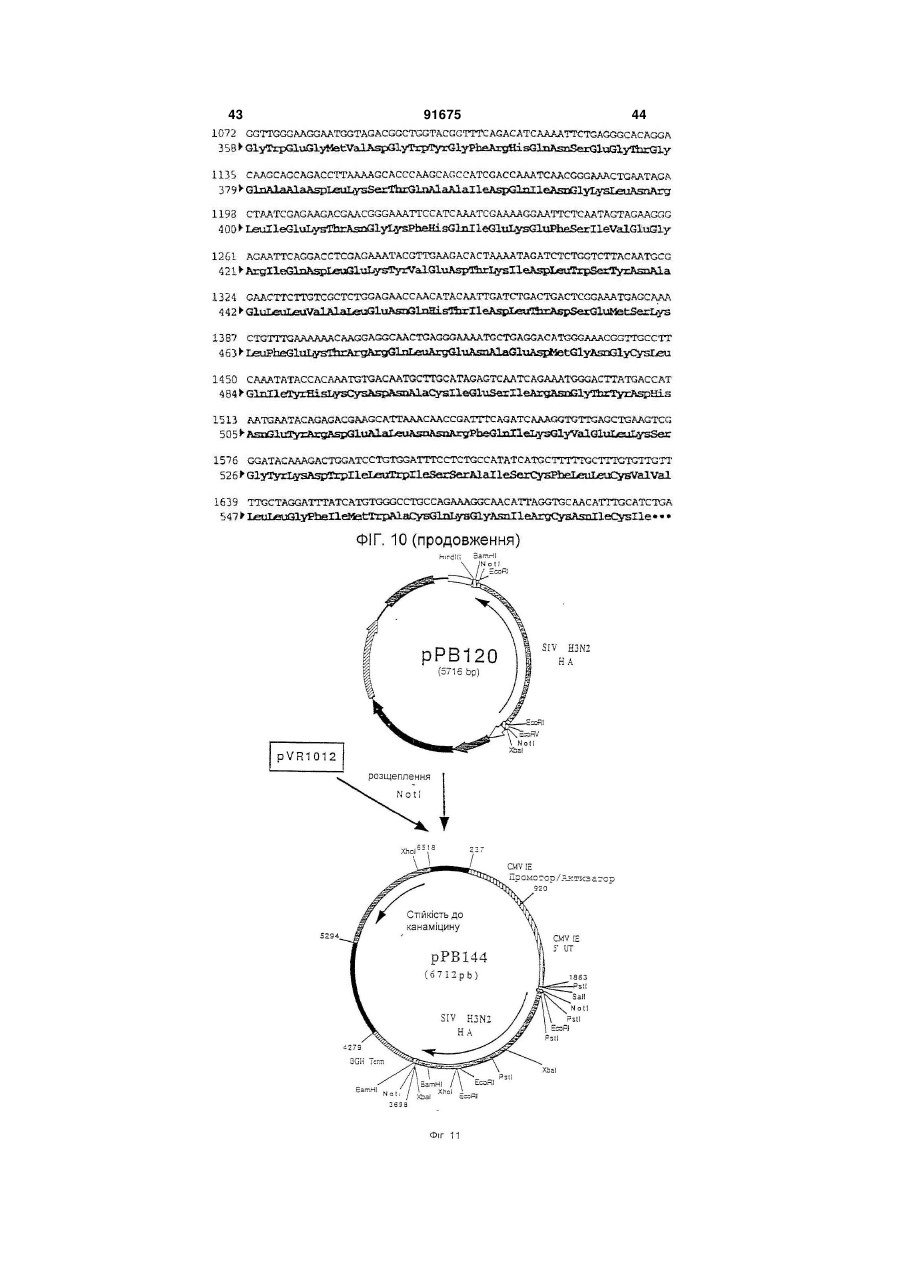
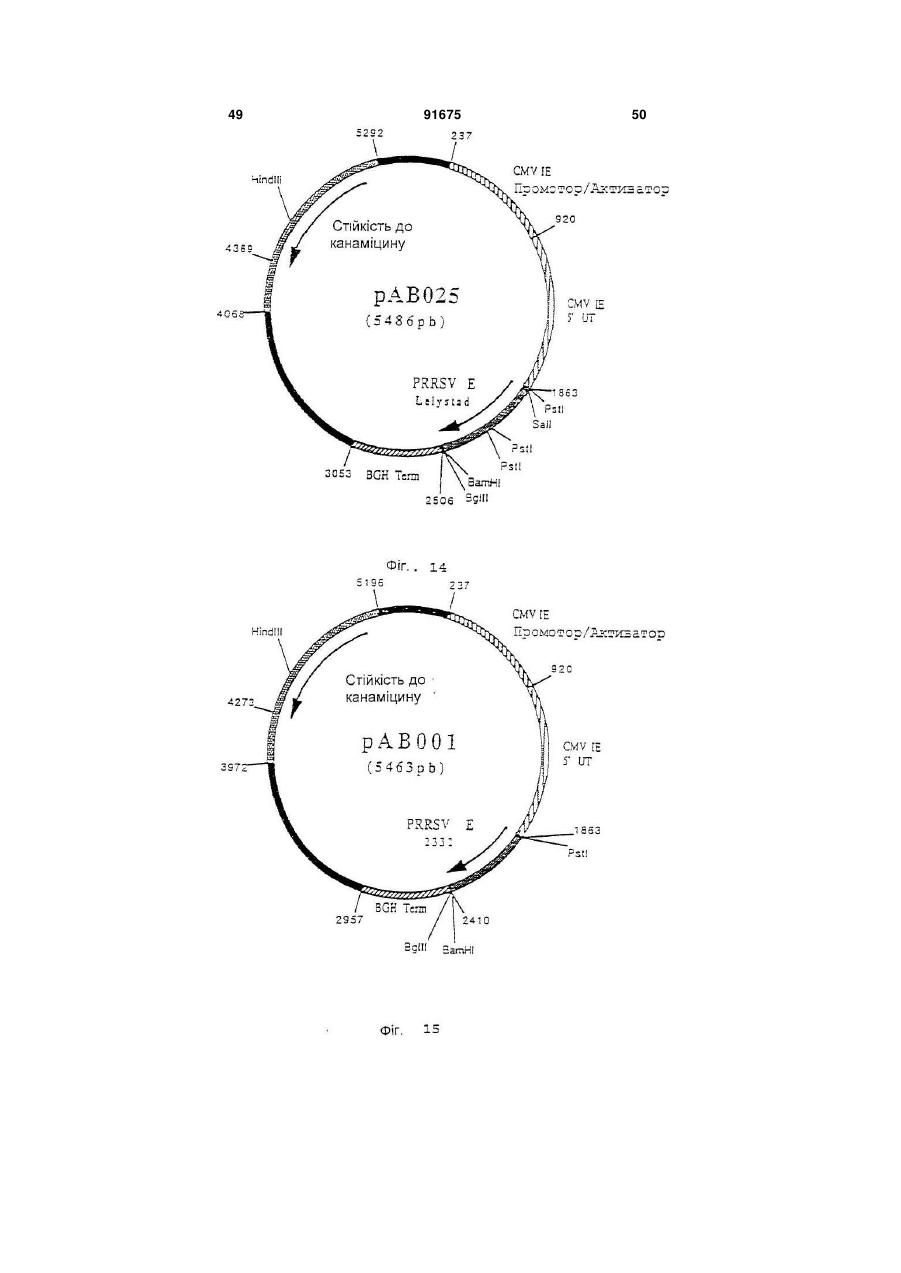
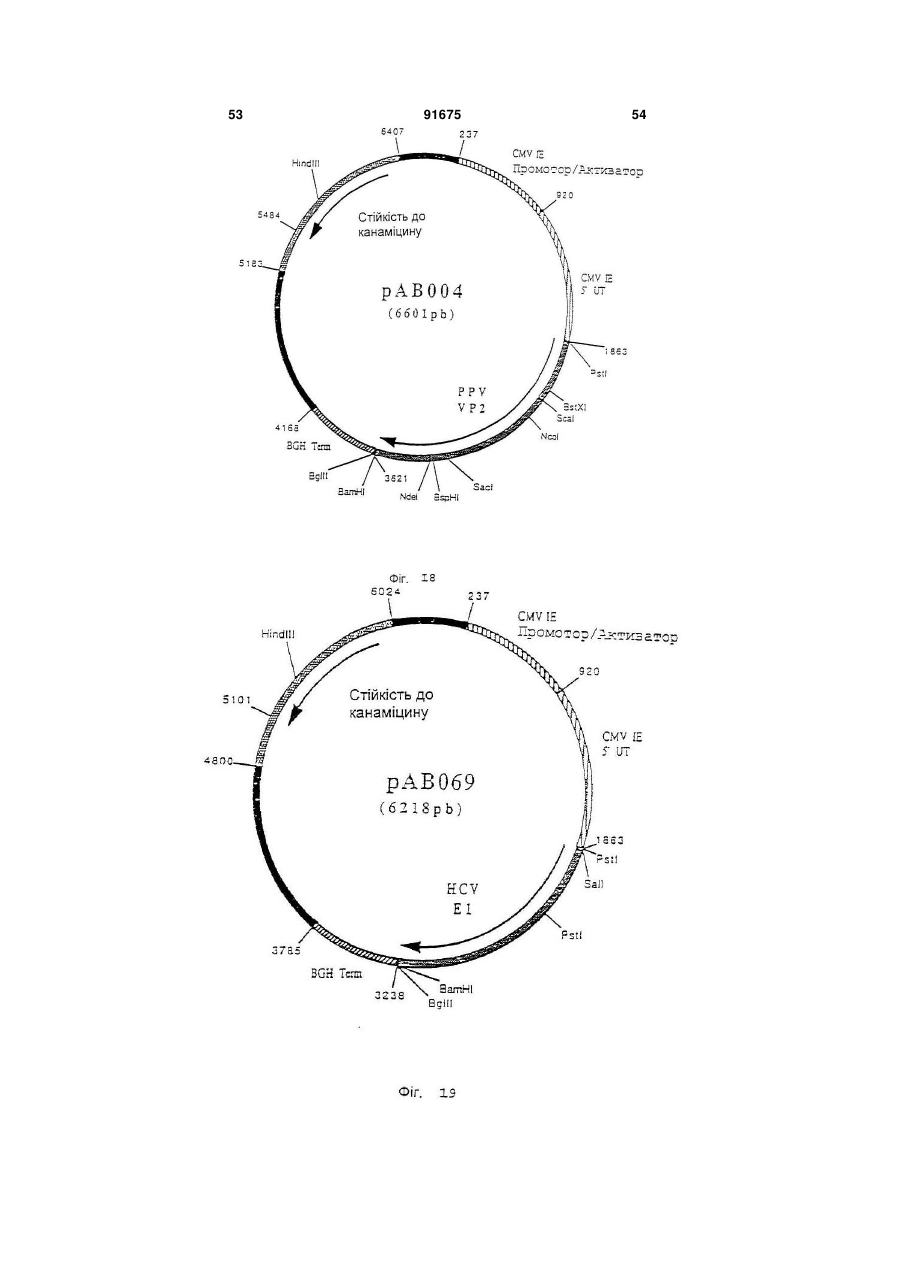
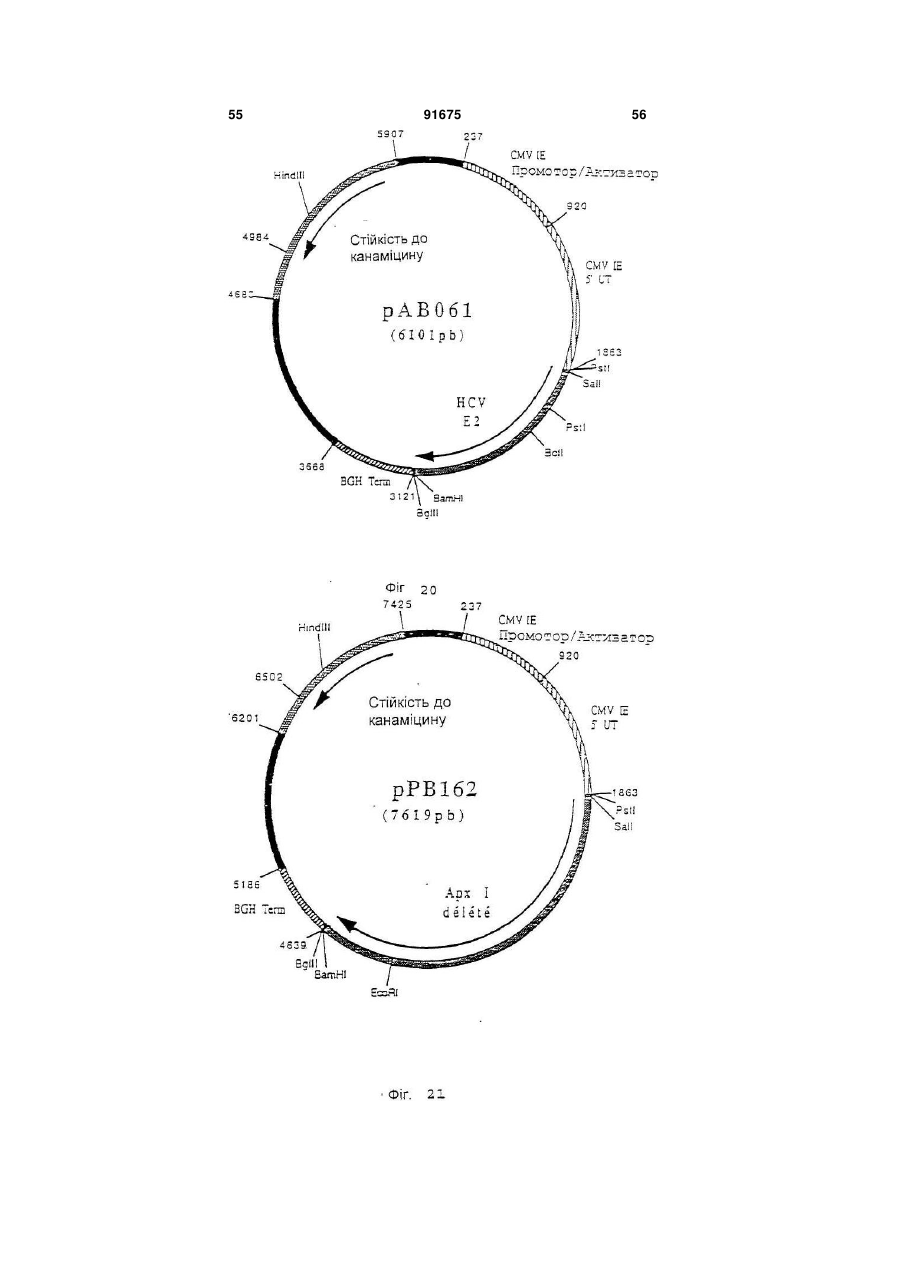
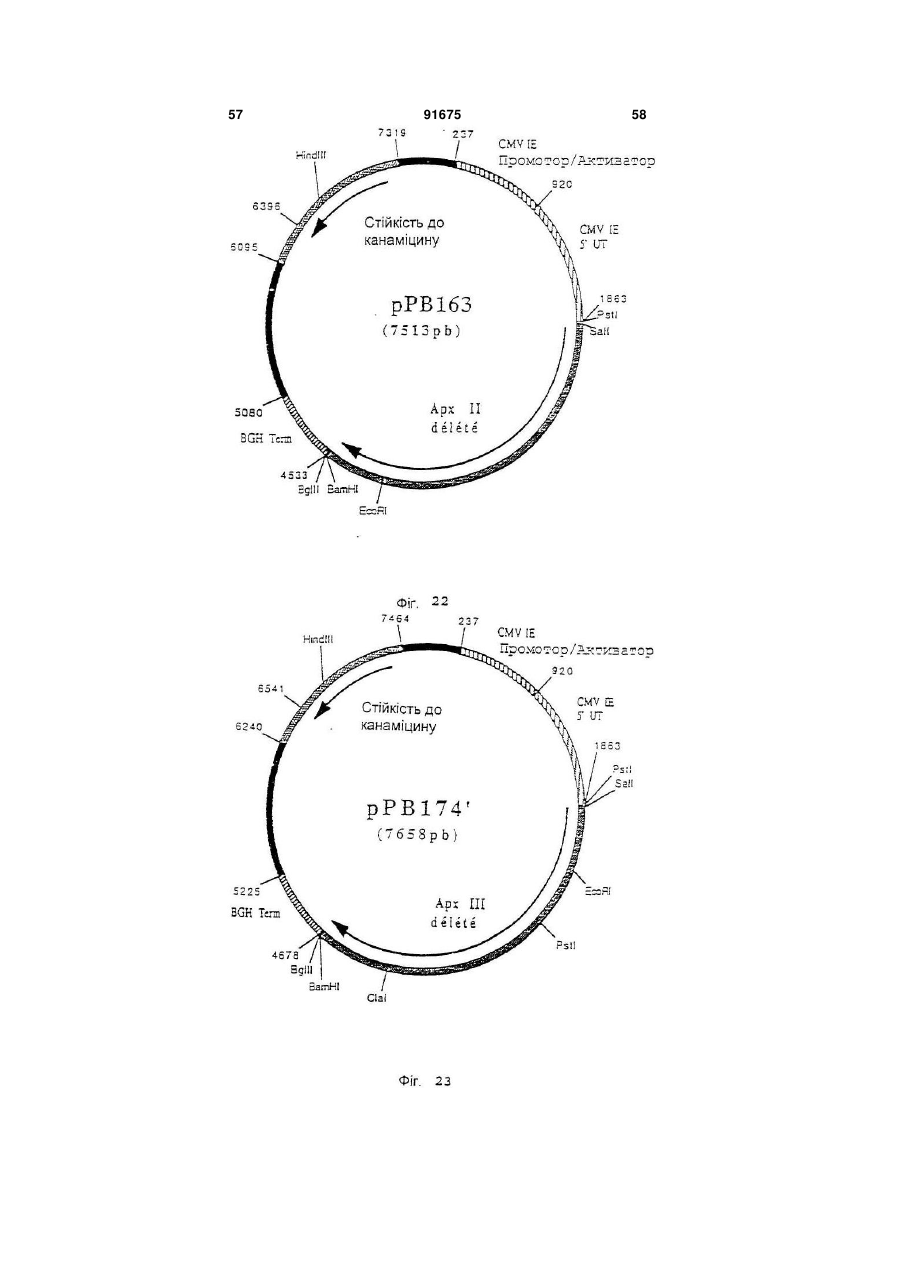
1. Вакцина свиней, що включає плазміду (плазміди), які містять і забезпечують експресію in vivo в клітинах свині-хазяїна послідовності (послідовностей) нуклеїнової кислоти з пестивірусу, що кодує білок (білки), вибрані з групи, що складається з Е1, Е2 і Е1 і Е2, і фармацевтично прийнятний носій. 2. Вакцина за п. 1, в якій плазміда містить послідовність нуклеїнової кислоти, що кодує білок Е1. 3. Вакцина за п. 1, в якій плазміда містить послідовність нуклеїнової кислоти, що кодує білок Е2. 4. Вакцина за п. 1, в якій плазміда містить послідовності нуклеїнової кислоти, що кодує обидва білки Е1 і Е2. C2 2 91675 1 3 91675 4 Pleuropneumoniae. Смертність можлива у випадку M-F Le Potier і ін. (Second International A.Pleuropneumoniae, HCV і PRV. Symposium on the Eradication of Aujeszky's Disease Крім цього, у респіраторному комплексі свиней (pseudo rabies) Virus August 6th tu 8th 1995 дуже важливе значення мають взаємодії між мікCopenhagen, Denmark) і Μ. Monteil і ін. (Наукові дні роорганізмами. Дійсно, велика частина патогенних Кафедри Патології тварин, INRA-ENV, Ліонськая бактерій складає звичайну мікрофлору носоглотДержавна Ветеренарна школа. 13-14 грудня 1994, кових зон і мигдалин молодої тварини. Ці патогени Ліон, Франція) здійснили спробу вакцинації свиней виділяються свиноматкою і часто вдихаються попроти вірусу хвороби Aujeszky за допомогою пларосятами в перші години життя, коли колостральзміди, що забезпечує експресію гена gD під контний імунітет ще не ефективний. Організми, що ролем сильного промотору, пізнього мажорного живуть у верхніх відділах дихальних шляхів мопромотору аденовірусу типу 2. Незважаючи на жуть поширюватися в нижні відділи, якщо захисні хороший рівень антитільної відповіді, не було вимеханізми дихальної системи ослаблені впливом явлено встановлення захисту. Однак, гарні репопереднього агента, такого як Actinobacillus зультати у відношенні захисту були зафіксовані pieuropneumoniae або віруси. Інвазія в легені може після зараження свиней рекомбінантним аденовібути дуже швидкою, зокрема, якщо попередніми русом, в який був вбудований ген gD і той же пропатогенами є такі, як Actinobacillus мотор, що доводить, що наявність глікопротеїну pieuropneumoniae, що виділяють сильні цитотокgD достатньо для встановлення захисту у свиней. сини, здатні викликати ушкодження війок клітин Практика минулого не дала ніяких результатів дихального епітелію і альвеолярних макрофагів. у відношенні встановлення захисту у свиней метоВажливі вірусні інфекції, такі як грип, респірадом полінуклеотидної вакцинації. торні коронавірусні інфекції і вірус Aujeszky, моЗавданням винаходу є розробка формули пожуть відігравати роль у патогенезі респіраторного лівалентної вакцини, що дозволяє здійснити ваккомплексу разом із бактеріями респіраторного цинацію свиней проти певного числа патогенних тропізму і мікоплазмами. агентів, що викликають, зокрема, респіраторні паНарешті, деякі агенти впливають одночасно на тології і/ або патології репродукції. Іншою метою дихальну і на репродуктивну системи. Також мовинаходу є розробка такої формули вакцини, що жуть відбутися взаємодії в плані патології репросполучить різні валентності, при збереженні всіх дукції. Необхідно, отже, подбати про налагодженнеобхідних критеріїв сумісності і стабільності ваня ефективних превентивних методів проти лентностей. головних патогенних агентів, що викликають ресІншою метою винаходу є розробка такої форпіраторні патології репродукції свиней. мули вакцини, що дозволяє сполучити різні валенСполучення, які розроблені до цього часу, бутності в одному носії. Іншою метою винаходу є ли одержані на основі інактивованих вакцин або розробка такої формули вакцини, яка була б просживих вакцин і, у деяких випадках, сумішей таких тою в застосуванні і не була б дорогою. вакцин. При їхньому застосуванні виникають проЩе одною метою винаходу є розробка формублем сумісності валентностей і стабільності. Так, ли вакцини і методики вакцинації свиней, яка допотрібно забезпечити одночасно сумісність різних зволяє домогтися встановлення високоефективновалентностей вакцини, як по відношенню різних го довгострокового захисту, у т.ч. полівалентного, антигенів, що використовуються, так і по віднопри нешкідливості і відсутності відходів. Таким шенню самих сумішей, зокрема, якщо використочином, об'єктом винаходу є формула вакцини, вують разом інактивовані вакцини і живі вакцини. зокрема, проти респіраторних патологій і/ або паТакож виникає проблема зберігання таких комбітологій репродукції свиней, що включає щонайменованих вакцин і їхньої нешкідливості, зокрема, у нше 3 валентності полінуклеотидної вакцини, кожприсутності добавок. Ці вакцини звичайно досить на з яких включає плазміду, що містить і дорогі. У заявках WO-A-9011092, WO-A-9319183, (забезпечує його експресію in vivo в клітинах хазяWO-A-9421997 і WO-A-9520660 описане викорисїна) ген однієї валентності патогена свиней, притання нещодавно розробленої технології полінукчому ці валентності обирають із групи, представлеотидних вакцин. Відомо, що в цих вакцинах виленої вірусом хвороби Aujeszky (вірус PRV або користовують плазміду, що забезпечує в клітинах псевдосказу), вірусом грипу свиней (SIV), вірусом хазяїна експресію вбудованого антигена. Були таємничої хвороби свиней (вірус PRRS), вірусом запропоновані всі шляхи введення (внутрішньобпарвовірозу (вірус PRV), вірусом класичної чуми рюшинний, внутрішньовенний, внутрішньом'язосвиней (вірус HCV або Hog Cholera virus) і бактерівий, підшкірний, внутрішньошкірний, через слизову єю, що викликає актинобацильоз і т.д.). Також можуть бути використані різні засоби (A.pleuropneumoniae), а плазміди містять для кожвакцинації, такі як ДНК, що вміщена на поверхню ної валентності один або декілька генів, обраних із часток золота і разпорошується таким чином, що групи, поданої gВ і gD для вірусу хвороби вона проникає в шкіру тварини (Tang і ін., Nature Aujeszky, НА, NP, N для вірусу грипу свиней, ORFS 356, 152-154, 1992), і рідиннострумінні упорскувачі, (Е), ORF3, ORF 6 (М) для вірусу PRRS, VP2 для що дозволяють здійснити трансфекцію одночасно вірусу парвовірозу. El, E2 для вірусу класичної в шкіру, у м'язи, у жирові тканини і у тканині молочуми свиней і архI, архII і архІІІ для чної залози (Furth і ін., Analytical Biochemistry, 205, A.pleuropneumoniae. 365-368, 1992). Під валентністю в рамках цього винаходу роУ полінуклеотидних вакцинах можна викорисзуміють щонайменше один антиген, що забезпечує товувати як голі ДНК, так і ДНК, наприклад, у склазахист проти вірусу розглянутого патогена, причоді ліпосом і катіонних ліпідів. му валентність може містити в якості підвалентно 5 91675 6 сті один або декілька змінених природних генів ти один із трьох вище названих генів або комбінаодного або декількох штамів розглянутого патогецію 2 або 3 із цих генів, у різних плазмідах або в на. Під геном патогенного агенту розуміють не складі змішаних плазмід, щоб забезпечити захист тільки повний ген, але і різні нуклеотидні послідовпроти різних серотипів A.PIeuropheumoniae. В анності, включаючи фрагменти, що зберігають здаттигенах арх І, II і III можна змінити кодуючі посліність індукувати захисну відповідь. довності, щоб одержати нетоксичні антигени, зокУ поняття гена входять нуклеотидні послідоврема, як зазначено в прикладах. ності, еквівалентні в точності описаним у приклаОб'єм дози формули вакцини за винаходом дах, тобто різні, але не кодуючі один і той самий може складати, у цілому, від 0,1 до 10 мл, і зокрепротеїн послідовності. У нього також входять нукма, від 1 до 5 мл для вакцинації при внутрішньолеотидні послідовності інших штамів розглянутого м'язовому введенні. патогена, що забезпечують перехресний захист Доза складає від 10 нг до 1 мг, краще, від 100 або специфічний захист від штаму або групи штанг до 500 мкг, ще краще - від 1 мкг до 250 мкг на мів. В нього також входять нуклеотидні послідовкожний тип плазміди. ності, що були змінені для полегшення експресії in Використовують, краще, голі плазміди, просто vivo в організмі тваринного хазяїна, але кодуючі вміщені у вакцинуючий носій, яким звичайно є фітой же протеїн. зіологічний розчин (0,9% NaCI ), ультрачиста вода, Краще, формула вакцини за винаходом вклюбуфер ТЕ і т.п. Можна використовувати, зрозуміло, чає валентності Aujeszky і грипу свиней, до яких будь-які форми полінуклеотидних вакцин, описані можуть бути додані інші валентності, обрані, перев практиці минулого. важно, із валентностей PRRS і A.pieuropneumoniae Кожна плазміда містить промотор, здатний за(актинобацильоз). Можна додати інші валентності, безпечити експресію залежного від нього гена в обрані з валентностей парвовірозу і класичної чуклітинах хазяїна. Таким промотором є сильний ми свиней. еукаріотичний промотор, і зокрема, ранній промоМожливі, зрозуміло, усі комбінації валентностор цитомегаловірусу CMV-IE, що походить від тей. Однак, у рамках винаходу, перевагу віддають людини або миші, або, можливо, від іншої тваривалентностям Aujeszky і грипу свиней, потім PRRS ни, такої як пацюк, свиня, морська свинка. і A.PIeuropheumoniae. Для вакцинації, спрямованої Загалом, промотор може бути як вірусного, так більш конкретно проти респіраторних патологій у і клітинного походження. У якості вірусного промосвиней, перевагу при виборі валентностей віддатора можна назвати ранній або пізній промотор ють Aujeszky, грипу свиней, PRRS і актинобацивірусу SV 40 або промотор LTR вірусу Саркоми льозу. Руса. Це може бути також промотор вірусу, від Для вакцинації, спрямованої конкретно проти якого походить ген, наприклад, власний промотор патологій репродукції, валентності вибирають, гена. переважно, із PRRS, парвовірозу, класичної чуми У якості клітинного промотору можна назвати свиней і Aujeszky. У відношенні валентності промотор гена цитоскелету, наприклад, промотор Aujeszky можна використовувати один із генів gВ і десміну (Bolmont і ін., Journal of Submicroscopic gD. Краще, використовують обидва гени, які у Cytology and Patholigy, 1990, 22, 117-122; і цьому випадку, вбудовують у різні плазміди або в ZHENLIN і ін., GENE, 1989, 78, 243-254) або проодну плазміду. мотор актину. Якщо одна плазміда містить декільУ відношенні валентності грипу свиней, викока генів, вони можуть знаходитися в одній одиниці ристовують, переважно, гени НА і NP. Можна витранскрипції або в двох різних одиницях. Комбінакористовувати один із цих двох генів або обидва ція різних валентностей вакцини за винаходом гени одночасно, вбудовані в різні плазміди або в може бути отримана, переважно, змішуванням одну плазміду. Переважно, в одній вакцині сполуполінуклеотидних плазмід, що забезпечують ексчають послідовності НА більше одного штаму віпресію одного або декількох антигенів кожної варусу грипу, зокрема, штамів, що зустрічаються в лентності, але можна також здійснити експресію даній місцевості. На відміну від цього, NP забезпеантигенів декількох валентностей за допомогою чує перехресний захист і, отже, можна обмежитися однієї плазміди. використанням послідовності одного єдиного штаЩе одним об'єктом винаходу є формули одному вірусу. валентної вакцини, що включають одну або декіУ відношенні валентності PRRS використовулька плазмід, що кодують один або декілька генів ють, переважно, гени Ε і ORF3 або, також, М. Моодного з вірусів, обраних із групи, поданої PRV, жна використовувати ці гени окремо або в комбіPRRS, PPV, HCV і A.PIeuropneumoniae, причому нації; у випадку комбінації, гени можна гени такі, як описано вище. За винятком одновавбудовувати в окремі плазміди або в плазміди, що лентного характеру, ці формули можуть мати викомбінують два або три з цих генів. Вигідно викоще перераховані характеристики в тому, що стористовувати в одній вакцині гени, що походять від сується вибору генів, їхніх комбінацій, композицій принаймні двох штамів, зокрема, від одного європлазмід, об'ємів доз, доз і т.д. Формули одновалепейського штаму і від одного американського штантної вакцини можуть бути використані (і) для одему. ржання формули полівалентної вакцини, як вона У відношенні валентності класичної чуми свиописана вище, (іі) в індивідуальному порядку, проней можна використовувати один із генів Е1 і Е2 ти конкретної патології, (ііі) в сполученні з вакциабо гени Е1 і Е2 разом. В двох різних плазмідах ною іншого типу (суцільної живої або інактивоваабо, можливо, в одній плазміді. У відношенні ваної, рекомбінантної, субодиничної), проти іншої лентності актинобацильозу, можна використовувапатології або (iv) у якості вторинної вакцини після 7 91675 8 вакцини, описаної нижче. Дійсно, ще одним об'єксубодиничну вакцину таким чином, щоб забезпетом цього винаходу є використання однієї або дечити первинну вакцинацію, і, через, переважно, 2-6 кількох плазмід за винаходом для одержання вактижнів, вводять полівалентну або одновалентну цини, призначеної для вакцинації свиней, вакцину за винаходом. первинновакцинованих за допомогою першої, звиВинахід також стосується методики одержання чайної, вакцини з числа тих, що використовувалиформул вакцини, а саме, до одержання валентнося в практиці минулого, а саме, обраної з групи, стей і їхніх сумішей, як випливає з даного опису. представленої цілісною живою вакциною, цілісною Далі іде більш детальний опис винаходу за інактивованою вакциною, субодиничною вакцидопомогою способів здійснення винаходу з посиною, рекомбінантною вакциною, причому ця перша ланням на малюнки додатку. вакцина (одновалентна або полівалентна) несе Список фігур (тобто містить або забезпечує його експресію) Фігура №1: Плазміда pVR1012 один або декілька антигенів, що кодуються однією Фігура №2: Послідовність гена PRV gB (штам або декількома використовуваними плазмідами, NIA3) або антигенів, що забезпечують перехресний заФігура №3: Конструкція плазміди рАВ090 хист. Заслуговує на увагу той факт, що полінуклеФігура №4: Послідовність гена PRV g (штам отидна вакцина спричиняє сильну дію в якості втоNIA3) ринної вакцини, що виявляється в посилені імунної Фігура №5: Конструкція плазміди рРВ098 відповіді і у встановленні довгострокового імунітеФігура №6: Послідовність гена грип свиней НА ту. (штам Н1 N1) В цілому, вакцини для первинної вакцинації Фігура №7: Конструкція плазміди рРВ143 можуть бути обрані з числа вакцин, запропоноваФігура №8: Послідовність гена грип свиней NP них для продажу різними виробниками ветеринар(штам Н1 N1) них вакцин. Ще одним об'єктом винаходу є набір Фігура №9: Конструкція плазміди рРВ142 для вакцинації, у який входять вакцина для перФігура №10: Послідовність гена грип свиней винної вакцинації, як вона описана вище, і формуНА (штам Н3 N2) ла вакцини за винаходом для використання в якоФігура №11: Конструкція плазміди рРВ144 сті вторинної вакцини. Винахід також стосується Фігура №12: Послідовність гена грип свиней формули вакцини за винаходом з прикладеною до NP (штам Н3 N2) неї інструкцією, у якій зазначене використання цієї Фігура №13: Конструкція плазміди рРВ132 формули для вторинної вакцинації після первинної Фігура №14: Плазміда рАВ025 вакцинації як вона описана вище. Фігура №15: Плазміда рАВ001 Також об'єктом цього винаходу є методика ваФігура №16: Плазміда рАВ091 кцинації свиней проти респіраторних патологій і/ Фігура №17: Плазміда рАВ092 або патологій репродукції свиней, що включає Фігура №18: Плазміда рАВ004 введення ефективної дози формули вакцини як Фігура №19: Плазміда рАВ069 вона описана вище. Ця методика вакцинації вклюФігура №20; Плазміда рАВ061 чає введення однієї або декількох доз формули Фігура №21: Плазміда рРВ162 вакцини, причому ці дози можуть бути введені поФігура №22: Плазміда рРВ163 слідовно через короткі проміжки часу і/ або посліФігура №23: Плазміда рРВ174 довно через тривалі проміжки часу. Фігура №24: Плазміда рРВ189 Формули вакцини за винаходом можуть бути Фігура №25: Плазміда рРВ190 введені, у рамках даної методики вакцинації, різСписок послідовностей SEQ ID No ними шляхами введення, запропонованими в SEQ ID №1: Послідовність гена PRV gB (штам практиці минулого для полінуклеотидної вакцинаNIA3) ції, і за допомогою будь-якої відомої техніки ввеSEQ ID №2: Олігонуклеотид АВ166 дення. Зокрема, можна використовувати вакцинаSEQ ID №3: Олігонуклеотид АВ167 цію внутрішньошкірним шляхом за допомогою SEQ ID №4: Олігонуклеотид АВ168 рідкострумінного, краще, багатострумінного упорсSEQ ID №5: Олігонуклеотид АВ169 кувача, і зокрема, упорскувача з упорскуючою наSEQ ID №6: Послідовність гена PRV gD (штам садкою, що має декілька отворів, зокрема, від 5 до NIA3) 6 отворів, як в апараті Pigjet, що випускається SEQ ID №7: Олігонуклеотид РВ101 компанією Endoscoptic, Laons, Франція. Об'єм дози SEQ ID №8: Олігонуклеотид РВ102 при використанні такого апарату складає, переваSEQ ID №9: Олігонуклеотид pB107 жно, від 0,1 до 0,9 мл, зокрема, від 0,2 до 0,6 мл, SEQ ID №10: Олігонуклеотид РВ108 краще, від 0,4 до 0,5 мл, причому даний об'єм моSEQ ID №11: Послідовність гена грип свиней же бути введений за один або декілька разів, пеНА (штам H1N1) реважно, за два рази. SEQ ID №12: Олігонуклеотид РВ097 Нарешті, об'єктом винаходу є методика вакциSEQ ID №13: Олігонуклеотид РВ098 нації, що полягає в тому, що проводять первинну SEQ ID №14: Послідовність гена грип свиней вакцинацію, як вона описана вище і вторинну вакNP (штам H1N1) цинацію формулою вакцини за винаходом. ВідпоSEQ ID №15: Олігонуклеотид РВ095 відно до кращої форми здійснення способу за виSEQ ID №16: Олігонуклеотид РВ096 находом, спочатку тварині вводять ефективну SEQ ID №17: Послідовність гена грип свиней дозу вакцини класичного типу, зокрема, інактивоНА (штам H3N2) вану, живу, ослаблену або рекомбінантну, або 9 91675 10 SEQ ID №18: Послідовність гена грип свиней присутності натрійдодецілсульфату (SDS) (0,5% NP (штам H3N2) кінц.) протягом 2 годин при 37°С. Потім вірусну SEQ ID №19: Олігонуклеотид АВ055 ДНК екстрагують сумішшю фенол/хлороформ, SEQ ID №20: Олігонуклеотид АВ056 потім преципітують 2 об'ємами абсолютного етаSEQ ID №21: Олігонуклеотид АВ001 нолу. Через ніч при -20°С, ДНК центрифугують при SEQ ID №22: Олігонуклеотид АВ002 10000 g і +4°С протягом 15 хвилин. Осаджену ДНК SEQ ID №23: Олігонуклеотид АВ170 висушують, потім збирають у мінімальному об'ємі SEQ ID №24: Олігонуклеотид АВ171 стерильної ультрачистої води. Після цього вона SEQ ID №25: Олігонуклеотид АВ172 може бути розщеплена рестрикційними ферменSEQ ID №26: Олігонуклеотид АВ173 тами. SEQ ID №27: Олігонуклеотид АВ007 Приклад 4: Ізолювання геномних вірусних РНК SEQ ID №28: Олігонуклеотид АВО10 Віруси, що містять РНК, очищали за добре віSEQ ID №29: Олігонуклеотид АВ126 домими фахівцям технологіями. Потім ізолювали SEQ ID №30: Олігонуклеотид АВ127 РНК геному кожного вірусу, використовуючи техніSEQ ID №31: Олигонуклеотид АВ118 ку екстракції «тіоціонат гуаніSEQ ID №32: Олігонуклеотид АВ119 дію/фенолхлороформ», що описана SEQ ID №33: Олігонуклеотид РВ174 P.Chomczynski і N.Sacchi (Anal. Biochem. 1987, SEQ ID №34: Олігонуклеотид РВ189 162, 156-159). SEQ ID №35: Олігонуклеотид РВ190 Приклад 5: Технології молекулярної біології SEQ ID №36: Олігонуклеотид РВ175 Всі плазмідні конструкції були отримані з викоSEQ ID №37: Олігонуклеотид РВ176 ристанням стандартних технологій молекулярної SEQ ID №38: Олігонуклеотид РВ191 біології, описаних J. Sambrook і ін. (Molecular SEQ ID №39: Олігонуклеотид РВ192 Cloning: A Laboratory Manual. 2 видання. Cold SEQ ID №40: Олігонуклеотид РВ177 Spring Harbor Laboratory. Cold Spring Harbor New SEQ ID №41: Олігонуклеотид РВ278 York. 1989). Усі рестрикційні фрагменти, що були SEQ ID №42: Олігонуклеотид РВ279 використані в цьому винаході, ізолювали, викорисSEQ ID №43: Олігонуклеотид РВ280 товуючи набір Gene-clean (BIO 101 Inc. La Jolla, SEQ ID №44: Олігонуклеотид РВ307 CA). SEQ ID №45: Олігонуклеотид РВ303 Приклад 6: Технологія RT-PCR SEQ ID №46: Олігонуклеотид РВ306 Специфічні олігонуклеотиди ( що містять на SEQ ID №47: Олігонуклеотид РВ304 своїх 5'кінцях сайти рестрикції для полегшення SEQ ID №48: Олігонуклеотид РВ305 клонування розширених фрагментів) були синтеПриклади зовані таким чином, що вони цілком охоплюють Приклад 1: Культура вірусів кодуючі ділянки генів, які повинні бути розширені Віруси культивують на відповідній системі клі(див. Специфічні приклади). Реакцію зворотної тин до прояву цитопатичного ефекту. Системи транскрипції (RT) і розширення в ланцюзі полімеклітин, що використовуються для кожного вірусу, разою (PCR) проводили за стандартними технолодобре відомі фахівцям. Коротко кажучи, клітини, гіями (J. Sambrook (Molecular Cloning: A Laboratory чутливі до використаного вірусу, що культивуютьManual. 2 видання. Cold Spring Harbor Laboratory. ся в мінімальному необхідному середовищі Ігла Cold Spring Harbor. New York. 1989)). Кожну реак(середовище «MEM») або в іншому відповідному цію RT-PCR проводили з парою специфічних ампсередовищі, заражають досліджуваним вірусним лімерів, використовуючи в якості матриці екстраштамом, використовуючи множинність зараження говану геномну вірусну РНК. Розширену 1. Інфіковані клітини інкубують при 37°С протягом комплементарну ДНК екстрагували сумішшю фечасу, що необхідний для прояву повного цитопанол/хлороформ/ізоаміловий спирт (25:24:1) перед тичного ефекту ( у середньому 36 годин). розщепленням рестрикційнними ферментами. Приклад 2: Культура бактерій і екстракція бакПриклад 7: Плазміда pVR1012 теріальної ДНК Плазміда pVR1012 (Фігура №1) була отримана Штами Actinobacillus pleuropneumoniae культипри Vical Inc. San Diedo, CA, USA. Її конструкція вують як описано A.Rycroft і ін. (J. Gen. Microbiol. була описана в J.Hartikka і ін. (Human Gene 1991, 137, 561-568). ДНК із великою молекулярною Therapy. 1996, 7, 1205-1217). масою (хромосомна ДНК) була отримана за станПриклад 8: Конструювання плазміди рАВ090 дартними технологіями, описаними J.Sambrook і (ген PRV gB) ін. (Molecular Cloning: A Laboratory Manual. 2 виПлазміду pPR 2.15 (Μ.Riviere і ін. J.Virol. 1992. дання. Cold Spring Harbor Laboratory, Cold Spring 66. 3424-3434) розщепили за допомогою АраІ і Harbor. New York. 1989.). Nael, щоб визволити фрагмент Apal-Nael (2665 pb) Приклад 3: Екстракція геномних вірусних ДНК (фрагмент А), що містить ген, кодуючий глікопроПісля культивування, збирають зруйновані клітеїн gB вірусу хвороби Aujeszky (Штам NIA3) (Фігутки і їхні плаваючі на поверхні уламки, вірусну сура №2 і SEQ ID No 1). спензію центрифугують при 1000 g і +4°С протягом Гібридизацією наступних 2 олігонуклеотидів: 10 хвилин, щоб видалити уламки клітин. Вірусні АВ166 (33 mer) (SEQ ID №2) частинки осаджують ультрацентрифугуванням при 5'GATGCCCGCTGGTGGCGGTCTTTGGCGCG 400000 g і +4°С протягом 1 години. Осад збирають GGCC 3' у мінімальному об'ємі буферу (Tris 10 мМ, EDTA 1 AB167 (33 mer) (SEQ ID №3) мМ; рН 8,0). Цю концентровану вірусну суспензію 5'ACGTCTACGGGCGACCACCGCCAGAAACC обробляють протеїназою К (100 мкг/мл кінц.) в GCGC 3' 11 91675 12 фрагмент (33 pb), що містить послідовність гелад 7), попередньо розщепленим за допомогою на gD, від ініціального кодону ATG до сайта АраІ, EcoRVи Notl, і одержали плазміду рРВ143 (6726 був перетворений з утворенням сайта Pstl на 5' pb) (Фігура №7). (фрагмент В). Приклад 11: Конструювання плазміди рРВ142 Гібридизацією наступних 2 олігонуклеотидів: (ген Грипу свиней NP штам H1N1) АВ168 (45 mer) (SEQ ID №4) За технологією з прикладу 6 провели реакцію. 5'GGCACTACCAGCGCCTCGAGAGCGAGGAC RT-PCR із РНК геному вірусу грипу свиней (штам CCCGACGCCCTGTAGG 3' S1Y HINI «SW»), отриманої за технологією, описаAB169 (49mer) (SEQ ID №5) ною в прикладі 4, і з такими олігонуклеотидами: 5'GATCCCTACAGGGCGTCGGGGTCCTCGCT PS097 (36 mer) (SEQ ID №12) CTCGAGGCGCTGGTAGTGCC 3' 5'CCGGTCGACCGGGATAATCACTCACTGAGT фрагмент (45 pb), що містить послідовність геGACATC 3' на gD, від сайта Nael до стоп-кодона TAG був пеPB098 (33 mer) (SEQ ID №13) ретворений з утворенням сайта BamHI на 3' (фра5'TTGCGGCCGCTGTAGAAACAAGGGTATTTT гмент С). Фрагменти А, В і С разом зшили з TCT 3' вектором pVR1012 (приклад 7, попередньо розщещоб точно ізолювати ген, який кодує протеїн пленим за допомогою Pstl і BamHI, і одержали NP SIV HINI (Фігура №8 SEQ ID №14), у формі плазміду рАВ090 (7603 pb) (Фігура №3). фрагменту Sall-Notl. Після очищення, продукт RTПриклад 9: Конструювання плазміди рРВ098 PCR (1566 pb) зшили з вектором PCRII-прямої (ген PRV gD) (Інвітроген № К2000-01), щоб одержати вектор Плазміду pPR29 (Μ.Riviere і ін. J.Virol. 1992, рРВ127 (5519 pb). Вектор рРВ127 розщепили за 66, 3424-3434) розщепили за допомогою Sall і Bglll, допомогою Sall і Notl, щоб визволити фрагмент щоб визволити фрагмент Sall-Bglll (711 pb) (фрагSall-Notl (1560 pb), що містить ген NP. мент А), що містить 3'-кінець гена, що кодує глікоПотім цей фрагмент зшили з вектором протеїн gD вірусу хвороби Aujeszky (Штам NIA3) pVB.1012 (приклад 7). попередньо розщепленим (Фігура №4 і SEQ ID No 6). за допомогою Sall і Notl, і одержали плазміду Плазміду pPR29 розщепили за допомогою рРВ142 (6451 pb) (Фігура №9) Есо47ІІІ і Sall, щоб визволити фрагмент Eco47lllПриклад 12: Конструювання плазміди рРВ144 Sall (498 pb), що містить 5'-кінець гена, що кодує (ген грипу свиней НА штам H3N2) глікопротеїн gD вірусу хвороби Aujeszky (Штам За технологією з прикладу 6 провели реакцію N1A3) (фрагмент В). RT-PCR з РНК геному вірусу грипу свиней (штам Гібридизацією наступних 2 олігонуклеотидів: SIV H3N2 Cotes du Nord 1987), отриманої за техРВ101 (15 mer) (SEQ ID №7): нологією, що описана в прикладі 4, і з такими олі5'GATGCTGCTCGCAGC 3' гонуклеотидами: PB102 (19 mer) (SEQ ID №8) P9095 (31 mer) (SEQ ID №15) 5'GCTGCGAGCAGCATCTGCA 3' 5'GTTCTGCAGGCAGGGGATAATTCTATCAAC фрагмент (55 pb), що містить послідовність 5' C 3' гена gD, від ініціального кодона ATG до сайта PB096 (36 mer) (SEQ ID №16) Есо47ІІІ, був перетворений з утворенням сайта 5'TTGCGGCCGCAAGGGTGTTTTTAATTACTAA Pstl на 5' (фрагмент С). Після очищення, фрагменTATAC 3' ти А, В і С разом були зшиті з вектором pVR1012 щоб точно ізолювати ген, який кодує протеїн (приклад 7), попередньо розщепленим за допомоНА вірусу SIV H3N2 (Фігура №10 і SEQ ID №17), у гою Pstl і Bglll, і одержали плазміду рРВ098 (6076 формі фрагменту Pstl-Notl. Після очищення, проpb) (Фігура №5). дукт RT-PCR (1765 pb) зшили з вектором PCRIIПриклад 10: Конструювання плазміди рРВ143 прямої (Інвітроген № К2000-01), щоб одержати (ген Грипу свиней НА штам H1N1) вектор рРВ120 (5716 pb). За технологією з прикладу 6 провели реакцію Вектор рРВ120 розщепили за допомогою Notl, RT-PCR з РНК геному вірусу грипу свиней (штам щоб визволити фрагмент Notl-Notl (1797 pb), що SIV HINI «SW»), отриманої за технологією, що містить ген НА. Потім цей фрагмент зшили з векописана в прикладі 4, і з такими олігонуклеотидатором pVR1012 (приклад 7), попередньо розщепми: леним за допомогою Notl, і одержали плазміду РВ107 (32mer) (SEQ ID №9) рРВ144 (6712pb). що містить ген НА H3N2 у нор5'GTTCTGCAGCACCCGGGAGCAAAAGCAGG мальній орієнтації в порівнянні з промотором (Фіг. GGA 3' №11). PB108 (33mer) (SEQ ID №10) Приклад 13: Конструювання плазміди рРВ132 5'ATTGCGGCCGCTAGTAGAAACAAGGGTGTT (ген грипу свиней NP штам H3N2) TTT3' За технологією з прикладу 6 провели реакцію щоб точно ізолювати ген, який кодує протеїн RT-PCR з РНК геному вірусу грипу свиней (штам НА вірусу SIV HINI (Фігура №6 і SEQ ID N 11), у SIV H3N2 Cotes du Nord 1987), що отримана за формі фрагменту PCR (1803 pb). Після очищення, технологією, описаною в прикладі 4, і з такими цей фрагмент зшили з вектором PCRII-прямої (Інолігонуклеотидами: вітроген № К2000-01), щоб одержати вектор РВ097 (36 mer) (SEQ ID №12) рРВ137 (5755 pb). Вектор рРВ137 розщепили за 5'CCGGTCGACCGGGATAATCACTCACTGAGT допомогою EcoRV і Notl, щоб визволити фрагмент GACATC 3' EcoRV-Notl (1820 pb), що містить ген НА. Потім PB098 (33 mer) (SEQ ID №13) цей фрагмент зшили з вектором PVR1012 (прик 13 91675 14 5'TTGCGGCCGCTGTAGAAACAAGGGTATTTT 5'AAACTGCAGCAATGGCTCATCAGTGTGCAC TCT 3' GC 3' щоб точно ізолювати ген, що кодує протеїн NP AB171 (30 mer) (SEQ ID №24) вірусу SIV H3N2 (Фігура №12 і SEQ ID №18), в фо5'CGCGGATCCTTATCGTGATGTACTGGGGA рмі фрагменту Sall-Notl. Після очищення, продукт 3' RT-PCR (1564 pb) зшили з вектором PCRII-прямої щоб точно ізолювати ген «ORF3», що кодує (Інвітроген № К2000-01), щоб одержати вектор оболонковий глікопротеїн (gp45) вірусу PRRS рРВ123 (5485 pb). Вектор рРВ123 розщепили за штама Leiystad. Після очищення, продукт RT-PCR допомогою Sall і Notl, щоб визволити фрагмент (818 pb) розщепили за допомогою Pstl і BamHІ, Sall і Notl (1558 pb), що містить ген NP. Потім цей щоб ізолювати фрагмент Pstl-BamHI (802 pb). Цей фрагмент зшили з вектором pVR1012 (приклад 7), фрагмент зшили з вектором pVR1012 (приклад 7), попередньо розщепленим за допомогою Sall і Notl, попередньо розщепленим за допомогою Pstl і і одержали плазміду рРВ132 (6449 pb) (Фігура BamHІ, і одержали плазміду рАВ091 (5660 pb) (Фі№13). гура №16). Приклад 14: Конструювання плазміди рАВ025 Приклад 17: Конструювання плазміди рАВ092 (ген PRRSV ORF5)штам Leiystad (ген PPRSV ORF3 штам USA) За технологією з прикладу 6 провели реакцію За технологією з прикладу 6 провели реакцію RT-PCR з РНК геному вірусу PRRSV (штам RT-PCR з РНК геному вірусу PRRSV (Штам ATCCLeiystad) (J.Meulenberg і ін. Virology. 1993, 19, 62VR2332) (M.Murtaugh і ін. Arch. Virol. 1995, 140, 72), отриманої за технологією, що описана в прик1451-1460), отриманої за технологією, що описана ладі 4, і з такими олігонуклеотидами: в прикладі 4, і з такими олігонуклеотидами: AB055 (34 mer) (SEQ ID №19) АВ172 (32 mer) (SEQ ID №25) 5'ACGCGTCGACAATATGAGATGTTCTCACAA 5'AAACTGCAGCAATGGTTAATAGCTGTACATT ATTG 3' C 3' AB056 (33 mer) (SEQ ID №20) АВ173 (32 mer) (SEQ ID №26) 5'CGCGGATCCCGTCTAGGCCTCCCATTGCTC 5'CGCGGATCCCTATCGCCGTACGGCACTGA AGC 3' GGG 3' щоб точно ізолювати ген «ORF5», що кодує щоб точно ізолювати ген ORF3, що кодує обоглікопротеїн оболонки Ε (gp25) вірусу PRRS штаму лонковий глікопротеїн (gp45) вірусу PRRS штаму Leiystad. Після очищення, продукт RT-PCR (630 ATCC-VR2332. Після очищення, продукт RT-PCR pb) розщепили за допомогою Sall і BamHІ, щоб (785 pb) розщепили за допомогою Pstl і BamHІ, ізолювати фрагмент Sall-BamHI (617 pb). Цей фращоб ізолювати фрагмент Pstl-BamHI (769 pb). Цей гмент зшили з вектором pVR1012 (приклад 7), пофрагмент зшили з вектором pVR1012 (приклад 7), передньо розщепленим за допомогою Sall і BamHІ, попередньо розщепленим за допомогою Pstl і і одержали плазміду рДВ025 (5486 pb) (Фігура BamHІ, і одержали плазміду рАВ092 (5627 pb) (Фі№14). гура №17). Приклад 15: Конструювання плазміди рАВ00І Приклад 18: Конструювання плазміди рАВ004 PRRSV ORF5 штам USA (ген парвовірусу свиней VP2) За технологією з прикладу 6 провели реакцію За технологією з прикладу 6 провели реакцію RT-PCR з РНК геному вірусу PRRSV (Штам ATCCRT-PCR з РНК геному парвовірусу свиней (Штам VR2332) (M.Murtaugh і ін. Arch. Virol. 1995, 140, NADL2) (J. Vasudevacharya і ін. Virology. 1995, 178, 1451-1460), отриманої за технологією, що описана 611-616), отриманої за технологією, що описана в в прикладі 4, і з такими олігонуклеотидами: прикладі 4, і з такими олігонуклеотидами: АВ001 (30 mer) (SEQ ID №21) АВ007 (33 mer) (SEQ ID №27) 5'AACTGCAGATGTTGGAGAAATGCTTGACCG 5'AAAACTGCAGAATGAGTGAAAATGTGGAAC 3' AAC 3' AB002 (30 mer) (SEQ ID №22) AB010 (33 mer) (SEQ ID №28) 5'CGGGATCCCTAAGGACGACCCCATTGTTCC 5'CGCGGATCCCTAGTATAATTTTCTTGGTATA 3' AG 3' щоб точно ізолювати ген, що кодує оболонкощоб розширити фрагмент (1757 pb), що місвий глікопротеїн Ε (gp25) вірусу PRRS штаму тить ген, що кодує протеїн VP2 парвовірусу свиATCC-VR2332. Після очищення, продукт RT-PCR ней. Після очищення, продукт RT-PCR розщепили (620 pb) розщепили за допомогою Pstl і BamHІ, за допомогою Pstl і BarnHI, щоб ізолювати фрагщоб ізолювати фрагмент Pstl-BamHI (606 pb). Цей мент Pstl-BamHI (1740 pb). Цей фрагмент зшили з фрагмент зшили з вектором pVR1012 (приклад 7), вектором pVR1012 (приклад 7), попередньо розпопередньо розщепленим за допомогою Pstl і щепленим за допомогою Pstl і BamHІ, і одержали BamHІ, і одержали плазміду рАВ001 (5463 pb) (Фіплазміду рАВ004 (6601 pb) (Фігура №18). гура №15). Приклад 19: Конструювання плазміди рАВ069 Приклад 16: Конструювання плазміди рАВ091 (ген Чуми свиней HCVE1) (ген PRRSV ORF3 штам Leiystad) За технологією з прикладу 6 провели реакцію За технологією з прикладу 6 провели реакцію RT-PCR з РНК геному вірусу чуми свиней (Hog RT-PCR з РНК геному вірусу PRRSV (Штам Cholera Virus) (HCV) (Штам Alfort) (G. Meyers і ін. Leiystad) (J. Meulenberg і ін. Virology, 1993, 19, 62Virology. 1989, 171, 18-27), отриманої за технологі72), отриманою за технологією, що описана в приєю, що описана в прикладі 4, і з такими олігонуккладі 4, і з такими олігонуклеотидами: леотидами: АВ170 (32 mer) (SEQ ID №23) A3126 (36 mer) (SEQ ID №29) 15 91675 16 5'ACGCGTCGACATGAAACTAGAAAAAGCCCT 5'TTGAATTCTATCGCTACAGTAAGGAGTACG GTTGGC 3' G 3' АВ127 (34 mer) (SEQ ID №30) РВ175 (31 mer) (SEQ ID №36) 5'CGCGGATCCTCATAGCCGCCCTTGTGCCCC 5'TTGGATCCGCTATTTA7CATCTAAAAATAAC GGTC 3' 3' щоб ізолювати послідовність, що кодує протещоб розширити частину 3' гена apxl, що кодує їн Е1 вірусу HCV, у формі фрагмента RT-PCR протеїн гемолізин І (Actinobacillus (1364 pb). Після очищення, цей фрагмент розщеpleuropneumoniae), у формі фрагменту EcoRIпили за допомогою BamHІ і Sall, щоб одержати BamHI. Після очищення, продукт PCR (576 pb) фрагмент Sall-BamHI (1349 pb). Цей фрагмент розщепили за допомогою EcoRI і BaitiHI, щоб ізозшили з вектором pVR.1012 (приклад 7), попередлювати фрагмент EcoRI-BamHI(566 pb) (фрагмент ньо за допомогою BamHІ і Sall, і одержали плазміУ). Фрагменти А і В зшили разом із вектором ду рАВ069 (6218 pb) (Фігура №19). pVR1012 (приклад 7), попередньо розщепленим за Приклад 20: Конструювання плазміди рАВ061 допомогою Sall і BamHІ, і одержали плазміду (ген Чуми свиней HCV E2) рРВ3162 (7619 pb) (Фігура №21). За технологією з прикладу 6 провели реакцію Приклад 22: Конструювання плазміди рРВ163 RT-PCR з РНК геному вірусу чуми свиней (Hog (ген Actinobacillus pleuropneumoniae apxl I делеціCholera Virus) (HCV) (Штам Alfort) (G.Meyers і ін. льований) Virology. 1989. 171. 18-27), отриманої за технологіГен apxll (Actinobacillus pleuropneumoniae) був єю, що описана в прикладі 4, і з такими олігонукклонований таким чином, щоб мала місце делеція леотидами: ділянки амінокислот з великим вмістом гліцину (що А3118 (36 mer) (SEQ ID №31) бере участь у фіксації іона кальцію), який знахо5'ACGCGTCGACATGTCAACTACTGCGTTTCT диться між амінокислотами 716 і 813. CATTTG 3' Провели реакцію PCR з ДНК геному AB119 (33 mer) (SEQ ID №32) Actinobacillus pleuropneumoniae (Серотип 9) (Μ. 5'CGCGGATCCTCACTGTAGACCAGCAGCGA Smits і ін., Infection and Immunity. 1991, 59, 4497GCTG 3' 4504), отриманої за технологією, що описана в щоб ізолювати послідовність, що кодує протеприкладах 2 і 3, і з такими олігонуклеотидами: їн Е2 вірусу HCV, у формі фрагмента RT-PCR РВ176 (31 mer) (SEQ ID №37) (1246 pb). Після очищення, цей фрагмент розще5'TTGTCGACGATCAATTATATAAAGGAGACTC пили за допомогою BamHІ і Sall, щоб одержати 3' фрагмент Salll-BamHI (1232 pb). Цей фрагмент РВ191 (30 mer) (SEQ ID №38) зшили з вектором pVR1012 (приклад 7), поперед5'TTGAATTCCTCTTCAACTGATTTGAGTGAG ньо розщеплений за допомогою BamHІ і Sall, і 3' одержали плазміду рАВ061 (6101 pb) (Фігура щоб розширити частину 5' гена apxll, що кодує №20). протеїн гемолізин II (Actinobacillus Приклад 21: Конструювання плазміди рРВ162 pleuropneumoniae), у формі фрагменту Sall-EcoRI. (ген Actino-bacillus pleuropneumoniae apxl делеціПісля очищення, продукт PCR (2190 pb) розщепильований) ли за допомогою Sall і EcoRI, щоб ізолювати фраГен apxl (Actinobacillus pleuropneumoniae) був гмент Sall-EcoRI (2180 pb) (фрагмент А). клонований таким чином, щоб мала місце делеція Провели реакцію PCR з ДНК геному ділянки амінокислот з великим вмістом гліцину (що Actinobacillus pleuropneumoniae (Серотип 9) (Μ. бере участь у фіксації іона кальцію), який знахоSmits і ін., Infection and Immunity. 1991, 59, 4497диться між амінокислотами 719 і 846. 4504) і з такими олігонуклеотидами: Провели реакцію PCR із ДНК геному РВ192 (29 mer) (SEQ ID №39) Actinobacillus pleuropneumoniae (Серотип 1) (J. 5'TTGAATTCGTAAATCTTAAAGACCTCACC 3' Frey і ін. Infect. Immun. 1991, 59, 3026-3032), отриРВ177 (30 mer) (SEQ ID №40) маної за технологією, що описана в прикладах 2 і 5'TTGGATCCACCATAGGATTGCTATGATTTG 3, і з такими олігонуклеотидами: 3' РВ174 (32 mer) (SEQ ID №33) щоб розширити частину 3' гена apxll, що кодує 5'TTGTCGACGTAAATAGCTAAGGAGACAACA протеїн гемолізин II (Actinobacillus TG 3' pleuropneumoniae) у формі фрагменту EcoRIРВ189 (29 mer) (SEQ ID №34) BamHI. Після очищення, продукт PCR (473 pb) 5'TTGAATTCTTCTTCAACAGAATGTAATTC 3' розщепили за допомогою EcoRI і BamHІ, щоб ізощоб розширити частину 5' гена apxl, що кодує лювати фрагмент EcoRI-BamHI (463 pb) (фрагмент протеїн гемолізин І (Actinobacillus В). Фрагменти А і В зшили разом із вектором pleuropneumoniae), у формі фрагменту Sall-EcoRI. pVR1012 (приклад 7), попередньо розщепленим за Після очищення, продукт PCR (2193 pb) розщепидопомогою Sall і BamHІ, і одержали плазміду ли за допомогою Sall і EcoRI, щоб ізолювати фрарРВ163 (7513 pb) (Фігура №22). гмент Sall-EcoRI (2183 pb) (фрагмент А). Провели Приклад 23: Конструювання плазмід рРВ174', реакцію PCR з ДНК геному Actinobacillus рРВ189 і рРВ190 (ген Actinobacillus pleuropneumoniae (Серотип 1) (J.Frey і ін. Infect pleuropneumoniae архШ делецільований). Перший linmun. 1991, 59, 3026-3032) і з наступними олігоприклад делеції в АрхІII (плазміда рРВ174'): нуклеотидами: Ген apxIll (Actinobacillus pleuropneumoniae) був РВ190 (31 mer) (SEQ ID №35) клонований таким чином, щоб мала місце делеція ділянки амінокислот з великим вмістом гліцину (що 17 91675 18 бере участь у фіксації іона кальцію), який знахоSmits 1992. № доступу послідовності в диться між амінокислотами 733 і 860. Genbank=X68815), і з такими олігонуклеотидами: Провели реакцію PCR з ДНК геному РВ306 (31 mer) (SEQ ID №46) Actinobacillus pleuropneumoniae (Серотип 8) (Μ. 5'TTTATCGATTCTGATT77TCCTTCGATCGTC Smits 1992. № доступу послідовності в 3' Genbank=X68815), отриманої за технологією, що РВ307 (32 mer) (SEQ ID №44) описана в прикладах 2 і 3, і з такими олігонуклео5'TTGGATCCTTAAGCTGCTCTAGCTAGGTTAC гидами: C 3' РВ278 (30 mer) (SEQ ID №41) щоб розширити частину 3' гена архІІІ, що ко5'TTTGTCGACATGAGTACTTGGTCAAGCATG дує протеїн гемолізин III (Actinobacillus 3' pleuropneumoniae), у формі фрагменту Clal-BamHI. РВ279 (29 mer) (SEQ ID №42) Після очищення, продукт PCR (518 pb) розщепили 5'TTTATCGATTCTTCTACTGAATGTAATTC 3' за допомогою СІаІ і BamHІ, щоб ізолювати фрагщоб розширити частину 5' гена архІІІ, що комент Clal-BamHI (506 pb) (фрагмент В). Фрагменти дує протеїн гемолізин III (Actinobacillus А і В зшили разом із вектором pVR1012 (приклад pleuropneumoniae) у формі фрагменту Sall-Clal. 7), попередньо розщепленим за допомогою Sall і Після очищення, продукт PCR (2216 pb) розщепиBamHІ, і одержали плазміду рРВ189 (7496 pb) (Філи за допомогою Sall і СІаІ, щоб ізолювати фраггура №24). мент Sall-Clal (2205 pb) (фрагмент А). Провели Третій приклад делеції в АрхІІІ (плазміда реакцію PCR з ДНК геному Actinobacillus рРВ190): pleuropneumoniae (Серотип 8) (Μ. Smits 1992. № Ген архІІІ (Actinobacillus pleuropneumoniae) був доступу послідовності в Genbank=X68815), і з таклонований таким чином, щоб мала місце делеція кими олігонуклеотидами: ділянки амінокислот з великим вмістом гліцину (що РВ280 (33 mer) (SEQ ID №43) бере участь у фіксації іона кальцію), який знахо5'TTTATCGATTTATGTTTATCGTTCCACTTCAG диться між амінокислотами 718 і 876. G 3' Провели реакцію PCR з ДНК геному PS307 (32 mer) (SEQ ID №44) Actinobacillus pleuropneumoniae (Серотип 8) (Μ. 5'TTGGATCCTTAAGCTGCTCTAGCTAGGTTAC Smits 1992. № доступу послідовності в C 3' Genbank=X68815), отриманої за технологією, що щоб розширити частину 3' гена apxlll, що кодує описана в прикладах 2 і 3, і з такими олігонуклетипротеїн гемолізин III (Actinobacillus дами: pleuropneumoniae), у формі фрагменту Clal-BamHI. PB278 (30 mer) (SEQ ID №41) Після очищення, продукт PCR (596 pb) розщепили 5'TTTGTCGACATGAGTACTTGGTCAAGCATG за допомогою СІаІ і BamHІ, щоб ізолювати фраг3' мент Clal-BamHI (583 pb) (фрагмент В). Фрагменти PB304 (33 mer) (SEQ ID №47) А і В зшили разом із вектором pVR1012 (приклад 5'TTTATCGATACCTGATTGCGTTAATTCATAAT 7), попередньо розщепленим за допомогою Sall і C 3' BamHІ, і одержали плазміду РРВ174' (7658 pb) щоб розширити частину 5' гена apxlll, що кодує (Фігура №23). протеїн гемолізин III (Actinobacillus Другий приклад делеції в АрхІІІ (плазміда pleuropneumoniae), у формі фрагменту Sall-Clal. рРВ189): Після очищення, продукт PCR (2172 pb) розщепиГен архІІІ (Actinobacillus pleuropneumoniae) був ли за допомогою Sall і СІаІ, щоб ізолювати фрагклонований таким чином, щоб мала місце делеція мент Sall-Clal (2161 pb) (фрагмент А). ділянки амінокислот із великим вмістом гліцину Провели реакцію PCR з ДНК геному (що бере участь у фіксації іона кальцію), який знаActinobacillus pleuropneumoniae) (Серотип 8) (Μ. ходиться між амінокислотами 705 і 886. Smits 1992. № доступу послідовності в Провели реакцію PCR з ДНК геному Genbank=X68815), і з такими оліго-нуклеотидами: Actinobacillus pleuropneumoniae (Серотип 8) РВ305 (31 mer) (SEQ ID №48) (M.Smis 1992. № доступу послідовності в 5'TTTATCGATAAATCTAGTGATTTAGATAAAC Genbank=X68815), отриманої за технологією, опи3' саної в прикладах 2 і 3, і з такими олігонуклеотиРВ307 (32 mer) (SEQ ID №44) дами: 5'TTGGA7CCTTAAGCTGCTCTAGCTAGGTTAC РВ278 (30 mer) (SEQ ID №41) C 3' 5'TTTGTCGACATGAGTACTTGGTCAAGCATG щоб розширити частину 3' гена apxlll, що кодує 3' протеїн гемолізин III (Actinobacillus РВ303 (32 mer) (SEQ ID №45) pleuropneumoniae), у формі фрагменту СІаІ і 5'TTTATCGATTTCTTCACGTTTACCAACAGCA BamHІ. Після очищення, продукт PCR (548 pb) 3' розщепили за допомогою СІаІ і BamHІ, щоб ізолющоб розширити частину 5' гена архІІІ, що ковати фрагмент Clal-BamHI (536 pb) (фрагмент В). дує протеїн гемолізин III (Actinobacillus Фрагменти А і В зшили разом із вектором pVR1012 pleuropneumoniae), у формі фрагменту Sall-Clal. (приклад 7), попередньо розщепленим за допомоПісля очищення, продукт PCR (2133 pb) розщепигою Sall і BamHІ, і одержали плазміду рРВ190 ли за допомогою Sall і СІаІ, щоб ізолювати фраг(7565 pb). (Фігура №25). мент Sall-Clal (2122 pb) (фрагмент А). Приклад 24: Одержання і очищення плазмід Провели реакцію PCR з ДНК геному Для одержання плазмід, призначених для вакActinobacillus pleuropneumoniae (Серотип 8) (Μ. цинації тварин, можна використовувати будь-яку 19 91675 20 технологію, що дозволяє одержати суспензію відповідала ефективній дозі кожної плазміди. У очищених плазмід, здебільшого, у надскрученій якості розчинів для доведення кінцевої концентраформі. Ці технології добре відомі фахівцям. Можна ції вакцини можна використовувати або розчин назвати, зокрема, техніку лужного лізису з наступ0,9% NaCI, або буфер PBS. ним дворазовим центрифугуванням у градієнті Особливі компоненти, такі як ліпосоми, катіонхлориду цезію і в присутності броміду етидію, як ні ліпіди, також можуть бути використані для одеописано J.Sambrook і ін. (Molecular Cloning: A ржання вакцин. Laboratory Manual. 2-е вид. Cold Spring Harbor Приклад 26: Вакцинація свиней Laboratory. Cold Spring Harbor. New York. 1989). Свиней вакцинують дозами 100 мкг, 250 мкг Можна також звернутися до матеріалів заявок РСТ або 500 мкг на кожну плазміду. WO 95/21250 і РСТ WO 96/02658, у яких описані Ін'єкції можна проводити за допомогою голки методики одержання в промисловому масштабі внутрішньом'язово. У цьому випадку, вакцинні доплазмід для вакцинації. Для одержання вакцин зи вводять в об'ємі 2 мл. (див. приклад 17), готують суспензію очищених ін'єкції можна проводити внутрішньошкірно, плазмід таким чином, щоб одержати висококонцевикористовуючи рідиннострумінний (без голки) нтровані розчини (>2 мг/мл), придатні для зберіін'єкційний апарат і вводячи дози 0,2 мл в 5 точках гання. Для цього готують суспензію плазмід або в (0,04 мл у кожній точці) (наприклад, апарат ультрачистій воді, або в буфері ТЕ (Tris-HCI 10 «PIGJET»). У цьому випадку, вакцини вводять в мМ; EDTA 1 мМ, рН 8,0). об'ємах 0,2 або 0,4 мл, що відповідає, одному або Приклад 25: Одержання асоційованих вакцин двом введенням. Якщо два послідовних введення Різні плазміди, необхідні для одержання асоздійснюють за допомогою апарата PIGJET, ці ввеційованої вакцини, змішують у їхніх концентровадення здійснюють таким чином, щоб обидві ін'єкних розчинах (приклад 16). Суміші готують таким ційні зони були віддалені одна від другої на відсчином, щоб кінцева концентрація кожної плазміди тань біля 1-2 сантиметрів. 21 91675 22 23 91675 24 25 91675 26 27 91675 28 29 91675 30 31 91675 32 33 91675 34 35 91675 36 37 91675 38 39 91675 40 41 91675 42 43 91675 44 45 91675 46 47 91675 48 49 91675 50 51 91675 52 53 91675 54 55 91675 56 57 91675 58 59 91675 60 В описі до патенту на винахід графічні зображення та текст подаються в редакції заявника Комп’ютерна верстка О. Гапоненко Підписне Тираж 28 прим. Міністерство освіти і науки України Державний департамент інтелектуальної власності, вул. Урицького, 45, м. Київ, МСП, 03680, Україна ДП “Український інститут промислової власності”, вул. Глазунова, 1, м. Київ – 42, 01601
ДивитисяДодаткова інформація
Назва патенту англійськоюFormula of polynucleotide vaccine against respiratory and reproduction hog pathologies
Автори англійськоюAudonnet, Jean-Christophe Francois, Bouchardon, Annabelle, Baudu,Philippe, Riviere, Michel
Назва патенту російськоюФормула полинуклеотидной вакцины против дыхательных паталогий и паталогий размножения свиней
Автори російськоюОдонне Жан-Кристоф Франсис, Бушардон Аннабель, Бодю Филипп, Ривьер Мишель
МПК / Мітки
МПК: C12N 15/00, A61K 39/295
Мітки: формула, свиней, вакцини, патологій, дихальних, розмноження, полінуклеотидної
Код посилання
<a href="https://ua.patents.su/30-91675-formula-polinukleotidno-vakcini-proti-dikhalnikh-patologijj-ta-patologijj-rozmnozhennya-svinejj.html" target="_blank" rel="follow" title="База патентів України">Формула полінуклеотидної вакцини проти дихальних патологій та патологій розмноження свиней</a>
Попередній патент: Система і спосіб, що дозволяє використовувати технологію wusb при здійсненні розподіленого керування доступом до надширокосмугового середовища передавання даних
Наступний патент: Композиція, що містить інгібітор jnk та циклоспорин
Випадковий патент: Плаваюча фасонна втулка високочастотних центрувально-фіксуючих опор магнітних підшипників