Вакцина на основі свинячого вірусу torque teno та спосіб діагностики інфекцій, викликаних цим вірусом
Формула / Реферат
1. Інфекційна молекула нуклеїнової кислоти вірусу Torque teno свиней (PTTV), що являє собою молекулу нуклеїнової кислоти, яка кодує інфекційний PTTV, що містить щонайменше одну копію геномної послідовності, вибраної з групи, що складається з PTTV1a-VA, PTTV1b-VA, PTTV2b-VA і PTTV2c-VA.
2. Інфекційна молекула нуклеїнової кислоти за п. 1, вякій щонайменше одна копія геномної послідовності, принаймні на 95 % гомологічна геномній послідовності, вибраній з групи, що складається з PTTV1a-VA, PTTV1b-VA, PTTV2b-VA і PTTV2c-VA.
3. Інфекційна молекула нуклеїнової кислоти за п. 1, в якій геномну послідовність вибирають з послідовностей, представлених SEQ ID NO: 9, SEQ ID NO: 10, SEQ ID NO: 11 і SEQ ID NO: 12.
4. Біологічно функціональна плазміда або вірусний вектор, які містять інфекційну молекулу нуклеїнової кислоти за п. 3.
5. Біологічно функціональна плазміда або вірусний вектор за п. 4, які містять більше, ніж одну копію інфекційної молекули нуклеїнової кислоти.
6. Прийнятна клітина-господар, що трансфікується вектором, який містить інфекційну молекулу нуклеїнової кислоти за п. 4.
7. Авірулентний інфекційний PTTV, який продукується клітинами, що містять інфекційну молекулу нуклеїнової кислоти за п. 6.
8. Вакцина, яка містить нетоксичний фізіологічно прийнятний носій та імуногенну кількість елемента, вибраного з групи, що складається з:
(a) молекули нуклеїнової кислоти, яка містить щонайменше одну копію геномної послідовності, принаймні на 80 % гомологічної геномній послідовності, вибраній з групи, що складається з PTTV генотипів або підтипів PTTV1a-VA, PTTV1b-VA, PTTV2b-VA або PTTV2c-VA, або її комплементарну нитку;
(б) біологічно функціональної плазміди або вірусного вектора, які містять молекулу нуклеїнової кислоти, що включає щонайменше одну копію геномної послідовності, принаймні на 80 % гомологічної геномній послідовності, вибраній з групи, що складається з PTTV генотипів або підтипів PTTV1a-VA, PTTV1b-VA, PTTV2b-VA або PTTV2c-VA, або її комплементарну нитку;
(в) авірулентного інфекційного непатогенного вірусу Torque teno свиней, отриманого при використанні геномної послідовності, що містить геномну послідовність, вибрану з групи, що складається з PTTV генотипів або підтипів PTTV1a-VA, PTTV1b-VA, PTTV2b-VA або PTTV2c-VA; і
(г) імуногенного фрагмента поліпептидної послідовності або повного білка, трансльованих відповідно до полінуклеотидної послідовності, вибраної з групи, що складається з ORF1, ORF2, ORF1/1 і ORF2/2 генотипів або підтипів PTTV PTTV1a-VA, PTTV1b-VA, PTTV2b-VA і PTTV2c-VA.
9. Вакцина за п. 8, яка містить живий вірус PTTV.
10. Вакцина за п. 8, яка містить інактивований (вбитий) вірус PTTV.
11. Вакцина за п. 8, в якій імуногенний фрагмент поліпептидної послідовності або повний білок містять очищені експресовані в бактеріях або за допомогою бакуловірусів рекомбінантні білки, експресовані при використанні ORF1, ORF2, ORF1/1 і ORF2/2 PTTV генотипів або підтипів PTTV1a-VA, PTTV1b-VA, PTTV2b-VA і PTTV2c-VA.
12. Вакцина за п. 11, в якій імуногенний фрагмент поліпептидної послідовності або повний білок містять очищені експресовані в бактеріях або за допомогою бакуловірусів рекомбінантні білки, експресовані при використанні ORF1 PTTV генотипів або підтипів PTTV1a-VA, PTTV1b-VA, PTTV2b-VA і PTTV2c-VA.
13. Вакцина за п. 8, яка додатково містить ад'ювант.
14. Спосіб імунізації свині проти PTTV вірусної інфекції, який включає введення свині імунологічно ефективної кількості вакцини за п. 8.
15. Спосіб за п. 14, який включає введення свині вакцини парентерально, інтраназально, інтрадермально або трансдермально.
16. Спосіб за п. 14, який включає введення свині вакцини всередину лімфатичного вузла або внутрішньом'язово.
17. Виділений полінуклеотид, який містить полінуклеотидну послідовність, що гомологічна нуклеотидній послідовності PTTV1a-VA, представленій SEQ ID NO: 9.
18. Виділений полінуклеотид за п. 17, полінуклеотидна послідовність якого на 95 % гомологічна нуклеотидній послідовності PTTV1a-VA, представленій SEQ ID NO: 9.
19. Виділений полінуклеотид за п. 17, полінуклеотидна послідовність якого містить нуклеотидну послідовність PTTV1a-VA, представлену SEQ ID NO: 9.
20. Виділений полінуклеотид, який містить полінуклеотидну послідовність, що гомологічна нуклеотидній послідовності PTTV1b-VA, представленій SEQ ID NO: 10.
21. Виділений полінуклеотид за п. 20, полінуклеотидна послідовність якого на 95 % гомологічна нуклеотидній послідовності PTTV1b-VA, представленій SEQ ID NO: 10.
22. Виділений полінуклеотид за п. 20, полінуклеотидна послідовність якого містить нуклеотидну послідовність PTTV1b-VA, представлену SEQ ID NO: 10.
23. Виділений полінуклеотид, який містить полінуклеотидну послідовність, що гомологічна нуклеотидній послідовності PTTV2b-VA, представленій SEQ ID NO: 11.
24. Виділений полінуклеотид за п. 23, полінуклеотидна послідовність якого на 95 % гомологічна нуклеотидній послідовності PTTV2b-VA, представленій SEQ ID NO: 11.
25. Виділений полінуклеотид за п. 23, полінуклеотидна послідовність якого містить нуклеотидну послідовність PTTV2b-VA, представлену SEQ ID NO: 11.
26. Виділений полінуклеотид, який містить полінуклеотидну послідовність, що гомологічна нуклеотидній послідовності PTTV2с-VA, представленій SEQ ID NO: 12.
27. Виділений полінуклеотид за п. 26, полінуклеотидна послідовність якого на 95 % гомологічна нуклеотидній послідовності PTTV2с-VA, представленій SEQ ID NO: 12.
28. Виділений полінуклеотид за п. 26, полінуклеотидна послідовність якого містить нуклеотидну послідовність PTTV2с-VA, представлену SEQ ID NO: 12.
29. Вакцина, яка містить імуногенний фрагмент поліпептидної послідовності або повний білок, трансльований відповідно до полінуклеотидної послідовності, обраної з групи, що складається з ORF1, ORF2, ORF1/1 і ORF2/2 PTTV генотипів або підтипів PTTV1a-VA, PTTV1b-VA, PTTV2b-VA і PTTV2c-VA.
30. Вакцина за п. 29, в якій імуногенний фрагмент або повний білок являє собою капсидний білок PTTV генотипів або підтипів PTTV1a-VA, PTTV1b-VA, PTTV2b-VA і PTTV2c-VA.
31. Вакцина за п. 29, в якій полінуклеотидну послідовність обирають з групи, що складається з ORF1 PTTV генотипів або підтипів PTTV1a-VA, PTTV1b-VA, PTTV2b-VA і PTTV2c-VA.
32. Вакцина за п. 31, в якій полінуклеотидна послідовність являє собою ORF1 PTTV генотипу PTTV1a-VA.
33. Вакцина за п. 31, в якій полінуклеотидна послідовність являє собою ORF1 PTTV генотипу PTTV1b-VA.
34. Вакцина за п. 31, в якій полінуклеотидна послідовність являє собою ORF1 PTTV підтипу PTTV2с-VA.
35. Вакцина за п. 29, в якій поліпептидну послідовність обирають з групи, що складається з послідовностей, представлених SEQ ID NO: 13, SEQ ID NO: 14, SEQ ID NO: 15, SEQ ID NO: 16, SEQ ID NO: 17, SEQ ID NO: 18, SEQ ID NO: 19, SEQ ID NO: 20, SEQ ID NO: 21, SEQ ID NO: 22, SEQ ID NO: 23, SEQ ID NO: 24, SEQ ID NO: 25, SEQ ID NO: 26, SEQ ID NO: 27 і SEQ ID NO: 28.
36. Вакцина за п. 35, в якій поліпептидна послідовність представлена SEQ ID NO: 13.
37. Вакцина за п. 36, в якій поліпептидна послідовність являє собою С-кінцеву область (а. к. (амінокислоти) 317-635) SEQ ID NO: 13.
38. Вакцина за п. 35, в якій поліпептидна послідовність представлена SEQ ID NO: 14.
39. Вакцина за п. 38, в якій поліпептидна послідовність являє собою С-кінцеву область (а. к. 322-639) SEQ ID NO: 14.
40. Вакцина за п. 35, в якій поліпептидна послідовність представлена SEQ ID NO: 16.
41. Вакцина за п. 40, в якій поліпептидна послідовність являє собою С-кінцеву область (а. к. 310-625) SEQ ID NO: 16.
42. Вакцина за п. 35, в якій поліпептидна послідовність представлена SEQ ID NO: 20.
43. Вакцина за п. 29, яка додатково містить ад'ювант.
44. Спосіб імунізації свині проти PTTV вірусної інфекції, що полягає у введенні свині імунологічно ефективної кількості вакцини за п. 29.
45. Спосіб за п. 44, який включає введення свині імуногенного фрагмента або повного білка.
46. Спосіб за п. 44, який включає введення свині вакцини парентерально, інтраназально, інтрадермально або трансдермально.
47. Спосіб за п. 44, який включає введення свині вакцини в лімфатичний вузол або внутрішньом'язово.
48. Спосіб діагностики PTTV1 інфекції і кількісного визначення PTTV1 навантаження, який включає:
екстракцію ДНК із зразка з очікуваною PTTV1 інфекцією;
проведення полімеразної ланцюгової реакції (ПЛР) із застосуванням праймерів, які містять послідовності, представлені SEQ ID NO: 29 і SEQ ID NO: 30; і
детекцію PTTV1-специфічної ампліфікації.
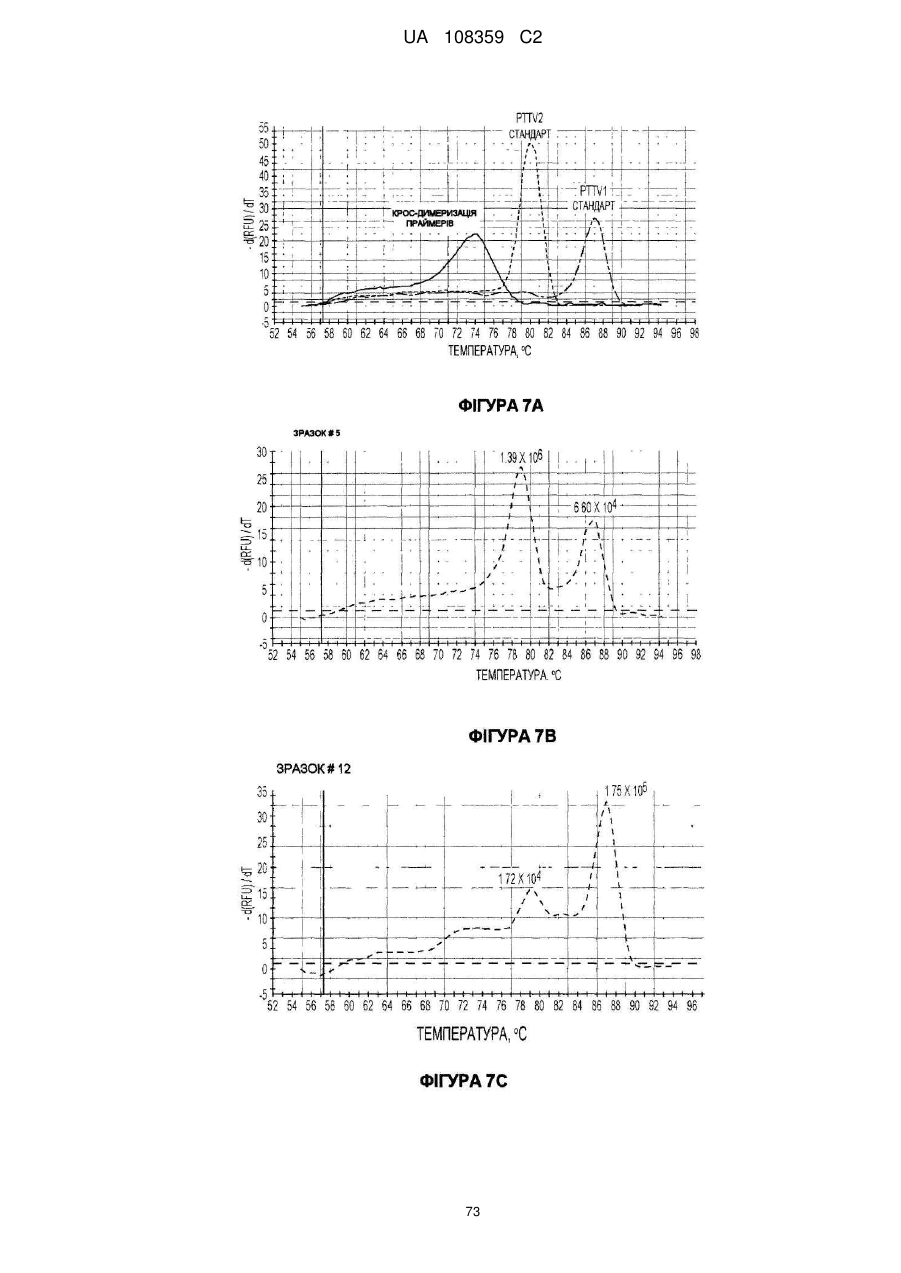
49. Спосіб за п. 48, в якому полімеразна ланцюгова реакція являє собою ПЛР в реальному часі з барвником SYBR green.
50. Спосіб діагностики PTTV2 інфекції і кількісного визначення PTTV2 навантаження, який включає:
екстракцію ДНК із зразка з очікуваною PTTV2 інфекцією;
проведення полімеразної ланцюгової реакції (ПЛР) із застосуванням праймерів, які містять послідовності, представлені SEQ ID NO: 29, SEQ ID NO: 30, SEQ ID NO: 31 і SEQ ID NO: 32; і
детекцію PTTV2-специфічної ампліфікації.
51. Спосіб за п. 50, в якому полімеразна ланцюгова реакція являє собою ПЛР в реальному часі з барвником SYBR green.
52. Спосіб одночасного виявлення і діагностики PTTV1 і PTTV2 інфекції, який включає:
екстракцію ДНК із зразка з очікуваною PTTV інфекцією;
проведення полімеразної ланцюгової реакції (ПЛР) із застосуванням праймерів, які містять послідовності, представлені в SEQ ID NO: 31 і SEQ ID NO: 32; і
детекцію PTTV1- і PTTV2-специфічної ампліфікації.
53. Спосіб за п. 52, в якому полімеразна ланцюгова реакція являє собою ПЛР в реальному часі з барвником SYBR green.
54. Спосіб одночасного виявлення і діагностики PTTVа1 і PTTV1b інфекції, який включає:
екстракцію ДНК із зразка з очікуваною PTTV1 інфекцією;
проведення першої полімеразної ланцюгової реакції (ПЛР) із застосуванням праймерів, які містять послідовності, представлені SEQ ID NO: 33 і SEQ ID NO: 34;
проведення другої ПЛР із застосуванням праймерів, які містять послідовності, представлені SEQ ID NO: 35, SEQ ID NO: 36, SEQ ID NO: 37 і SEQ ID NO: 38; і
детекцію PTTV1а- і PTTV1b-специфічної ампліфікації.
55. Спосіб діагностики PTTV інфекції, який включає:
іммобілізацію імуногенного фрагмента або повного білка з поліпептидною послідовністю, трансльованою відповідно до полінуклеотидної послідовності, вибраної з групи, що складається з ORF1, ORF2, ORF1/1 і ORF2/2 PTTV підтипів PTTV1a-VA, PTTV1b-VA, PTTV2b-VA і PTTV2c-VA;
контактування зразка сироватки свині з очікуваною PTTV інфекцією з іммобілізованим імуногенним фрагментом або повним білком; і
детекцію іммобілізованого антитіла, специфічного до імуногенного фрагмента.
56. Спосіб за п. 55, в якому полінуклеотидну послідовність вибирають з групи, яка складається з ORF1 PTTV генотипів і підтипів PTTV1a-VA, PTTV1b-VA, PTTV2b-VA і PTTV2c-VA.
57. Спосіб за п. 56, в якому полінуклеотидна послідовність являє собою ORF1 PTTV генотипу PTTV1a-VA.
58. Спосіб за п. 56, в якому полінуклеотидна послідовність являє собою ORF1 PTTV генотипу PTTV1b-VA.
59. Спосіб за п. 56, в якому полінуклеотидна послідовність являє собою ORF1 PTTV генотипу PTTV2с-VA.
60. Спосіб за п. 55, в якому поліпептидну послідовність вибирають з групи, що складається з послідовностей, представлених в SEQ ID NO: 13, SEQ ID NO: 14, SEQ ID NO: 15, SEQ ID NO: 16, SEQ ID NO: 17, SEQ ID NO: 18, SEQ ID NO: 19, SEQ ID NO: 20, SEQ ID NO: 21, SEQ ID NO: 22, SEQ ID NO: 23, SEQ ID NO: 24, SEQ ID NO: 25, SEQ ID NO: 26, SEQ ID NO: 27 і SEQ ID NO: 28.
61. Спосіб за п. 60, в якому поліпептидна послідовність представлена SEQ ID NO: 13.
62. Спосіб за п. 60, в якому поліпептидна послідовність представлена SEQ ID NO: 16.
63. Спосіб за п. 62, в якому імуногенний фрагмент являє собою С-кінцеву область (а. к. 317-635) SEQ ID NO: 13, (а. к. 322-639) SEQ ID NO: 14 або (а. к. 310-625) SEQ ID NO: 16.
64. Спосіб за п. 60, в якому поліпептидна послідовність представлена SEQ ID NO: 20.
65. Спосіб за п. 55, в якому детекцію іммобілізованого антитіла здійснюють методом Вестерн-блотингу (імуноблотингу).
66. Спосіб за п. 55, в якому детекцію іммобілізованого антитіла здійснюють твердофазним імуноферментним аналізом (ELISA).
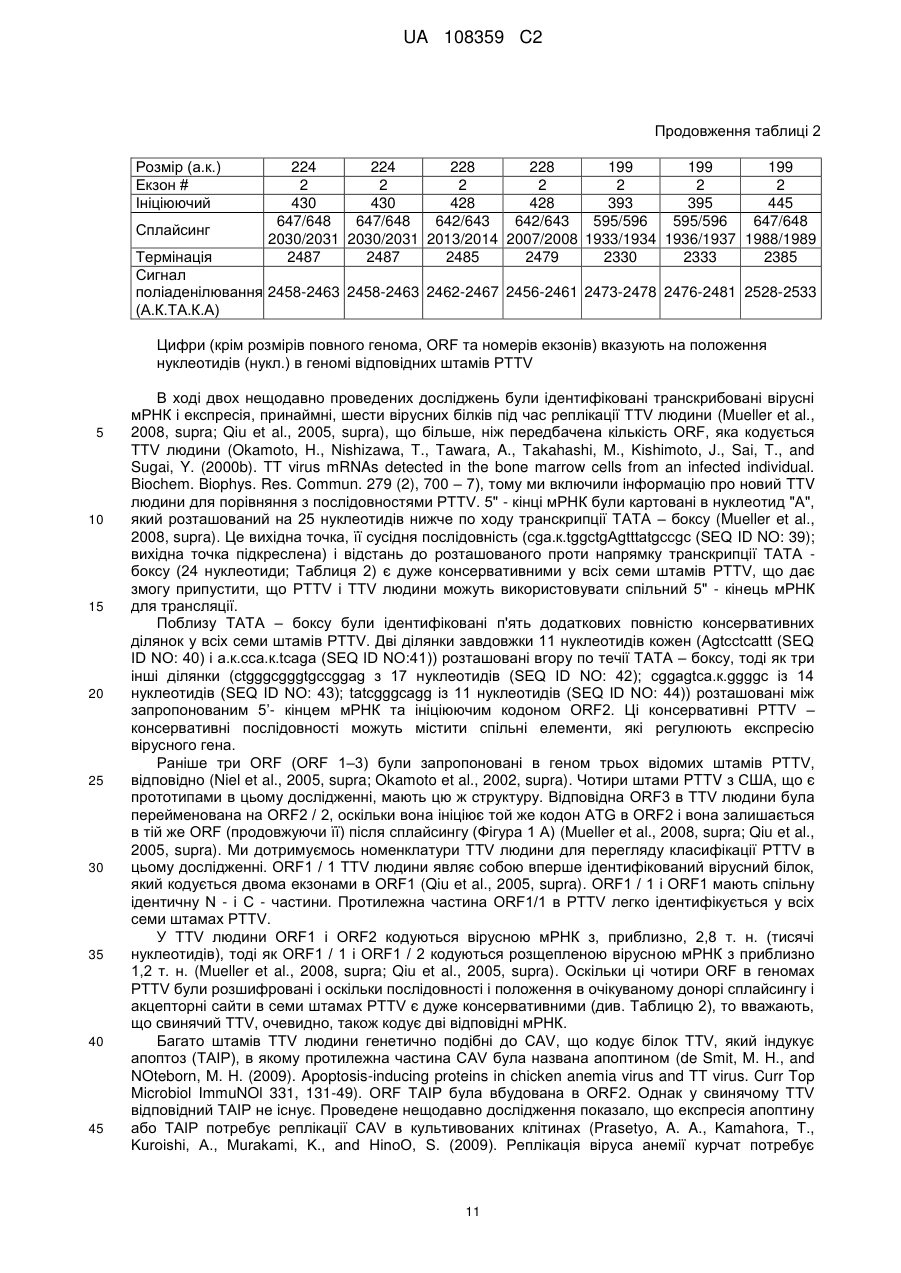
Текст
Реферат: Винахід належить до інфекційної полінуклеотидної молекули, що кодує генотипи або підтипи вірусу Torque teno свиней (PTTV): PTTV1a-VA, PTTV1b-VA, PTTV2b-VA і PTTV2c-VA; до біологічно функціональної плазміди або вірусного вектора, який містить інфекційну нуклеотидну UA 108359 C2 (12) UA 108359 C2 геномну послідовність; до вакцини, що містить PTTV; до способу діагностики PTTV інфекції за допомогою полімеразної ланцюгової реакції (ПЛР) з використанням специфічних праймерів для PTTV1, PTTV2 і генотипів індивідуального PTTV1. UA 108359 C2 5 10 15 20 25 30 35 40 45 50 55 60 Ця заявка претендує на переваги попередньої заявки США на патент № 61/235,833, поданої 21 серпня 2009 р., і попередньої заявки США на патент № 61 / 316,519, поданої 23 березня 2010 р., описи яких включені в дану заявку шляхом посилань. ОБЛАСТЬ ТЕХНІКИ, ДО ЯКОЇ НАЛЕЖИТЬ ДАНИЙ ВИНАХІД Ця заявка належить до вакцин для захисту від інфекції, викликаної свинячим вірусом Torque Teno (Торкве Тено) (TTV), та інфекцій, викликаних інфекційними ДНК, - клонами свинячого TTV (РTTV), та до їхнього застосування. Цей винахід належить також до діагностики інфекції, викликаної свинячим вірусом Torque Teno (РTTV), особливо інфекції, викликаної різними типами та типоспецифічними вірусами РTTV, та інфекції, викликаної багатьма штамами різних генотипів. РІВЕНЬ ТЕХНИКИ Вірус Torque Teno (TTV) вперше був виявлений у 1997 р. в Японії у пацієнта з посттрансфузійним гепатитом не - А і не - Е типу (Nishizawa, T., Okamoto, H., Konishi, K., Yoshizawa, H., Miyakawa, Y., and Mayumi, M. (1997). A novel DNA virus (TTV) associated with elevated transaminase levels in posttransfusion hepatitis of unknown etiology. Biochem Biophys Res Commun 241(1), 92-7). З того часу була ідентифікована велика кількість штамів TTV людини та дві групи TTV – споріднених вірусів, названих як вірус Torque teno mini (TTMV) і вірус Torque teno midi (TTMDV), з високими показниками поширення в сироватці та інших тканинах здорових людей (Hino, S., and Miyata, H. (2007). Torque teno virus (TTV): current status. Rev. Med. Virol. 17(1), 45-57; Okamoto, H. (2009a). History of discoveries and pathogenicity of TT viruses. Curr. Top. Microbiol. Immunol 331, 1-20). TTV, TTMV і TTMDV людини є сферичними вірусами без оболонки, які містять геноми кільцевої однониткової ДНК (онДНК) завдовжки 3,6-3,9, 2,8-2,9 та 3,2 т.н., відповідно, і тепер вони класифікуються в нову родину вірусів Anelloviridae в International Committee on Taxonomy of Viruses (ICTV; http://www.ictvonline.org/virusTaxoNOmy.asp?bhcp=1) (Biagini, P. (2009). Classification of TTV and related viruses (anelloviruses). Curr. Top. Microbiol. Immunol. 331, 21-33). Ці три групи вірусів, що належать до TTV, виявляють високий ступінь гетерогенності, при цьому кожна група складається з сукупності геногруп та генотипів (Biagini, P., Gallian, P., Cantaloube, J.F., Attoui, H., de Micco, P., and de Lamballerie, X. (2006). Distribution and genetic analysis of TTV and TTMV major phylogenetic groups in French blood donors. J. Med. Virol. 78 (2), 298-304; Jelcic, I., Hotz-Wagenblatt, A., Hunziker, A., Zur Hausen, H., and de Villiers, E.M. (2004). Isolation of multiple TT virus genotypes from spleen biopsy tissue from a Hodgkin's disease patient: genome reorganization and diversity in the hypervariable region. J. Virol. 78(14),7498-507). Є повідомлення про поширення множинних інфекцій, викликаних TTV з різними генотипами, а також подвійних чи потрійних інфекцій, викликаних TTV, TTMV і TTMDV, у людей, і ці інфекції розглядаються як звичайні події у здорових людей (Niel, C., Saback, F.L., and Lampe, E. (2000) ). Коінфекція, викликана багатьма штамами TTV, що належать до різних генотипів, розповсюджена у здорових дорослих людей в Бразилії (J. Clin. Microbiol. 38(5), 192630; Ninomiya, M., Takahashi, M., Hoshino, Y., Ichiyama, K., Simmonds, P., and Okamoto, H. (2009). Analysis of the entire genomes of torque teno midi virus variants in chimpanzees: infrequent crossspecies infection between humans and chimpanzees. J. Gen. Virol. 90 (Pt 2), 347-58; Okamoto, H. (2009a). History of discoveries and pathogenicity of TT viruses. Curr. Top. Microbiol. Immunol. 331, 120; Takayama, S., Miura, T., Matsuo, S., Taki, M., and Sugii, S. (1999). Prevalence and persistence of a Novel DNA TT virus (TTV) infection in Japanese haemophiliacs. Br. J. Haematol. 104 (3), 626-9). TTV викликають інфекцію не лише у людей, але й також у різних видів тварин, включаючи інших, ніж людина, приматів, тупай, свиней, худобу, кішок, собак та морських левів (Biagini, P., Uch, R., Belhouchet, M., Attoui, H., Cantaloube, J. F., Brisbarre, N., and de Micco, P. (2007). Circular genomes related to anelloviruses identified in human and animal samples by using a combined rolling-circle amplification / sequence-independent single primer amplification approach. J. Gen. Virol. 88(Pt 10), 2696-701; Inami, T., Obara, T., Moriyama, M., Arakawa, Y., and Abe, K. (2000). Full-length nucleotide sequence of a simian TT virus isolate obtained from a chimpanzee: evidence for a new TT virus-like species. Virology 277(2), 330-5; Ng, T. F., Suedmeyer, W. K., Wheeler, E., Gulland, F., and Breitbart, M. (2009). novel anellovirus discovered from a mortality event of captive California sea lions. J. Gen. Virol.90 (Pt 5), 1256-61; Okamoto, H. (2009b). TT viruses in animals. Curr. Top. Microbiol. Immunol. 331, 35-52; Okamoto, H., Nishizawa, T., Takahashi, M., Tawara, A., Peng, Y., Kishimoto, J., and Wang, Y. (2001). Genomic and evolutionary characterization of TT virus (TTV) in tupaias and comparison with species-specific TTV in humans and non-human primates. J Gen Virol. 82(Pt 9), 2041-50; Okamoto, H., Nishizawa, T., Tawara, A., Peng, Y., Takahashi, M., Kishimoto, J., Tanaka, T., Miyakawa, Y., and Mayumi, M. (2000a). Species-specific TT viruses in humans and nonhuman primates and their phylogenetic relatedness. Virology 277(2), 368-78; Okamoto, H., Takahashi, M., Nishizawa, T., Tawara, A., Fukai, K., Muramatsu, U., Naito, Y., and Yoshikawa, A. (2002). Genomic 1 UA 108359 C2 5 10 15 20 25 30 35 40 45 50 55 60 characterization of TT viruses (TTV) in pigs, cats and dogs and their relatedness with species-specific TTV in primates and tupaias. J. Gen. Virol. 83 (Pt 6), 1291-7). Кріме того, шимпанзе також інфікується TTMV і TTMDV (Ninomiya, M., Takahashi, M., Hoshino, Y., Ichiyama, K., Simmonds, P., and Okamoto, H. (2009). Analysis of the entire genomes of torque teno midi virus variants in chimpanzees: infrequent cross-species infection between humans and chimpanzees. J. Gen. Virol. 90(Pt 2), 347-58; Okamoto et al., 2000a, supra). Хоча розміри геномів ідентифікованих штамів TTV у тварин, особливо TTV у тварин, що не є приматами, порівняно менші, ніж у штамів TTV людини, вони мають одну й ту ж структуру генома, як мінімум, з двома відкритими рамками зчитування, які частково перекриваються (ORF1 та ORF2), трансльованими з негативної ДНК, а також з коротким відрізком нетрансльованої ділянки (UTR) з високим вмістом (близько 90 %) GC (гуаніну та цитозину) (Okamoto, 2009b, supra). Конструкція ORF TTV дуже подібна до конструкції віруса анемії курчат (CAV), що належить до роду Gyrovirus з родини Circoviridae, але відрізняється від конструкції свинячих цирковірусів (PCV) типів 1 (PCV1) і 2 (PCV2), які також відносять до цієї ж родини (Davidson, I., and Shulman, L. M. (2008). Unraveling the puzzle of human anellovirus infections by comparison with avian infections with the chicken anemia virus. Virus Res. 137(1), 1-15; HiNO, S., and Prasetyo, A. A. (2009). Relationship of Torque teno virus to chicken anemia virus. Curr. Top Microbiol. Immunol. 331, 117-30). Геноми PCV1 і PCV2 є амбісмисловими, причому ORF1 закодована геномною ниткою, а ORF2 закодована антигеномною ниткою (Hino and Miyata, 2007, supra). Нещодавно шаблон для транскрипції та трансльовані продукти обох генотипів 1 і 6 TTV людини були ідентифіковані шляхом трансфекції відповідних інфекційних ДНК – клонів TTV у культивовані клітини (Mueller, B., Maerz, A., Doberstein, K., Finsterbusch, T., and Mankertz, A. (2008). Gene expression of the human Torque Teno Virus isolate P/1C1. Virology 381(1), 36-45; Qiu, J., Kakkola, L., Cheng, F., Ye, C., Soderlund-Venermo, M., Hedman, K., and Pintel, D. J. (2005)). (Mueller, B., Maerz, A., Doberstein, K., Finsterbusch, T., and Mankertz, A. (2008). Gene expression of the human Torque Teno Virus isolate P/1C1. Virology 381(1), 36-45; Qiu, J., Kakkola, L., Cheng, F., Ye, C., Soderlund-Venermo, M., Hedman, K., and Pintel, D. J. (2005). Цирковірус TTV людини генотипу 6 експресує шість білків після трансфекції клону, що має повну довжину (J. Virol. 79(10), 6505-10). Експресія щонайменше шести білків, позначених як ORF1, ORF2, ORF1 / 1, ORF2 / 2, ORF1 / 2 і ORF2 / 3, із трьох або більше сплайсованих мРНК була описана Kakkola, L., Hedman, K., Qiu, J., Pintel, D., and Soderlund-Venermo, M. (2009). Replication of and protein synthesis by TT viruses. Curr. Top Microbiol. Immunol. 331, 53-64; Mueller et al., 2008, supra; Qiu et al., 2005, supra). Відповідно, є ймовірним, що, коли стануть доступними додаткові дані стосовно тваринного ТТV, очікувану структуру геному тваринного ТТV можна буде модифікувати. Не зважаючи на те, що ТТV вперше був ідентифікований у пацієнта з криптогенним гепатитом, виконані після цього дослідження не дали змоги отримати докази істотної ролі TTV у патогенезі гепатиту або інших захворювань (Hino and Miyata, 2007, supra; Maggi, F., and Bendinelli, M. (2009). Immunobiology of the Torque teNO viruses and other anelloviruses. Curr. Top Microbiol. Immunol. 331, 65-90; Okamoto, 2009a, supra)). Попри те, що вважають, що TTV людини не пов'язаний безпосередньо із захворюванням, нещодавно було встановлено, що свинячий TTV (PTTV) частково сприяє експериментальній індукції свинячого дерматиту та синдрому нейропатії (PDNS), у поєднанні з виникненням репродуктивно-респіраторного синдрому у свиней (PRRSV) (Krakowka, S., Hartunian, C., Hamberg, A., Shoup, D., Rings, M., Zhang, Y., Allan, G., and Ellis, J. A. (2008). Evaluation of induction of porcine dermatitis and nephropathy syndrome in gnotobiotic pigs with negative results for porcine circovirus type 2. Am. J. Vet. Res. 69(12), 1615-22), а також з експериментальною індукцією мультисистемного синдрому виснаження після відлучення поросят від свиноматки (PMWS), у поєднанні з інфекцією, викликаною PCV2 в моделі гнотобіотичної свині (Ellis, J. A., Allan, G., and Krakowka, S. (2008). Effect of coinfection with genogroup 1 porcine torque teno virus on porcine circovirus type 2-associated postweaning multisystemic wasting syndrome in gnotobiotic pigs. Am. J. Vet. Res. 69(12), 1608-14). Ці дані дали змогу висловити припущення, що свинячий вірус TTV є патогенним для свиней. Однак необхідні поглиблені дослідження біологічно чистої форми віруса PТТV для повної характеристики захворювань та патологічних змін, обумовлених інфекцією, викликаною PТТV. У порівнянні з ТТV людини, інформація про геном РТТV дуже обмежена. Наразі описані лише одна геномна послідовність з повною довжиною та дві геномні послідовності з майже повною довжиною для РТТV у свиней в Японії та в Бразилії, відповідно (Niel, C., Diniz-Mendes, L., and Devalle, S. (2005). Rolling-circle amplification of Torque teno virus (TTV) complete genomes from human and swine sera and identification of a novel swine TTV genogroup. J. Gen. Viro.l 86(Pt 5), 1343-7; Okamoto et al., 2002, supra.). Серед трьох відомих штамів PTTV були згруповані разом штами Sd-TTV31 і TTV-1p в геногрупу 1 (PTTV1). Натомість TTV-2p був єдиним штамом, 2 UA 108359 C2 5 10 15 20 25 30 35 40 45 50 55 60 віднесеним до геногрупи 2 (PTTV2) (Niel et al., 2005, supra). Однак класифікація в геногрупах складає "розмиту" концепцію в таксономії вірології, тому необхідна розробка наступної, більш точної класифікації PTTV, однак цього можна досягти лише тоді, коли стануть доступними інші геномні послідовності з повною довжиною нових штамів PTTV, що становлять собою численні генотипи. Раніше було показано, що інфекції, викликані PTTV, широко розповсюджені у свиней в шести країнах, включаючи США, Канаду, Іспанію, Китай, Корею і Таїланд (McKeown, N.E., Fenaux, M., Halbur, P.G., and Meng, X.J. (2004). Molecular characterization of porcine TT virus, an orphan virus, in pigs from six different countries. Vet. Microbiol. 104 (1-2), 113-7). Питання про те, чи відіграють TTV істотну роль в патогенезі різних захворювань у свиней, досі залишається суперечливим. На прикладі моделі гнотобіотичної свині було показано, що сама собою інфекція, викликана PTTV1, не призводить до розвитку будь-яких клінічних захворювань, однак спричиняє патологічні зміни (Krakowka, S. and Ellis, J.A., 2008. Evaluation of the effects of porcine genogroup 1 torque teno virus in gnotobiotic swine. Am. J. Vet. Res. 69, 16239). У свиней, експериментально заражених як PTTV1, так і вірусом репродуктивно – респіраторного синдрому у свиней (PRRSV), развивались клінічний свинячий дерматит та синдром нефропатії (PDNS) (Krakowka, S., Hartunian, C., Hamberg, A., Shoup, D., Rings, M., Zhang, Y., Allan, G. and Ellis, J.A., 2008. Evaluation of induction of porcine dermatitis and nephropathy syndrome in gnotobiotic pigs with negative results for porcine circovirus type 2. Am. J. Vet. Res. 69, 1615-22), в той час, як у свиней, заражених як PTTV1, так і свинячим цирковірусом типу 2 (PCV2), спостерігався хронічний мультисистемний синдром виснаження після відлучення поросят від свиноматки (PMWS) (Ellis et al., 2008, supra). Хоча PCV2 розглядається як агент, що в основному викликає клінічний PMWS або захворювання, які асоціюються з PCV (PCVAD), в Іспанії спостерігалось значне розповсюдження інфекції, викликаної PTTV2, у свиней, які потерпали від PMWS, з низьким вмістом PCV2 або відсутністю PCV2, у порівнянні зі свинями без PMWS (Kekarainen et al., 2006, supra). Всі ці дані дають змогу висловити припущення, що свинячі TTV можуть слугувати кофакторами, які беруть участь в ініціації та прискоренні розвитку захворювань у свиней. Свинячий TTV був виявлений у зразках свинячої сироватки, фекалій, слини, сперми і тканин у інфікованих свиней, що свідчить про те, що він передається різними шляхами, включаючи вертикальну та горизонтальну трансмісію (Kekarainen et al., 2007, supra; Pozzuto, T., Mueller, B., Meehan, B., Ringler, S.S., McIntosh, K.A., Ellis, J.A., Mankertz, A. and Krakowka, S., 2009. In utero transmission of porcine torque teno viruses. Vet. Microbiol. 137, 375-9; Sibila, M., Martinez-GuiNO, L., Huerta, E., Llorens, A., Mora, M., Grau-Roma, L., Kekarainen, T. and Segales, J., 2009. Swine torque teno virus (TTV) infection and excretion dynamics in conventional pig farms. Vet. Microbiol. 139, 2138). Однак наразі виявлення інфекції, викликаної TTV, ґрунтується на застосуванні звичайного методу ПЛР. Таким чином, досі не були розроблені ні метод серологічного аналізу, ні система вірусної культури. Зокрема, методи "вкладеної ПЛР" з ампліфікацією консервативних ділянок в UTR PTTV1 і PTTV2, відповідно, розроблені групою іспанських дослідників, знайшли широке застосування (Kekarainen et al., 2006, supra). Оскільки показник розповсюдження віруса, очевидно, пов'язаний зі ступенем прояву клінічних захворювань, як свідчить PCVAD, обумовлений PCV2 (Opriessnig, T., Meng, X.J. and Halbur, P.G., 2007. Porcine circovirus type 2 associated disease: update on current terminology, clinical manifestations, pathogenesis, diagnosis, and intervention strategies. J. Vet. Diagn. Invest. 19, 591-615), було б важливішим визначити вірусне навантаження свинячого TTV за допомогою методу кількісної ПЛР в режимі реального часу, ніж наявність ДНК в TTV шляхом застосування методу звичайної ПЛР. Крім того, ПЛР в режимі реального часу є більш надійним, швидким і дешевшим методом у порівнянні з методом звичайної ПЛР. Нещодавно були описані два методи ПЛР в режимі реального часу, які основані на застосуванні TaqMan-зонду, для виявлення та кількісного визначення двох видів свинячого TTV (Brassard, J., Gagne, M.J., Houde, A., Poitras, E. and Ward, P., 2009. Development of a realtime TaqMan PCR assay for the detection of porcine and bovine Torque teno virus. J. Appl. Microbiol. Nov 14, 2009, E pub. ahead of print; Gallei, A., Pesch, S., Esking, W.S., Keller, C. and Ohlinger, V.F., 2009. Porcine Torque teno virus: Determination of viral genomic loads by genogroup-specific multiplex rt-PCR, detection of frequent multiple infections with genogroups 1 or 2, and establishment of viral full-length sequences. Vet. Microbiol. Dec 21, 2009, E pub. ahead of print). Основний недолік методів, основаних на застосуванні зондів, полягає в тому, що можуть бути отримані хибні негативні результати, якщо послідовності, що зв'язують зонди, містять мутації (Anderson, T.P., WerNO, A.M., BeyNOn, K.A. and Murdoch, D.R., 2003. Failure to genotype herpes simplex virus by real-time PCR assay and melting curve analysis due to sequence variation within probe binding sites. J. Clin. Microbiol. 41, 2135-7). 3 UA 108359 C2 5 10 15 20 25 30 35 40 45 50 55 З урахуванням високого ступеня гетерогенності серед послідовностей відомих штамів свинячого TTV, очікують варіювання послідовностей, які зв'язують зонд, у польових штамах PTTV. Метод ПЛР в режимі реального часу, оснований на застосуванні барвника SYBR Green, є альтернативним методом, який дає змогу, не зважаючи на його досить низьку специфічність, уникнути виникнення цієї проблеми та забезпечити універсальний шлях виявлення та кількісного визначення варіантів свинячого TTV. Понад те, аналіз кривих плавлення (МСА) після проведення ПЛР в режимі реального часу, основаної на застосуванні барвника SYBR Green, забезпечує специфічність реакції та дає змогу провести мультиплексне виявлення різних типів віруса (Ririe, K.M., Rasmussen, R.P. and Wittwer, C.T., 1997. Product differentiation by analysis of DNA melting curves during the polymerase chain reaction. Anal. Biochem. 245, 154-60). Методи ПЛР в режимі реального часу, що базуються на застосуванні барвника SYBR Green, були успішно застосовані щодо різних вірусів людини та тварин (Gibellini, D., Gardini, F., Vitone, F., Schiavone, P., Furlini, G. and Re, M.C., 2006. Simultaneous detection of HCV and HIV-1 by SYBR Green real time multiplex RT-PCR technique in plasma samples. Mol. Cell Probes 20, 223-9; Martinez, E., Riera, P., Sitja, M., Fang, Y., Oliveira, S. and Maldonado, J., 2008. Simultaneous detection and genotyping of porcine reproductive and respiratory syndrome virus (PRRSV) by real-time RT-PCR and amplicon melting curve analysis using SYBR Green. Res. Vet. Sci. 85, 184-93; Mouillesseaux, K.P., Klimpel, K.R. and Dhar, A.K., 2003. Improvement in the specificity and sensitivity of detection for the Taura syndrome virus and yellow head virus of penaeid shrimp by increasing the amplicon size in SYBR Green real-time RT-PCR. J. Virol. Methods 111, 121-7; Wilhelm, S., Zimmermann, P., Selbitz, H.J. and Truyen, U., 2006. Real-time PCR protocol for the detection of porcine parvovirus in field samples. J. Virol. Methods 134, 257-60). Наразі мало відомо про PTTV–специфічну гуморальну відповідь. Оскільки методи із застосуванням ПЛР не відображають розвиток інфекції, викликаної PTTV, у свиней, для оцінки переважання серотипу PTTV та характеристики участі PTTV в розвитку захворювань у свиней необхідно застосовувати ефективний метод твердофазного імуноферментного аналізу (ELISA). Таким чином, субодинична, інактивована або жива вакцина від TTV свиней відсутня. Бажано та переважно здійснити експресію рекомбінантних капсидних білків PTTV з різних генотипів з метою створення субодиничних вакцин та отримання інфекційних молекулярних ДНК - клонів PTTV з різних генотипів для розповсюдження біологічно чистої форми PTTV в системі клітинної культури, яка застосовується для створення інактивованих або живих вакцин. РОЗКРИТТЯ ВИНАХОДУ Даний винахід передбачає молекулу інфекційної нуклеїнової кислоти ("клон інфекційної ДНК") свинячого віруса Torque teno (PTTV), що являє собою молекулу нуклеїнової кислоти, яка кодує інфекційний PTTV, яка містить, щонайменше, одну копію геномної послідовності, що має ступінь гомологічності, який становить, принаймні, 80 % стосовно геномної послідовності, обраної з групи, що складається з генотипів PTTV1a-VA, PTTV1b-VA, PTTV2b-VA та PTTV2c-VA. Згідно з одним аспектом даного винаходу передбачені клони інфекційної ДНК PTTV за п. 1 формули винаходу, які характеризуються тим, що геномна послідовність обрана з послідовностей, що описуються в SEQ ID NO: 9, SEQ ID NO: 10, SEQ ID NO: 11 і SEQ ID NO: 12. Цей винахід передбачає також біологічно функціональну плазміду або вірусний вектор, що містять інфекційні геноми PTTV. Цей винахід передбачає прийнятну клітину-господаря, трансфіковану плазмідою чи вірусним вектором клону інфекційної ДНК. Цей винахід передбачає інфекційний PTTV, який продукується клітинами, трансфікованими клонами інфекційної ДНК PTTV. Цей винахід належить також до вірусної вакцини, яка містить нетоксичний фізіологічно прийнятний носій та імуногенну кількість члена, обраного з групи, яка складається з: (а) молекули нуклеїнової кислоти, яка містить щонайменше одну копію геномної послідовності, що має ступінь гомологічності, який становить, принаймні, 80 % по відношенню до геномної послідовності, обраної з групи, що складається з генотипів PTTV1a-VA, PTTV1b-VA, PTTV2b-VA та PTTV2c-VA або її комплементарну нитку, (б) біологічно функціональної плазміди або вірусного вектора, який містить молекулу нуклеїнової кислоти, що несе принаймні одну копію геномної послідовності, що має ступінь гомологічності, який становить, принаймні, 80 % по відношенню до геномної послідовності, обраної з групи, що складається з генотипів PTTV1a-VA, PTTV1b-VA, PTTV2b-VA та PTTV2c – VA, або її комплементарну нитку, і (в) авірулентного інфекційного непатогенного PTTV, який включає, щонайменше, одну копію геномної послідовності, що має ступінь гомологічності, який становить, принаймні, 80 % по відношенню 4 UA 108359 C2 5 10 15 20 25 30 35 40 45 50 55 60 до геномної послідовності, обраної з групи, що складається з генотипів PTTV1a-VA, PTTV1b-VA, PTTV2b-VA та PTTV2c-VA. Відповідно до одного аспекту даного винаходу вакцина містить живий вірус PTTV, отриманий з клонів інфекційного PTTV. Згідно з іншим аспектом даного винаходу вакцина містить інактивований вірус PTTV, отриманий з клонів інфекційного PTTV. Цей винахід передбачає очищені рекомбінантні білки, експресовані з генів капсиду ORF1 генотипів PTTV або підтипів PTTV1a-VA, PTTV1b-VA та PTTV2c-VA в бактеріальну систему для експресії та застосування цих рекомбінантних капсидних білків як субодиничної вакцини проти інфекцій, викликаних PTTV. Згідно з одним із варіантів даного винаходу, рекомбінантні капсидні білки, що застосовуються в складі субодиничних вакцин, експресуються в системі експресії бакуловірусів та в інших векторних системах експресії. Згідно із ще одним аспектом даного винаходу вакцина додатково містить ад'ювант. Даний винахід передбачає також спосіб імунізації свині проти вірусної інфекції, викликаної PTTV, який включає введення свині імунологічно ефективної кількості вірусної вакцини. Згідно із ще одним аспектом даного винаходу зазначений спосіб включає введення свині рекомбінантного субодиничного капсидного білка, молекули інфекційної нуклеїнової кислоти або живого віруса PTTV. Відповідно до іншого аспекту даного винаходу зазначений спосіб включає парентеральне, інтраназальне, інтрадермальне або трансдермальне введення вакцини свині. Цей винахід передбачає також виділений полінуклеотид, що складається з нуклеотидної послідовності PTTV1a – VA, представленої в SEQ ID NO: 9. Цей винахід передбачає також виділений полінуклеотид, що складається з нуклеотидної послідовності PTTV1b – VA, представленої в SEQ ID NO: 10. Цей винахід передбачає також виділений полінуклеотид, що складається з нуклеотидної послідовності PTTV2b –VA, представленої в SEQ ID NO: 11. Цей винахід передбачає також виділений полінуклеотид, який складається з нуклеотидної послідовності PTTV2c – VA, представленої в SEQ ID NO: 12. Цей винахід передбачає також субодиничну вакцину, що містить імуногенний фрагмент поліпептидної послідовності або повного білка, трансльований відповідно до полінуклеотидної послідовності, обраної з групи, що складається з ORF1, ORF2, ORF1 / 1 і ORF2 / 2 генотипів PTTV або підтипів PTTV1a-VA, PTTV1b-VA, PTTV2b-VA і PTTV2c-VA, зокрема, ORF 1, що кодує капсидний білок. Відповідно до ще одного аспекту даного винаходу полінуклеотидна послідовність обрана з групи, яка складається з ORF1 генотипів PTTV або підтипів PTTV1a-VA, PTTV1b-VA, PTTV2b-VA та PTTV2c-VA. Відповідно до іншого аспекту даного винаходу полінуклеотидна послідовність становить собою ORF 1 генотипів PTTV1a. Відповідно до ще одного аспекту даного винаходу полінуклеотидна послідовність становить собою ORF 1 генотипів PTTV1b. Відповідно до ще одного аспекту даного винаходу полінуклеотидна послідовність становить собою ORF 1 підтипів PTTV2c – VA. Відповідно до одного аспекту даного винаходу поліпептидна послідовність обрана з групи, яка складається з послідовностей, представлених в SEQ ID NО: 13, SEQ ID NO: 14, SEQ ID NО: 15, SEQ ID NО: 16, SEQ ID NО: 17, SEQ ID NО: 18, SEQ ID NО: 19, SEQ ID NО: 20, SEQ ID NО: 21, SEQ ID NО: 22, SEQ ID NО: 23, SEQ ID NО: 24, SEQ ID NО: 25, SEQ ID NО: 26, SEQ ID NО: 27 і SEQ ID NО: 28. Відповідно до іншого аспекту даного винаходу поліпептидна послідовність становить собою SEQ ID NО: 13. Відповідно до іншого аспекту даного винаходу поліпептидна послідовність становить собою SEQ ID NО:14. Відповідно до іншого аспекту даного винаходу поліпептидна послідовність становить собою SEQ ID NО: 16. Згідно з одним конкретним варіантом даного винаходу поліпептидна послідовність становить собою С – термінальну ділянку (а.к. 310 – 625) SEQ ID NО: 16. Згідно з іншим аспектом даного винаходу поліпептидна послідовність становить собою SEQ ID NО: 20. Відповідно до іншого аспекту даного винаходу вакцина додатково містить ад'ювант. Цей винахід передбачає також спосіб імунізації свині проти вірусної інфекції, викликаної PTTV, який включає введення свині імунологічно ефективної кількості вірусної вакцини, що містить імуногенний фрагмент поліпептидної послідовності або повного білка, трансльований відповідно до полінуклеотидної послідовності, обраної з групи, що складається з ORF1, ORF 2, ORF1 / 1 і ORF2 / 2 генотипів PTTV або підтипів PTTV1a-VA, PTTV1b-VA, PTTV2b-VA та PTTV2c – VA. 5 UA 108359 C2 5 10 15 20 25 30 35 40 45 50 55 Згідно з іншим аспектом даного винаходу вказаний спосіб включає введення свині імуногенного фрагмента чи рекомбінантного капсидного білка. Згідно з іншим аспектом даного винаходу вказаний спосіб включає парентеральне, інтраназальне, інтрадермальне або трансдермальне введення вакцини свині. Згідно із ще одним аспектом даного винаходу вказаний спосіб включає введення свині вакцини в лімфу або внутрішньом'язово. Цей винахід належить також до способу діагностики інфекції, викликаної PTTV1 та кількісного визначення вірусного навантаження PTTV1, який включає екстрагування ДНК із зразка, здогадно інфікованого PTTV1, проведення полімеразної ланцюгової реакції (ПЛР) за допомогою праймерів, які містять послідовності, представлені в SEQ ID NO: 29 та SEQ ID NO: 30, і детектування специфічної ампліфікації PTTV1. Відповідно до одного із аспектів даного винаходу полімеразна ланцюгова реакція являє собою ПЛР в режимі реального часу із застосуванням барвника SYBR Green. Цей винахід передбачає також спосіб діагностики інфекції, викликаної PTTV2, та кількісного визначення вірусного навантаження PTTV2, який включає екстрагування ДНК із зразка, здогадно інфікованого PTTV2, проведення полімеразної ланцюгової реакції (ПЛР) за допомогою праймерів, які містять послідовності, представлені в SEQ ID NO: 29, SEQ ID NO: 30, SEQ ID NO: 31 і SEQ ID NO: 32, та детектування специфічної ампліфікації PTTV2. Згідно з одним із аспектів даного винаходу полімеразна ланцюгова реакція являє собою ПЛР в режимі реального часу із застосуванням барвника SYBR Green. Цей винахід передбачає також спосіб одночасного детектування та діагностики інфекції, викликаної PTTV1 і PTTV2, який включає екстрагування ДНК із зразка, здогадно інфікованого PTTV, проведення полімеразної ланцюгової реакції (PCR) за допомогою праймерів, які містять послідовності, представлені в SEQ ID NO: 31 та SEQ ID NO: 32, і детектування специфічної ампліфікації PTTV1 та PTTV2. Згідно з одним із аспектів даного винаходу полімеразна ланцюгова реакція являє собою ПЛР в режимі реального часу із застосуванням барвника SYBR Green. Крім того, цей винахід передбачає також спосіб одночасного детектування та діагностики інфекції, викликаної PTTV1а и PTTV1b, який включає екстрагування ДНК із зразка, здогадно інфікованого PTTV1, проведення першої полімеразної ланцюгової реакції (ПЛР) за допомогою праймерів, що містять послідовності, представлені в SEQ ID NO: 33 і SEQ ID NO: 34, проведення другої полімеразної ланцюгової реакції (ПЛР) за допомогою праймерів, що містять послідовності, представлені в SEQ ID NO: 35, SEQ ID NO: 36, SEQ ID NO: 37 та SEQ ID NO: 38, і детектування специфічної ампліфікації PTTV1а та PTTV1b. Цей винахід передбачає також спосіб діагностики інфекції, викликаної PTTV, який включає імобілізацію імуногенного фрагмента поліпептидної послідовності, трансльований відповідно до полінуклеотидної послідовності, обраної з групи, що складається з ORF1, ORF2, ORF1 / 1 і ORF2 / 2 генотипів PTTV або підтипів PTTV1a-VA, PTTV1b-VA, PTTV2b-VA і PTTV2c – VA; контактування зразка сироватки свині, здогадно інфікованої PTTV, з імобілізованим імуногенним фрагментом і детектування захопленого антитіла, специфічного до імуногенного фрагмента. Згідно з одним аспектом даного винаходу полінуклеотидна послідовність, обрана з групи, яка складається з ORF1 генотипів PTTV або підтипів PTTV1a-VA, PTTV1b-VA, PTTV2b-VA і PTTV2c – VA. Згідно з одним аспектом даного винаходу полінуклеотидна послідовність являє собою ORF1 генотипу PTTV1a – VA віруса PTTV. Згідно з іншим аспектом даного винаходу полінуклеотидна послідовність являє собою ORF1 генотипу PTTV1b – VA віруса PTTV. Згідно з ще одним аспектом даного винаходу полінуклеотидна послідовність являє собою ORF1 підтипу PTTV2с – VA віруса PTTV. Згідно з іншим аспектом даного винаходу поліпептидна послідовність обрана з групи, яка складається з послідовностей, представлених SEQ ID NO: 13, SEQ ID NO: 14, SEQ ID NO: 15, SEQ ID NO: 16, SEQ ID NO: 17, SEQ ID NO: 18, SEQ ID NO: 19, SEQ ID NO: 20, SEQ ID NO: 21, SEQ ID NO: 22, SEQ ID NO: 23, SEQ ID NO: 24, SEQ ID NO: 25, SEQ ID NO: 26, SEQ ID NO: 27 і SEQ ID NO: 28. Згідно з одним варіантом даного винаходу поліпептидна послідовність представлена в SEQ ID NО: 13. Згідно з іншим варіантом даного винаходу поліпептидна послідовність представлена в SEQ ID NО: 14. Згідно з одним варіантом даного винаходу поліпептидна послідовність представлена в SEQ ID NО: 16. Згідно з одним конкретним варіантом даного винаходу поліпептидна послідовність являє собою С-термінальну ділянку (а. к. 310 – 625) SEQ ID NO: 16. 6 UA 108359 C2 5 10 15 20 25 30 35 40 45 50 55 Згідно з іншим варіантом даного винаходу поліпептидна послідовність являє собою SEQ ID NO: 20. Цей винахід передбачає три стандартизовані методи твердофазного імуноферментного аналізу (ELISA) для діагностики інфекцій, викликаних PTTV та детектування антитіл в сироватці свиней, які інфіковані генотипами PTTV1a-VA, PTTV1b – VA віруса PTTV та всіма відомими підтипами PTTV типу 2. Діагностичні тести ELISA основані на застосуванні експресованого бактеріями або експресованого бакуловірусами ORF1 капсидного білка генотипів PTTV1a-VA, PTTV1b-VA і PTTV2c – VA віруса PTTV. Згідно з іншим аспектом даного винаходу детектування захопленого антитіла здійснюється з використанням методу Вестерн – блоттингу. Згідно з ще одним аспектом даного винаходу детектування захопленого антитіла здійснюється методом твердофазного імуноферментного аналізу (ELISA). КОРОТКИЙ ОПИС РИСУНКІВ Зазначені вище ознаки винаходу будуть більш зрозумілими із наступного докладного опису даного винаходу у поєднанні з кресленнями, на яких: Фігури 1 А та 1 В представляють собою схематичну діаграму геномних структур, стратегії геномного клонування і збирання чотирьох свинячого віруса TTV групи 1 (типу 1) і групи 2 (типу 2), прототипів американського штаму. На фігурі 2 представлено PASC(парне порівняння послідовностей), порівняння нуклеотидних послідовностей 121 штаму TTV, які є в базі даних GenBank. Показані рід, вид, типи, підтипи, варіанти та відповідні відсотки ідентичності нуклеотидних послідовностей. Фігура 3 А ілюструє філогенетичне дерево, сконструйоване методом "об'єднання сусідів", що ґрунтується на застосуванні повнорозмірних геномних нуклеотидних послідовностей. Фігура 3 В ілюструє філогенетичні дерева, сконструйовані на основі передбачених амінокислотних послідовностей ORF1 за допомогою семи штамів свинячого TTV. Фігура 3 С ілюструє філогенетичні дерева, сконструйовані на основі передбачених амінокислотних послідовностей ORF1/ 1 за допомогою семи штамів свинячого TTV. Фігура 3 D ілюструє філогенетичні дерева, сконструйовані на основі передбачених амінокислотних послідовностей ORF2 за допомогою семи штамів свинячого TTV. Фігура 3 E ілюструє філогенетичні дерева, сконструйовані на основі передбачених амінокислотних послідовностей ORF2 / 2 за допомогою семи штамів свинячого TTV. На Фігурі 4 показано вирівнювання повних амінокислотних послідовностей ORF1 серед семи штамів TTV. На Фігурі 5 показано вирівнювання повних амінокислотних послідовностей ORF2 серед семи штамів TTV. Фігура 6 А ілюструє криві плавлення продуктів ПЛР РTTV1 в режимі реального часу після 40 циклів ампліфікацій зразків стандартної матриці (показано блакитним кольором) 20 зразків свинячої сироватки. Фігура 6 В ілюструє криві плавлення продуктів ПЛР РTTV2 в режимі реального часу після 40 циклів ампліфікацій зразків стандартної матриці та 20 зразків свинячої сироватки. На Фігурах 7 А - 7 В представлені результати аналізу кривих плавлення (МСА) продуктів дуплексної ПЛР РTTV1 / РTTV2 із застосуванням барвника SYBR Green в режимі реального часу. Фігура 8 представляє вирівнювання нуклеотидних послідовностей, розташованих на N – кіцевій ділянці очікуваної ORF1 для семи штамів РTTV. На Фігурах 9 А і 9 В представлені профілі гідрофільності та консервативні ділянки чотирьох відомих свинячих TTV2. Фігури 10 А та 10 В ілюструють експресію та очищення рекомбінантного капсидного білка ORF1 РTTV2с. На Фігурах 11 А та 11 В представлені репрезентативні результати аналізу семи зразків свинячої сироватки методом Вестерн – блоттингу. Фігура 12 ілюструє узгодженість результатів аналізу РTTV2-ORF1 методами Вестерн блоттингу та ELISA. На Фігурі 13 наведені графіки Бокса–Віскера, які свідчать про залежність кількості антитіл до РTTV2 в сироватці свиней від вірусного навантаження для 138 свиней з різних джерел. На Фігурі 14 А представлена ретроспективна оцінка вірусного навантаження РTTV2. Фігура 14 В відображає кількість антитіл до капсидного білка РTTV2-ORF1 для 10 свиней, вирощених до досягнення 2 міс. після прибуття. 7 UA 108359 C2 5 10 15 20 25 30 35 Фігури 15 А – 15 С ілюструють стадії експресії та очищення рекомбінантного капсидного білка ORF 1 в РTTV1а і РTTV1b. На Фігурі 16 представлені приклади результатів аналізів РTTV1а - ORF1 методом Вестерн – блоттингу в зразках сироватки, відібраних у свиней на фермі в штаті Вісконсін. ВТІЛЕННЯ ВИНАХОДУ У відповідності з даним винаходом в одному конкретному прикладі зазначені вище чотири нові підтипи свинячого віруса TTV були виділені у одного кнура у Віргинії. На Фігурі 1 А обидва геноми РTTV1 і РTTV2 показані жирним шрифтом, а номери та напрямок чотирьох очікуваних ORF (ORF1, ORF2, ORF1/1 і ORF2/2) вказані стрілками. Показані також ділянки, збагачені GC. Пунктирними лініями в колах А і D показані ділянки, використані для детектування РTTV1 і РTTV2, відповідно, в зразках сироватки і сперми методом "вкладеної ПЛР". Кола В і С, показані пунктирними лініями, показують два фрагменти ПЛР, що перекриваються, для геномного клонування РTTV1, а пунктирні кола Е і F показують два фрагменти ПЛР, що перекриваються, для геномного клонування РTTV2. Місця положення праймерів, використаних у цьому досліді (див. Таблицю 1), також показані у відповідних позиціях. Один зразок сироватки, відібраний у кнура (SR # 5), дав позитивний результат для РTTV1 і РTTV2 у першому раунді ПЛР, що свідчить про велике вірусне навантаження. Він був використаний для подальшого клонування повних геномів РTTV. Несподівано виявилось, що початкові спроби застосування двох наборів праймерів (NG372 / NG373 і NG384 / NG385) у випадку зворотної ПЛР для клонування першого штаму Sd-TTV31 РTTV з метою ампліфікації вірусної геномної ДНК виявились невдалими (Okamoto et al., 2002, supra). Після декількох спроб продукт ПЛР отримано не було. Грунтуючись на вихідній послідовності ділянки А PTTV1 і ділянки D PTTV2, було отримано дві нові пари праймерів (TTV1-If (SEQ ID NO:1)/TTV1-2340R (SEQ ID NO: 2) і TTV1-2311F(SEQ ID NO: 3)/TTV1-IR(SEQ ID NO: 4) з метою ампліфікації ділянок В і С, які перекривають очікуваний геном PTTV1, і дві додаткових пари праймерів (TTV2-IF(SEQ ID NO:5)/TTV22316R(SEQ ID NO:6) і TTV2-GCF(SEQID NO:7)/TTV2-IR(SEQ ID NO:8)) з метою ампліфікації ділянок Е і F, що перекривають очікуваний геном PTTV2, відповідно (Фігура 1 А Таблиця 1). Праймери TTV1-2340R (SEQ ID NO:2) і TTV1-2311F (SEQ ID NO:3) були отримані із загальної послідовності в штамах Sd-TTV31 (Okamoto et al., 2002, supra) і TTV-1p (Niel et al., 2005), яка присутня в штамі TTV-2p PTTV2 (Niel et al., 2005, supra), в той час як праймери TTV22316R(SEQID NO:6) і TTV2-GCF(SEQID NO:7) були отримані з послідовності штаму TTV-2p, яка відсутня в двох штамах PTTV1. Кожен з отриманих чотирьох різних продуктів ПЛР з очікуваними розмірами був інсертований в клонуючий вектор з тупим кінцем, а отримані рекомбінантні плазміди були трансформовані в Escherichia coli. Були ідентифіковані від восьми до п'ятнадцяти позитивних (білого кольору) клонів для кожного конструкта, що являють собою фрагменти В, С, Е і F, після чого вони були секвеновані. Таблиця 1 Олігонуклеотидні праймери, використані для проведення вкладеної ПЛР та ПЛР-ампліфікації геномів свинячих вірусів TT Праймер TTV1-mF (EQ ID NO:45) TTV1-mR (SEQ ID NO:46) TTV1-nF (SEQ ID NO:47) TTV1-nR (SEQ ID NO:48) TTV2-mF (SEQ ID NO:49) TTV2-mR (SEQ ID NO:50) TTV2-nF (SEQ ID NO:51) Послідовність (від 5" до 3") TACACTTCCGGGTTCAGGAGGCT ACTCAGCCATTCGGА.К.CCTCAC CА.К.TTTGGCTCGCTTCGCTCGC TACTTATATTCGCTTTCGTGGGА.К.C AGTTACACATА.К.CCACCА.К.ACC ATTACCGCCTGCCCGATAGGC CCА.К.ACCACAGGА.К.ACTGTGC 40 8 Призначення: Детектування свинячого TTV1 Детектування свинячого TTV1 Детектування свинячого TTV1 Детектування свинячого TTV1 Детектування свинячого TTV1 Детектування свинячого TTV2 Детектування свинячого TTV2 UA 108359 C2 Продовження таблиці 1 TTV2 – nR (SEQ ID NO:52) TTV1 – IF (SEQ ID NO:1) TTV1-2340R (SEQ ID NO:2) TTV1-2311F (SEQ ID NO:3) TTV1 – IR (SEQ ID NO:4) TTV2 – IF (SEQ ID NO:5) TTV2-2316R (SEQ ID NO:6) TTV2-GCF (SEQ ID NO:7) TTV2-IR (SEQ ID NO:8) 5 10 15 20 25 30 35 CTTGACTCCGCTCTCAGGAG CATAGGGTGTА.К.CCА.К.TCAGATTTА.К.GGCGTT GGTCATCAGACGATCCATCTCCCTCAG CTTCTGAGGGAGATGGATCGTCTGATGA TTGAGCTCCCGACCА.К.TCAGА.К.TTGACT TTGTGCCGGAGCTCCTGAGAGC AGGTGCTTGAGGAGTCGTCGCTTG CTCА.К.GCACGAGCAGTGGATCCTCTCA TACCCAGGCGGTTAGACACTCAGCTCT Детектування свинячого TTV2 Клонування геному (фрагмент B) Клонування геному (фрагмент B) Клонування геному (фрагмент С) Клонування геному (фрагмент С) Клонування геному (фрагмент Е) Клонування геному (фрагмент Е) Клонування геному (фрагмент F) Клонування геному (фрагмент F) Несподівано виявилось, що у однієї і тієї ж свині існують два типи PTTV в геногрупі 1 і в геногрупі 2, що показало наявність послідовностей в кожному конструкті. Для диференціювання і збирання чотирьох штамів PTTV проводили порівняння послідовностей з трьома відомими штамами PTTV, Sd-TTV31, TTV-1p і TTV-2p (Фігури 1 B і 1 C). На Фігурі 1 В показані стадії диференціювання і збирання повних геномних послідовностей штамів PTTV1a-VA і PTTV1b – VA віруса PTTV1 з фрагментами ПЛР В і С, які після цього клонували. Ініціюючі кодони ORF1 і ORF2 у фрагменті В, а також термінуючі кодони ORF1 у фрагменті С відмічені знаками “^” або “*”. Відповідні послідовності двох відомих штамів PTTV1, Sd-TTV31 і TTV-1p, також показані. Консервативні послідовності заштриховані, а делеції нуклеотидів показані пунктиром. У випадку PTTV1 ініціюючий кодон ATG і термінуючий кодон TGA очікуваної ORF1 розташовані у фрагментах В і С, відповідно (Фігура 1 В). Розташування кодонів у двох групах PTTV1 було різним, у першого кодона - ідентичне Sd-TTV31, а у другого – ідентичне TTV-1p (Фігура 1 В). Крім того, ініціюючі кодони ORF2 у двох групах також розташовувались у різних місцях, що відповідало ORF1. Крім того, філогенетичні аналізи для чотирьох різних послідовностей ділянки В (дві за даними секвенування і дві в штамах Sd-TTV31 і TTV-1p) і чотирьох різних послідовностей на ділянці С підтвердили, що перша послідовність кластеризувалась з Sd-TTV31, а друга – кластеризувалась з TTV-1p (ці дані не представлені). Відповідно, ми змогли диференціювати та зібрати дві групи бібліотек послідовностей з обох фрагментів В і С в два геноми PTTV1 повної довжини, які були позначені як штами PTTV1a-VA (SEQ ID NO:9) і PTTV1b-VA (SEQ ID NO:10), відповідно (див. Фігуру 1 В). На Фігурі 1 С показані стадії диференціювання і збирання повних геномних послідовностей штамів PTTV2b-VA і PTTV2c-VA з фрагментами ПЛР E і F, які після цього клонувались. Показана відповідна послідовність штаму TTV-2p, а консервативні послідовності заштриховані. Пунктирними лініями позначені делеції в нуклеотидах. Відповідно показані унікальні нуклеотиди з ділянкою, що перекривається (пунктирні лінії) для кожного штаму ("неперервний" "AG “ нуклеотид ” в PTTV2b –VA (SEQ ID NO: 11) і два поодиноких нуклеотиди "A" і "G" в PTTV2c-VA (SEQ ID NO: 12)). Процес диференціювання двох штамів PTTV2 відбувався легше. Унікальний неперервний "AG" нуклеотид, розташований на ділянці, що перекривається, двох ПЛР – фрагментів був спільним для двох груп послідовностей з фрагментів Е і F, відповідно (Фігура 1 С). "Зібрана" повна геномна послідовність представляла штам PTTV2 і була позначена як PTTV2b – VA (SEQ ID NO: 11). Аналогічним чином на основі двох унікальних поодиноких нуклеотидів "A" і "G", спільних для ділянки, що перекривається, іншого набору послідовностей з фрагментів Е і F, відповідно, була зібрана повна геномна послідовність іншого штаму, позначеного як PTTV2c-VA (SEQ ID NO: 12) (див. Фігуру 1 С). Філогенетичний аналіз із застосуванням чотирьох послідовностей фрагментів Е і F разом з двома відповідними послідовностями з TTV-2p підтвердили цю картину (дані не представлені). 9 UA 108359 C2 5 10 15 20 Цей винахід передбачає отримання чотирьох виділених генотипів або підтипів свинячого віруса TTV, які асоціюються з вірусною інфекцією у свиней. Цей винахід включає, але без обмеження, генотипи або підтипи свинячого віруса TTV PTTV1a-VA, PTTV1b-VA, PTTV2b-VA і PTTV2c-VA, при цьому ці генотипи або підтипи віруса мають нуклеотидні послідовності, представлені в SEQ ID NO: 9 (PTTV1a-VA), SEQ ID NO: 10 (PTTV1b-VA), SEQ ID NO: 11 (PTTV2b-VA) і SEQ ID NO: 12 (PTTV2c-VA), їх функціональному еквіваленті або в комплементарній нитці. Варто мати на увазі, що конкретна нуклеотидна послідовність, отримана з будь-якого свинячого TTV, буде мати незначні відмінності, що природно існують між окремими вірусами. Ці відмінності в послідовностях можуть стосуватись делецій, заміщень, інерцій тощо. Геномна структура, запропонована для кожного з чотирьох штамів PTTV, була детально проаналізована і представлена в Таблиці 2 разом з даними для трьох відомих штамів PTTV, SdTTV31, TTV-1p і TTV-2p. Всі чотири американські штами PTTV мають подібний розмір геномів, що складає 2,878 п.о. (PTTV1a-VA SEQ ID NO: 9), 2,875 п.о. (PTTV1b-VA SEQ ID NO: 10), 2,750 п.о. (PTTV2b-VA SEQ ID NO: 11) і 2,803 п.о. (PTTV2c-VA SEQ ID NO: 12), відповідно. Обидва штами PTTV1a-VA (SEQ ID NO: 9) і Sd-TTV31 мали одну й ту ж довжину геному. Раніше описані послідовності в штамах TTV-1p і TTV-2p всі містять численні неохарактеризовані нуклеотиди на ділянці UTR, збагаченій GC. Після штучного заповнення цих нуклеотидів консенсусними послідовностями, що відповідають PTTV1 і PTTV2, було показано, що TTV-1p тісніше пов'язаний з PTTV1b-VA (SEQ ID NO: 10), в той час як TTV-2p тісніше пов'язаний з PTTV2b-VA (SEQ ID NO: 11) в геномі, відповідно (дані не представлені). Зібрані геномні послідовності генотипів і підтипів свинячого віруса TTV, PTTV1a-VA (SEQ ID NO: 9), PTTV1b-VA (SEQ ID NO: 10), PTTV2b-VA (SEQ ID NO: 11) і PTTV2c-VA (SEQ ID NO: 12) були зареєстровані в Genbank® (Nucleic Acids Research, 2008 Jan, 36 (Database issue): D25 – 30) під номерами доступу GU 456383, GU456384, GU456385 і GU456386, відповідно. 25 Таблиця 2 Порівняння організації геномів і ORF в семи штамах свинячого TTV Вірус Штамм Свинячий TTV вид 1 Тип 1a Тип 1b PTTV1a-VASd-TTV31 PTTV1b-VATTV-1p Країна США Повна довжина 2878 (нукл.) Номер доступу в GU456383 Genbank TATA бокс 288-291 Очікув. 5’-кінець 316 мРНК ORF1 Розмір (а.к.) 635 Екзон # 1 Ініціюючий 534 Термінуючий 2441 ORF2 Розмір (а.к.) 73 Екзон # 1 Ініціюючий 430 Термінуючий 651 ORF 1 / 1 Розмір (а.к.) 174 Екзон # 2 Термінуючий 534 647/648 Сплайсинг 2030/2031 Термінація 2441 ORF2 / 2 (ORF3) Японія 2878 США Бразилія 2875 Свинячий TTV вид 2 Підтип 2a Підтип 2b Підтип 2c PTTV2b- PTTV2cTTV-2p VA VA Бразилія США США Не закінч. Не закінч. 2750 2803 AB076001 GU456384 AY823990 AY823991 GU456385 GU456386 288-291 288-291 288-291 233-236 233-236 285-288 316 316 316 261 261 313 635 1 534 2441 639 1 517 2436 637 1 517 2430 624 1 476 2350 625 1 476 2353 625 1 528 2405 73 1 430 651 72 1 428 646 72 1 428 646 68 1 393 599 68 1 393 599 68 1 445 651 174 182 182 178 178 178 2 2 2 2 2 2 534 517 517 476 476 528 647/648 642/643 642/643 595/596 595/596 647/648 2030/2031 2013/2014 2007/2008 1933/1934 1936/1937 1988/1989 2441 2436 2430 2350 2353 2405 10 UA 108359 C2 Продовження таблиці 2 Розмір (а.к.) Екзон # Ініціюючий Сплайсинг 224 224 228 228 199 199 199 2 2 2 2 2 2 2 430 430 428 428 393 395 445 647/648 647/648 642/643 642/643 595/596 595/596 647/648 2030/2031 2030/2031 2013/2014 2007/2008 1933/1934 1936/1937 1988/1989 2487 2487 2485 2479 2330 2333 2385 Термінація Сигнал поліаденілювання 2458-2463 2458-2463 2462-2467 2456-2461 2473-2478 2476-2481 2528-2533 (А.К.TА.К.A) Цифри (крім розмірів повного генома, ORF та номерів екзонів) вказують на положення нуклеотидів (нукл.) в геномі відповідних штамів PTTV 5 10 15 20 25 30 35 40 45 В ході двох нещодавно проведених досліджень були ідентифіковані транскрибовані вірусні мРНК і експресія, принаймні, шести вірусних білків під час реплікації TTV людини (Mueller et al., 2008, supra; Qiu et al., 2005, supra), що більше, ніж передбачена кількість ORF, яка кодується TTV людини (Okamoto, H., Nishizawa, T., Tawara, A., Takahashi, M., Kishimoto, J., Sai, T., and Sugai, Y. (2000b). TT virus mRNAs detected in the bone marrow cells from an infected individual. Biochem. Biophys. Res. Commun. 279 (2), 700 – 7), тому ми включили інформацію про новий TTV людини для порівняння з послідовностями PTTV. 5" - кінці мРНК були картовані в нуклеотид "A", який розташований на 25 нуклеотидів нижче по ходу транскрипції ТАТА – боксу (Mueller et al., 2008, supra). Це вихідна точка, її сусідня послідовність (cgа.к.tggctgAgtttatgccgc (SEQ ID NO: 39); вихідна точка підкреслена) і відстань до розташованого проти напрямку транскрипції ТАТА боксу (24 нуклеотиди; Таблиця 2) є дуже консервативними у всіх семи штамів PTTV, що дає змогу припустити, що PTTV і TTV людини можуть використовувати спільний 5" - кінець мРНК для трансляції. Поблизу ТАТА – боксу були ідентифіковані п'ять додаткових повністю консервативних ділянок у всіх семи штамів PTTV. Дві ділянки завдовжки 11 нуклеотидів кожен (Agtcctcattt (SEQ ID NO: 40) і а.к.ccа.к.tcaga (SEQ ID NO:41)) розташовані вгору по течії ТАТА – боксу, тоді як три інші ділянки (ctgggcgggtgccggag з 17 нуклеотидів (SEQ ID NO: 42); cggagtcа.к.ggggc із 14 нуклеотидів (SEQ ID NO: 43); tatcgggcagg із 11 нуклеотидів (SEQ ID NO: 44)) розташовані між запропонованим 5’- кінцем мРНК та ініціюючим кодоном ORF2. Ці консервативні PTTV – консервативні послідовності можуть містити спільні елементи, які регулюють експресію вірусного гена. Раніше три ORF (ORF 1–3) були запропоновані в геном трьох відомих штамів PTTV, відповідно (Niel et al., 2005, supra; Okamoto et al., 2002, supra). Чотири штами PTTV з США, що є прототипами в цьому дослідженні, мають цю ж структуру. Відповідна ORF3 в TTV людини була перейменована на ORF2 / 2, оскільки вона ініціює той же кодон ATG в ORF2 і вона залишається в тій же ORF (продовжуючи її) після сплайсингу (Фігура 1 А) (Mueller et al., 2008, supra; Qiu et al., 2005, supra). Ми дотримуємось номенклатури TTV людини для перегляду класифікації PTTV в цьому дослідженні. ORF1 / 1 TTV людини являє собою вперше ідентифікований вірусний білок, який кодується двома екзонами в ORF1 (Qiu et al., 2005, supra). ORF1 / 1 і ORF1 мають спільну ідентичну N - і C - частини. Протилежна частина ORF1/1 в PTTV легко ідентифікується у всіх семи штамах PTTV. У TTV людини ORF1 і ORF2 кодуються вірусною мРНК з, приблизно, 2,8 т. н. (тисячі нуклеотидів), тоді як ORF1 / 1 і ORF1 / 2 кодуються розщепленою вірусною мРНК з приблизно 1,2 т. н. (Mueller et al., 2008, supra; Qiu et al., 2005, supra). Оскільки ці чотири ORF в геномах PTTV були розшифровані і оскільки послідовності і положення в очікуваному донорі сплайсингу і акцепторні сайти в семи штамах PTTV є дуже консервативними (див. Таблицю 2), то вважають, що свинячий TTV, очевидно, також кодує дві відповідні мРНК. Багато штамів TTV людини генетично подібні до CAV, що кодує білок TTV, який індукує апоптоз (TAIP), в якому протилежна частина CAV була названа апоптином (de Smit, M. H., and NOteborn, M. H. (2009). Apoptosis-inducing proteins in chicken anemia virus and TT virus. Curr Top Microbiol ImmuNOl 331, 131-49). ORF TAIP була вбудована в ORF2. Однак у свинячому TTV відповідний TAIP не існує. Проведене нещодавно дослідження показало, що експресія апоптину або TAIP потребує реплікації CAV в культивованих клітинах (Prasetyo, A. A., Kamahora, T., Kuroishi, A., Murakami, K., and HinoO, S. (2009). Реплікація віруса анемії курчат потребує 11 UA 108359 C2 5 10 15 20 25 30 35 40 наявності апоптину та доповнюється VP3 віруса людини торкве тено (TTV) (Virology 385 (1), 8592). Парне порівняння послідовностей (PASC) являє собою корисний метод, який дає змогу побудувати графік залежності частоти розподілу відсотка ідентичності парних послідовностей нуклеотидів від всіх доступних геномних послідовностей вірусів в тій же самій родині (Bao, Y., Kapustin, Y., and Tatusova, T. (2008). Virus Classification by Pairwise Sequence Comparison (PASC). In "Encyclopedia of Virology, 5 vols." (B. W. J. Mahy, and M. H. V. Van Regenmortel, Eds.), Vol. 5, pp. 342-8. Elsevier, Oxford). Різні піки, що генеруються програмою PASC, здебільшого відображають групи родів, видів, типів, підтипів і штамів (див. Фігуру 2). У цьому дослідженні ми провели PASC TTV з використанням 121 повної геномної послідовності штамів людини і тварин, які належать до TTV, доступних в базі даних GenBank (Фігура 2). З урахуванням припущення, що члени TTV класифікуються в окрему родину, Anelloviridae, два піки, що відповідають 36-55 % і 55-67 % ідентичності нуклеотидних послідовностей, представляють групи родів і видів, відповідно (Фігура 2). Відповідно, тип TTV визначається як група TTV, що має 67-85 % ідентичності нуклеотидних послідовностей, а підтип TTV визначається як група TTV, що має 8595 % ідентичності нуклеотидних послідовностей. Штами TTV, які мають понад 95 % ідентичності нуклеотидних послідовностей, можуть класифікуватись далі як варіанти (див. Фігуру 2). Подібна класифікація для послідовностей ізолятів 103 TTV була запропонована Jelcic et al (Jelcic, I., Hotz-Wagenblatt, A., Hunziker, A., Zur Hausen, H., and de Villiers, E. M. (2004). Isolation of multiple TT virus genotypes from spleen biopsy tissue from a Hodgkin's disease patient: genome reorganization and diversity in the hypervariable region. J Virol 78(14), 7498-507). Ці запропоновані критерії класифікації TTV були застосовані при проведенні філогенетичних аналізів геномних послідовностей чотирьох прототипів штамів із США та трьох інших відомих штамів РTTV. Парне порівняння повних нуклеотидних послідовностей цих штамів показало, що чотири штами РTTV1 мають 54,0-56,4 % ідентичності нуклеотидних послідовностей у порівнянні з трьома штамами РTTV2 (Таблиця 3). Відповідно, раніше відмічену в літературі "геногрупу" РTTV, ймовірно, варто назвати "видом", а РTTV1 і РTTV2 будуть видами 1 і 2 свинячого TTV, відповідно. Вид 1 РTTV складається з двох типів вірусів, позначених як тип 1а (включаючи TTV1p і PTTV1b-VA (SEQ ID NO: 10)), відповідно, оскільки ідентичність нуклеотидних послідовностей в цих двох типах вірусів складає 69,8-70,7 % (див. Таблицю 3). Sd-TTV31 і TTV1a-VA (SEQ ID NO: 9) визнаються варіантами штамів одного й того ж виду через високе значення відсотка ідентичності їхніх нуклеотидних послідовностей (95,1 %). Однак два штами типу 1 b, TTV-1p і PTTV1b-VA (SEQ ID NO: 10), можуть належати до двох різних підтипів (ідентичність нуклеотидних послідовностей складає 86,4 %). Для виду 2 РTTV три штами, ймовірно, варто класифікувати в окремі підтипи (TTV-2p для підтипу 2a, PTTV2b-VA SEQ ID NO: 11) для підтипу 2b і PTTV2c-VA (SEQ ID NO: 12) для підтипу 2c, відповідно, на основі величини відсотка ідентичності їхніх нуклеотидних послідовностей, що становить 86,5-90,9 %. Ця запропонована нова система класифікації PTTV чітко простежується на філогенетичному дереві (див. Фігуру 3). Філогенетичні дерева, побудовані на основі очікуваних амінокислотних послідовностей в ORF1, ORF1/1, ORF2 і ORF2/2 в PTTV, також узгоджувались з цією запропонованою класифікацією (див. Фігури 3 В-3Е). Таблиця 3 Парне порівняння послідовностей в повних геномах семи штамів свинячого TTV Вид 1 свинячого TTV Тип 1a PTTV1a-VA Sd-TTV31 Тип 1a PTTV1a-VA Sd-TTV31 Тип 1b PTTV1b-VA TTV-1p Підтип 2a TTV-2p Підтип 2b Тип 1b PTTV1b-VA TTV-1p 95.1 70.5 70.7 Вид 2 свинячого TTV Підтип Підтип 2b Підтип 2c 2a PTTV2b- PTTV2cTTV-2p VA VA 69.8 70.1 55.1 56.0 56.2 56.4 86.4 55.7 55.9 54.0 55.2 54.7 54.7 55.2 55.4 86.5 86.8 12 UA 108359 C2 Продовження таблиці 3 PTTV2b-VA Підтип 2c PTTV2c-VA 5 10 15 20 25 30 35 40 45 50 55 90.9 Ці дані були отримані із застосуванням програми PASC, а значення виявляють % ідентичності нуклеотидних послідовностей. Унікальні мутації, делеції та / або інсерції розпорошені по всіх геномах в різних видах, типах і підтипах PTTV. Наприклад, положення ініціюючих і термінуючих кодонів ORF1 та ініціюючих кодонів ORF2, показане на Фігурі 1 В, відрізняється в типах 1а і 1b PTTV. Два штами PTTV1 b також несуть делецію 2-кодона після ініціації ORF2, на відміну від PTTV1 а (див. Фігуру 1 В). Варто зазначити, що обидва TTV-2p і PTTV2b – VA характеризуються делецією 52 т.н., яка знаходиться на відстані 39 т.н. вгору по течії від першої консервативної послідовності завдовжки 11т.н (Agtcctcattt (SEQ ID NO: 40)) в UTR, у порівнянні з PTTV2c-VA. У зв'язку з цією делецією розмір геному PTTV2b – VA (ймовірно, й TTV-2p) був значно меншим, ніж розмір геному PTTV2с – VA (див. Таблицю 2). Була виділена із зразків сироватки низка клонів субвірусного TTV людини. Вони розглядаються як повні геноми TTV, оскільки ORF у більшості цих субвірусних молекул залишаються інтактними (de Villiers et al., 2009; Leppik et al., 2007). Вони мають змінну довжину в UTR і повністю або частково делеціюються. Стан TTV-2p і PTTV2b – VA нагадує стан субвірусних молекул TTV у людини, що означає, що підтипи PTTV2а і PTTV2b можуть бути субвірусними молекулами, отриманими з підтипу PTTV2с. Варто зазначити, що 3’-кінцева послідовність праймера TTV2-nF при вкладеній ПЛР (див. Таблицю 1), яка зазвичай застосовується для детектування PTTV2 в польових зразках іншими групами (Ellis et al., 2008, supra; Kekarainen et al., 2007, supra; Kekarainen et al., 2006, supra; Krakowka et al., 2008, supra), розташована з обох боків делеції. Відповідно, метод вкладеної ПЛР, який застосовується в наш час для детектування РTTV2, очевидно, не є достатнім для ідентифікації генетично відмінних різних штамів підтипу PTTV2с. Джерелом виділеного вірусного штаму є зразки сироватки, фекалій, слини, сперми і тканин свиней, інфікованих вірусом TTV. Однак, вважають, що метод із застосуванням рекомбінантної ДНК може бути використаний для реплікації та хімічного синтезу нуклеотидної послідовності. Відповідно, цей винахід передбачає виділений полінуклеотид, який містить, але без обмеження, нуклеотидну послідовність, представлену в SEQ ID NO: 9, SEQ ID NO: 10, SEQ ID NO: 11 і SEQ ID NO: 12, або її комплементарного ланцюга; полінуклеотид, який гібридизується і є комплементарним, щонайменше, на 67 % нуклеотидної послідовності, представленої в SEQ ID NO: 9, SEQ ID NO: 10, SEQ ID NO: 11 і SEQ ID NO: 12, переважно, комплементарний на 85 % або більш переважно, комплементарний на 95 %; або імуногенний фрагмент, обраний з групи, яка складається з амінокислотної послідовності білка ORF1, представленого в SEQ ID NO: 13 (PTTV1a-VA), SEQ ID NO: 14 (PTTV1b-VA), SEQ ID NO: 15 (PTTV2b-VA), SEQ ID NO: 16 (PTTV2c-VA), амінокислотної послідовності білка ORF2, представленого в SEQ ID NO: 17 (PTTV1a-VA), SEQ ID NO: 18 (PTTV1b-VA), SEQ ID NO: 19 (PTTV2b-VA), SEQ ID NO: 20 (PTTV2c-VA), амінокислотної послідовності білка ORF1/ 1, представленого в SEQ ID NO: 21 (PTTV1a-VA), SEQ ID NO: 22 (PTTV1b-VA), SEQ ID NO:23 (PTTV2b-VA), SEQ ID NO: 24 (PTTV2cVA), амінокислотної послідовності білка ORF2/2, представленого в SEQ ID NO: 25 (PTTV1a-VA), SEQ ID NO:26 (PTTV1b-VA), SEQ ID NO: 27 (PTTV2b-VA), SEQ ID NO: 28 (PTTV2c-VA). Імуногенні та антигенні кодуючі ділянки можуть бути визначені з використанням методів, що відомі з рівня техніки, після чого вони можуть бути застосовані для отримання моноклональних або поліклональних антитіл для скринінгу імунореактивності або для інших діагностичних цілей. Цей винахід передбачає також очищений імуногенний білок, закодований виділеними полінуклеотидами. Бажано, щоб білок був виділеним або рекомбінантним білком ORF1 або білком ORF2, який міститься в, щонайменше, одному зі згаданих вище виділених підтипів TTV, бажаніше, щоб такий білок був білком ORF1. Вважають, що ORF1 свинячого TTV кодує структурний білок, асоційований з реплікацією (Maggi, F., and Bendinelli, M. (2009). Immunobiology of the Torque teNO viruses and other anelloviruses. Curr Top Microbiol Immunol. 331, 65-90). ORF1 – кодуючі продукти семи штамів РTTV, мають довжину, яка становить 624 – 635 а. к. і містять велику кількість аргінінових залишків на своєму N-кінці, що, як вважають, мають ДНК–зв'язуючу активність (див. Фігуру 4). На Фігурі 4 консервативні послідовності заштриховані. Пунктирними лініями позначені амінокислотні делеції. Мотиви RCR (тип реплікації – реплікація за типом "кільця, що котиться") 13 UA 108359 C2 5 10 15 20 25 30 35 40 45 50 55 показані суцільними лініями. Три HVRs (гіперваріабельні області) (PTTV1-HVRs 1, 2 і 3) в штамах PTTV1 і дві HVRs (PTTV2-HVRs 1 і 2) в штамах PTTV2 показані пунктирними лініями. Межі з'єднання ORF1/1 показані стрілками. Передбачені мотиви реплікації за типом "кільця, що котиться" (RCR) (Ilyina, T. V., and Koonin, E. V. (1992). Conserved sequence motifs in the initiator proteins for rolling circle DNA replication encoded by diverse replicons from eubacteria, eucaryotes and archaebacteria. Nucleic Acids Res. 20(13), 3279-85), знаходяться в різних положеннях у різних типів і підтипів PTTV, які можуть бути тип– або підтип– специфічними. Мотив – III RCR є консервативним у штамах PTTV типу 1а (положення а. к. 14 – 17 в PTTV1a-VA SEQ ID NO: 13) і в штамах типу 1 b (положення а. к. 379-382 в PTTV1b-VA SEQ ID NO: 14), відповідно, тоді як той же консервативний мотив, ідентифікований у всіх трьох штамів PTTV2, розташований в положенні а. к. 482-485 PTTV2b-VA SEQ ID NO: 15 (Фігура 4). Обидва PTTV2b-VA SEQ ID NO: 15 і PTTV2c-VA SEQ ID NO: 16 також містять консервативний мотив - II RCR (HxQ) в положенні а. к. 331-333 в PTTV2b –VA, який відсутній у TTV-2p (див. Фігуру 4). Білки ORF в штамах PTTV видів 1 і 2 мають дуже низьку величину відсотка ідентичності послідовностей, який складає лише 22,4 – 25,8 %, що робить важкою ідентифікацію дуже консервативних амінокислотних послідовностей у двох видів (див. Фігуру 4). Відсоток ідентичності а. к. ORF1 в PTTV, вид 1, штами типу 1а і 1b, складає 50,3-52,7 %. У чотирьох штамів PTTV1 були ідентифіковані три основні гіперваріабельні області (HVR), PTTV1 – HVRs 13, з досить великою кількістю амінокислотних заміщень, тоді як у трьох штамах PTTV2 спостерігались дві HVRs (PTTV2 – HVRs 1 і 2) (див. Фігуру 4). Три штами PTTV2 мають приблизно 20 амінокислотних делецій у відповідній області PTTV1 – HVR1. Більш того, дві HVRs PTTV2 розташовані у відповідній області PTTV1 – HVR3 (див. Фігуру 4). Три HVRs розташовані лише в ORF1. Очевидно, вони відіграють роль в уникненні "імунологічного нагляду" за господарем і сприяють виникненню стійкої інфекції PTTV, про що свідчить вивчення TTV людини. Амінокислотні послідовності в ORF2 значно відрізняються в чотирьох штамів PTTV1 (PTTV1a-VA SEQ ID NO: 17; PTTV1b-VA SEQ ID NO: 18) і трьох штамів PTTV2 (PTTV2b-VA SEQ ID NO: 19; PTTV2c-VA SEQ ID NO: 20) (див. Фігуру 5). Однак вони містять загальний консервативний протеїнтирозинфосфатазоподібний (PTPase) мотив (Wx7Hx3CxCx5H) на N – кінці (див. Фігуру 4). Цей мотив також є консервативним у всіх штамів TTV, TTMV і TTMDV людини, а також CAV. Білок ORF2 TTMV або CAV також виявляв активність серин/треонін/фосфатази (S/T Ppase) (Peters, M. A., Jackson, D. C., Crabb, B. S., and Browning, G. F. (2002). Chicken anemia virus VP2 is a novel dual specificity protein phosphatase. J. Biol. Chem. 277(42), 39566-73). Як вважають, подвійна специфічність білка ORF2 регулює транскрипцію гена господаря, сигнальну трансдукцію та реакцію цитокінів під час реплікації вірусів. Нещодавно аналіз мутагенезу двох консервативних основних амінокислотних залишків перед останнім гістидиновим залишком мотиву в CAV показав, що ці два залишки виявляють вплив на реплікацію віруса, цитопатологію in vitro та аттенуацію in vivo (Peters, M. A., Crabb, B. S., Washington, E. A., and Browning, G. F. (2006). Site-directed mutagenesis of the VP2 gene of Chicken anemia virus affects virus replication, cytopathology and host-cell MHC class I expression. J. Gen. Virol. 87(Pt 4), 823-31; Peters, M. A., Crabb, B. S., Tivendale, K. A., and Browning, G. F. (2007). Attenuation of chicken anemia virus by site-directed mutagenesis of VP2. J. Gen. Virol. 88(Pt 8), 2168-75). Два основних амінокислотних залишки ("KK") є консервативними в трьох штамах PTTV2. Однак лише перший основний залишок ("R") зберігається в двох штамах PTTV2, тоді як обидва основні залишки в штамах PTTV1b заміщені (див. Фігуру 5). На Фігурі 5 пунктирні лінії показують амінокислотні делеції. П'ять консервативних амінокислот в загальному мотиві Wx7Hx3CxCx5H (підкреслений), ідентифіковані в TTV, TTMV і CAV, заштриховані. Положення двох амінокислотних залишків перед останнім гістидиновим залишком вказаного мотиву, що, як було показано, впливають на реплікацію, цитопатологію віруса та аттенуацію in vivo у CAV, позначені значком “^”. Узагальнюючи вищесказане, варто відзначити, що цей винахід дав змогу визначити у зразку сироватки одного кнура зі штату Віргинія повні геномні послідовності чотирьох штамів свинячого TTV, які становлять різні генотипи або підтипи цього віруса. Дані, отримані в результаті даного дослідження, яскраво свідчать, що, як і у випадку TTV людини, існують численні види інфекції, викликані різними генотипами і підтипами РTTV, які, очевидно, є спільними для свиней. Ми також отримали нову інформацію стосовно організації геномів, ступеня варіабельності та характеристик консервативних нуклеотидних та амінокислотних мотивів РTTV, яка дасть змогу вдосконалити існуючий в даний час метод детектування із застосуванням ПЛР, допоможе отримати реагенти для серологічної діагностики та ініціювати структурно-функціональне 14 UA 108359 C2 5 10 15 20 25 30 35 40 45 50 55 вивчення РTTV. Запропонована також нова класифікація РTTV, основана на філогенетичних і генетичних аналізах геномних послідовностей семи відомих штамів РTTV. Цей винахід передбачає також способи діагностики інфекції, викликаної свинячим TTV, шляхом детектування вірусної ДНК у зразках, відібраних у свиней або у інших ссавців, інфікованих свинячим TTV. Один переважний варіант даного винаходу належить до способів детектування послідовностей нуклеїнових кислот у свинячому TTV у свиней та інших видів ссавців за допомогою олігонуклеотидних праймерів для полімеразної ланцюгової реакції (ПЛР) з метою подальшої діагностики вірусної інфекції або захворювання. Діагностичні тести, які придатні для виявлення наявності або відсутності послідовності вірусної нуклеїнової кислоти в свинячому TTV у свиней або інших видів ссавців, включають виділення вірусної ДНК із зразків, відібраних у свиней, інфікованих свинячим TTV, або у свиней, у яких підозрюють наявність такої інфекції, і проведення кількісної ПЛР в режимі реального часу за присутності барвника SYBR Green і РTTV1-специфічного (SEQ ID NO: 29/ SEQ ID NO: 30) або PTTV2-специфічного (SEQ ID NO: 31/ SEQ ID NO: 32) праймерів. Відповідно до іншого варіанту даного винаходу метод діагностики можна адаптувати для одночасного детектування РTTV1 і PTTV2 шляхом застосування методу дуплексної ПЛР в режимі реального часу. Більш конкретно, метод включає виділення вірусної ДНК із зразків, відібраних у свиней, інфікованих свинячим TTV, або у свиней, у яких підозрюють наявність такої інфекції, проведення ПЛР в режимі реального часу за допомогою обох РTTV1-специфічного (SEQ ID NO: 29/ SEQ ID NO: 30) або PTTV2-специфічного (SEQ ID NO: 31/ SEQ ID NO: 32) праймерів при проведенні тієї ж реакції ПЛР в режимі реального часу. Оскільки величина Тm у РTTV1 і PTTV2 може бути визначена методом МСА, одночасно може бути визначена наявність ДНК РTTV1 і PTTV2. Згідно ще з одним варіантом даного винаходу спосіб діагностики може використовувати метод дуплексної вкладеної ПЛР. Спосіб включає виділення вірусної ДНК зі зразків, відібраних у свиней, інфікованих свинячим TTV, або у свиней, у яких підозрюють наявність такої інфекції, здійснення першого раунду ПЛР за допомогою однієї пари праймерів P1ab-mF (SEQ ID NO:33)/P1ab-mR (SEQ ID NO:34) і проведення другого раунду ПЛР за допомогою суміші двох пар праймерів, P1a-nF (SEQ ID NO:35)/P1a-nR (SEQ ID NO:36) для детектування PTTV1a і P1bnF (SEQ ID NO: 37)/P1b-nR (SEQ ID NO: 38) для детектування PTTV1b та візуалізації продуктів ПЛР. Вказані вище способи діагностики можуть бути оптимізовані фахівцем у цій галузі з урахуванням методів, загальновідомих з рівня техніки. Відповідно, один з варіантів даного винаходу належить до двох нових одноплексних способів аналізу шляхом проведення ПЛР за присутності барвника SYBR Green в режимі реального часу для кількісного визначення вірусних навантажень двох видів свинячого TTV, відповідно. РTTV1 – і РTTV2 – специфічні праймери були отримані для націлювання (таргетингу) на дуже консервативні області в шести геномах РTTV1 і чотирьох геномах РTTV2, відповідно, доступних в наш час. Інший варіант даного винаходу об'єднує два одноплексні способи аналізу в спосіб дуплексної ПЛР в режимі реального часу з подальшим МСА вірусних ампліконів, які можуть бути ідентифіковані за різними температурами плавлення, з метою одночасного детектування двох видів свинячого TTV, РTTV1а і РTTV1b. Згідно з третім варіантом, метод аналізу із застосуванням дуплексної вкладеної ПЛР для одночасної ампліфікації вірусних ДНК з двох типів РTTV1 у першому раунді ПЛР і диференційного детектування типів 1а і 1b у другому раунді ПЛР був розроблений для ідентифікації двох типів видів свинячого TTV, РTTV1а і РTTV1b, в одному зразку. Ці аналізи є простими та практичними методами діагностики видоспецифічних і типоспецифічних свинячих TTV. Послідовності потенційних праймерів були ідентифіковані шляхом вирівнювання сукупності послідовностей 10 повних геномів доступного свинячого TTV. РTTV1- специфічні праймери TTV1F (SEQ ID NO: 29) і TTV1R (SEQ ID NO: 30) були сконструйовані на основі двох консервативних областей геному, розташованих безпосередньо перед очікуваною ORF2 в шести геномах РTTV1, тоді як РTTV2- специфічні праймери TTV2F4 (SEQ ID NO: 31) і TTV2R4 (SEQ ID NO: 32) були сконструйовані на основі двох консервативних областей геному, розташованих одразу ж за очікуваною ORF2/2 в чотирьох геномах PTTV2 (див. Таблицю 4). Праймери не виявили тендеції до самодимеризації та крос-димеризації. Очікувані розміри ампліконов складали 118 п. о. фрагменту з праймерів РTTV1, що відповідають геному РTTV1b – VA і 200 п. о. фрагменту з праймерів РTTV2, що відповідають геному РTTV2с – VA, відповідно. 15 UA 108359 C2 Таблиця 4 Олігонуклеотидні праймери, використані для детектування методом ПЛР в режимі реального часу та методом дуплексної вкладеної ПЛР свинячих TTV Ідент. праймер TTV1F SEQ ID NO:29 TTV1R SEQ ID NO:30 TTV2F4 SEQ ID NO:31 TTV2R4 SEQ ID NO:32 P1ab-mF SEQ ID NO:33 P1ab-mR SEQ ID NO:34 P1a-nF SEQ ID NO:35 P1a-nR SEQ ID NO:36 P1b-nF SEQ ID NO:37 P1b-nR SEQ ID NO:38 5 10 15 20 25 30 Послідовність (від 5" до 3") TCCGА.К.TGGCTGAGTTTATGC TCCGCTCAGCTGCTCCT GGTGGTА.К.AGAGGATGА.К. А.К.TAGATTGGACACAGGAG Призначення PTTV1- специфіч. ПЛР в режимі реального часу PTTV1- специфіч. ПЛР в режимі реального часу PTTV2- специфіч. ПЛР в режимі реального часу PTTV2- специфіч. ПЛР в режимі реального часу TATCGGGCAGGAGCAGCT Дуплексна вкладена ПЛР TAGGGGCGCGCTCTACGT Дуплексна вкладена ПЛР CCTACATGА.К.GGAGА.К.AGACT Дуплексна вкладена ПЛР CCAGCGTCTCCAGGGTC Дуплексна вкладена ПЛР А.К.GCTACCА.К.GGGCTGG Дуплексна вкладена ПЛР GCGGTC(T/G)GTAGCGGTAGT Дуплексна вкладена ПЛР Відповідно до одного конкретного варіанту даного винаходу метод ПЛР в режимі реального часу із застосуванням барвника SYBR Green, що передбачає використання РTTV1- і РTTV2специфічних праймерів, може бути застосований спеціально для детектування ДНК свинячого TTV1 і TTV2, відповідно. У випадку РTTV1 була побудована стандартна крива в інтервалі 1 8 концентрацій цільової ДНК в 25 мкл. Для інтервалу, що становить 4,4 × 10 – 4,4 × 10 копій, була показана лінійна залежність. Межа мінімального детектування (44 копії) відповідала пороговому циклу реакції (Сt), що дорівнює 33,57. У випадку РTTV2 була також побудована 0 стандартна крива і використана для детектування концентрації ДНК в межах від 8,6 × 10 до 8,6 8 × 10 копій в 25 мкл реакційного середовища. Відповідний С t мінімальної межі детектування (8,6 копій) становив 36,53. Згідно з іншим конкретним варіантом даного винаходу метод дуплексної вкладеної ПЛР із застосуванням SYBR Green використовується для одночасного детектування ДНК свинячих TTV1 і TTV2. Різниця величин Т m у РTTV1 (87,0 ºС) і РTTV2 (80,0º С), яка складає 7 º, дає можливість відрізнити їх один від другого за допомогою МСА. Відповідно, два методи одноплексного аналізу можуть бути об'єднані в один метод дуплексної ПЛР в режимі реального часу для одночасного детектування РTTV1 і РTTV2. Позитивний зразок виявився зразком, який мав симетричний пік плавлення серед Т m для цього продукта. Цей новий метод аналізу спочатку був підтверджений з використанням 10 – кратного розведення суміші стандартів РTTV1 і РTTV2. Нематричний негативний контроль, з використанням стерильної води як матриці, показав протікання неспецифічної ампліфікації, викликаної крос-димеризацією між праймерами РTTV1 і РTTV2, яка не спостерігається при проведенні одноплексних аналізів (див. Фігуру 7 А). Це призвело до чіткого піку плавлення між 72,0 º С і 76,0 º С. На Фігурі 7 А показані піки температур плавлення стандарту РTTV1 (червоний колір; Т m=87,0 º С), стандарту РTTV2 (зелений колір; Тm=80,0 º С) і нематричного негативного контролю (обумовленого крос-димеризацією праймерів; чорний колір). На Фігурах 7В-7Е показані пікові температури плавлення репрезентативних зразків сироватки з різними вірусними навантаженнями РTTV1 і РTTV2. На Фігурі 7В представлений зразок № 5 сироватки кнура: відносно високі навантаження РTTV1 і РTTV2, при РTTV2 > РTTV1; Фігура 7С представляє зразок № 12 сироватки кнура: відносно високі навантаження РTTV1 і РTTV2, при РTTV1 > РTTV2; Фігура 7D показує зразок № 14 сироватки кнура: низькі навантаження РTTV1 і РTTV2; Фігура 7Е показує зразок № 10 сироватки кнура: РTTV1 позитивний, але РTTV2 негативний. Вірусні навантаження (одиниця: геномні копії / 16 UA 108359 C2 5 10 15 20 25 30 35 40 45 50 55 60 мл) РTTV1 і РTTV2 в кожному зразку, які були визначені методом одноплексної ПЛР в режимі реального часу, зазначені над відповідним піком температури плавлення. В одному прикладі, коли для аналізу 20 зразків сироватки, відібраних у дорослих кнурів, застосовували метод дуплексного аналізу в режимі реального часу, зразки з досить високими вірусними навантаженнями обох РTTV1 і РTTV2 характеризувались двома різними кривими плавлення, які відповідають РTTV1 і РTTV2 без неспецифічного піку температури плавлення (Фігури 7В-7С), тоді як зразки з низьким вірусним навантаженням, РTTV1 або РTTV2, мали вірусоспецифічні, а також неспецифічні криві плавлення (див. Фігури 7 D-7E). Не зважаючи на те, що два піки температури плавлення для зразка № 14 були низькими, вони розглядались як позитивні, оскільки вони характеризувались візуально відмінним і симетричним зростанням та падінням відповідній Тm РTTV1 і РTTV2 (див. Фігуру 7 D). На противагу цьому, зразок № 10 розглядали як позитивний лише стосовно РTTV1, оскільки симетричний пік температури плавлення РTTV2 був відсутній (див. Фігуру 7Е). Ці результати узгоджувались з результатами двох одноплексних аналізів (див. Таблицю 5). Більше того, розмір і форма піків плавлення якісно відображали відповідне вірусне навантаження у зразку, який піддавали детекції. Згідно з ще одним аспектом даного винаходу спосіб дуплексної вкладеної ПЛР застосовується для диференційного детектування двох типів свинячого TTV, РTTV1 і РTTV2. Винахідник даного винаходу продемонстрував існування двох різних генотипів свинячого TTV виду 1, позначених як РTTV1а і РTTV1b. Для того щоб з'ясувати, чи є коінфекція, викликана РTTV1а та РTTV1b, розповсюдженим захворюванням у свиней, був розроблений новий спосіб дуплексної вкладеної ПЛР для швидкого визначення відмінностей між цими двома генотипами. Вирівнювання геномних послідовностей ДНК свинячого TTV дало змогу ідентифікувати консервативну область геному, розташовану в N – кінцевій частині здогадної ORF1, що кодує вірусний капсидний білок (див. Фігуру 8). Ця область містить також повну ORF2 і неповну UTR "вгору по течії". Були сконструйовані праймери P1ab-mF (SEQ ID NO: 33)/P1ab-mR (SEQ ID NO: 34) для одночасної ампліфікації ДНК і РTTV1а, і РTTV1b в першому раунді ПЛР. Для диференційної ампліфікації кожного генотипу під час другого раунду ПЛР була використана суміш РTTV1а-специфічних праймерів P1a-nF (SEQ ID NO: 35)/P1a-nR (SEQ ID NO: 36) і PTTV1b-специфічних праймерів P1b-nF (SEQ ID NO: 37)/P1b-nR (SEQ ID NO: 38). Кінцеві продукти ПЛР для РTTV1а і РTTV1b мали розмір 162 п. о. і 96 п. о., відповідно, їх можна дуже легко відрізнити один від одного методом гелевого електрофорезу на 1 %-ному агарозному гелі, забарвленому етидію бромідом. Вважають, що цей метод аналізу не дасть змоги детектувати ДНК РTTV2 через специфічність праймерів (див. Фігуру 8). На Фігурі 8 консервативні послідовності показані точками і заштриховані. Пунктирними лініями показані нуклеотидні делеції. Розташування і напрямки трьох пар праймерів, застосованих для дуплексної вкладеної ПЛР, були відмічені стрілками. В одному прикладі 20 зразків сироватки, відібраних у дорослих кнурів, аналізували методом дуплексної вкладеної ПЛР і виявились позитивними для РTTV1а і РTTV1b, про що свідчила візуалізація двох смуг очікуваного розміру, з наступним підтвердженням продуктів ПЛР секвенуванням (дані не показані). В 19 зразках сперми не були ампліфіковані продукти ПЛР, що узгоджувалось з результатами звичайної вкладеної ПЛР для РTTV1 і ПЛР в режимі реального часу, описаними вище. У ході ретроспективного дослідження (Segales et al., 2009, supra) було встановлено, що інфекція, викликана двома видами свинячого TTV, була виявлена у свиней в 1985 р. на іспанських свинячих фермах. Разом з тим, питання про те, чи пов'язані свинячі TTV з будьякими захворюваннями у свиней, залишається невирішеним. Оскільки обидва види свинячого TTV широко розповсюджені у домашніх свиней, ймовірно, визначення вірусних навантажень TTV є важливішим, ніж з'ясування наявності ДНК TTV. Було показано, що рівень вірусного навантаження в зразках сироватки і сперми є важливим маркером для PCVAD при інфекції PCV2 (Opriessnig et al., 2007, supra). Відповідно, наявність методів кількісного РTTV – специфічного аналізу із застосуванням ПЛР в режимі реального часу допоможе ідентифікувати умови розвитку потенційного захворювання, пов'язаного зі свинячими TTV. Нещодавно були описані два способи аналізу із застосуванням ПЛР в режимі реального часу, основані на TaqMan-зондах. Спосіб одноплексного аналізу, розроблений канадською групою, не був видоспецифічним і був розроблений лише для кількісного визначення загальних вірусних навантажень двох видів РTTV (Brassard et al., 2009, supra). Спосіб дуплексного аналізу, запропонований німецькою групою, дав змогу провести специфічне та одночасне детектуванняобох видів (Gallei et al., 2009, supra). Таргетовані послідовності праймерів, використаних при проведенні цих аналізів, були визначені шляхом вирівнювання трьох геномних послідовностей свинячого TTV (Sd-TTV31, TTV-1p і TTV-2p) і були розташовані в UTR. При проведенні цього 17 UA 108359 C2 5 10 15 20 25 30 35 40 45 50 55 60 дослідження за допомогою семи додаткових доступних повних геномних послідовностей РTTV (чотири послідовності РTTV1 і три послідовності РTTV2) ми проаналізували та знову визначили консервативні області в 10 повних геномах РTTV. На основі скоригованого результату такого вирівнювання, отриманого в результаті зазначеного дослідження, були розроблені два видоспецифічні методи одноплексної ПЛР в режимі реального часу за умови присутності барвника SYBR Green для кількісного визначення вірусних навантажень РTTV1 і РTTV2, відповідно. Праймери, використані в наших методах, які можуть підвищити точність кількісного визначення, були сконструйовані для зв'язування з консервативними геномними областями, що відрізняються від областей у відомих методах. Наші аналізи показали суттєву специфічність та чутливість детектування 44 геномних копій для РTTV1 і 8,8 геномних копій / 25 мкл середовища для РTTV2, тоді як було повідомлено, що межа детектування при проведенні дуплексної ПЛР в режимі реального часу за допомогою TaqMan-зонду становить 10 геномних копій (Gallei et al., 2009, supra). Крім того, ПЛР в режимі реального часу з барвником SYBR Green є гнучким і недорогим методом, який можна проводити безпосередньо без необхідності застосування флуоресцентних міток. Насамкінець, з урахуванням високого ступеня генетичного різноманіття свинячих TTV, результати аналізів за допомогою SYBR Green навряд чи зазнають впливу різних відомих варіантів свинячого TTV, які, очевидно, несуть мутації в послідовностях, що зв'язують зонд, при проведенні аналізів за допомогою TaqMan-зонду. Не зважаючи на наявність ДНК TTV, всі зразки сироватки, відібрані у здорових свиней, випробувані у цьому дослідженні, містили низьку кількість РTTV1 і РTTV2, яка становила менше 6 2 × 10 копій/мл. Крім того, в трьох зразках сперми був виявлений надзвичайно низький титр ДНК РTTV2. Більша частина випробуваних зразків сироватки також була позитивною щодо ДНК PCV2, як було визначено методом звичайної вкладеної ПЛР (дані не представлені). У багатьох PCV2-позитивних свиней з низьким вірусним навантаженням, клінічна картина PCVАD (цирковірусна інфекція) не розвивалась. Запропонований поріг для розвитку PCVАD становить 7 10 або більше геномних копій PCV2/ мл сироватки (Opriessnig et al., 2007, supra). Крім того, ДНК-позитивність сперміїв стосовно PCV2 також є винятковим маркером статусу захворювання (Opriessnig et al., 2007, supra; Pal, N., Huang, Y.W., Madson, D.M., Kuster, C., Meng, X.J., Halbur, P.G. and Opriessnig, T., 2008. Development and validation of a duplex real-time PCR assay for the simultaneous detection and quantification of porcine circovirus type 2 and an internal control on porcine semen samples. J. Virol. Methods 149, 217-25). Положення з видоспецифічними РTTV може бути аналогічним ситуації з PCV2, тому індукція захворювань свиней може потребувати 7 високого титру РTTV, що складає понад 10 копій/мл. Видоспецифічні методи ПЛР в режимі реального часу, розроблені у цьому дослідженні, є простими та практичними засобами для подальших досліджень зв'язку РTTV із захворюваннями з використанням великої кількості клінічних зразків у випадку різних захворювань. Крім того, шляхом поєднання двох видів видоспецифічного одноплексного аналізу ми розробити та обґрунтували швидкий, рентабельний і надійний метод скринінгу для одночасного детектування та диференціації двох видів свинячого TTV, РTTV1 і РTTV2, що передбачає застосування дуплексної ПЛР в режимі реального часу, основаного на MCA. Не зважаючи на те, що цей метод не призначений для точного кількісного визначення обох видів РTTV, він становить собою більш зручний підхід, здатний замінити звичайну "вкладену ПЛР" при детектуванні. У порівнянні з ПЛР в режимі реального часу, звичайний метод вкладеної ПЛР для детектування свинячих TTV є тривалим (потребує проведення чотирьох раундів ПЛР), трудомістким та пов'язаним із забрудненням зображень, що відбувається під час численних раундів ПЛР. Завдяки різниці величин Т m РTTV1 і РTTV2 метод МСА, який проводиться після ампліфікації із застосуванням дуплексної ПЛР, дає змогу забезпечити очевидну специфічність реакції. Іншою перевагою цього дуплексного методу в режимі реального часу є те, що включення стандартів РTTV1 і РTTV2 є несуттєвим при проведенні відповідного протоколу, що робить цей метод найбільш прийнятним для застосування у будь-яких діагностичних лабораторіях, устаткованих автоматичними приладами для проведення ПЛР в режимі реального часу. Були описані численні випадки інфекції, викликаної свинячими TTV з різними генотипами або підтипами одного й того ж виду (Gallei et al., 2009, supra). Зокрема, наше попереднє дослідження показало, що вид 1 свинячого TTV складається з двох різних типів, РTTV1а (включаючи штами Sd-TTV31 і PTTV1a-VA) і PTTV1b (включаючи штами TTV-1p і PTTV1b-VA). Два нещодавно описані ізоляти PTTV1 з повними геномами, swSTHY-TT27 (GQ120664) із Канади та TTV1 #471819 (GU188045) - з Німеччини, на основі філогенетичного аналізу (дані не представлені) класифікувались як представники типу 1b. Дуплексна вкладена ПЛР, описана нами, підтвердила, що подвійна інфекція, спричинена двома генотипами PTTV1, зустрічалась у 18 UA 108359 C2 5 10 15 20 25 30 35 40 45 50 55 60 свиней часто. Цей новий метод аналізу досі є першим діагностичним підходом із застосуванням ПЛР при визначенні відмінностей між інфекціями, викликаними PTTV1а та PTTV1b. Оскільки в наш час невідомо, один чи обидва види інфекції, викликаної PTTV1а і PTTV1b, є релевантним чинником, що асоційований із захворюваннями, наш диференційний метод ПЛР повинен мати велике значення у майбутньому для з'ясування зв'язку між цими двома типами PTTV. Відповідно ще до одного аспекту даного винаходу, білки ORF свинячого TTV були експресовані та використані при проведенні імунологічного аналізу для детектування наявності специфічних антитіл до свинячого TTV. Згідно з одним із варіантів даного винаходу, три вкорочені та мічені гістидином білки ORF1 PTTV1а, PTTV1b і PTTV2 були експресовані та очищені в Escherichia coli (E. coli), відповідно. По тому, були розроблені та обґрунтовані два методи аналізу сироватки, Вестерн-блоттинг і ELISA, основані на цих рекомбінантних антигенах з використанням зразків свинячої сироватки, отриманих із різних джерел. Зокрема, серологічне визначення за допомогою PTTV1а-, PTTV1b- і PTTV2-специфічного методу ELISA забезпечує точний і простий інструмент для виявлення зв'язку інфекції, викликаної свинячим TTV, із захворюваннями. Відповідно до подальшого аспекту даного винаходу білки ORF свинячого TTV були експресовані і очищені як рекомбінантні капсидні білки ORF1 в системі E. coli (див. Фігуру 10, Фігуру 15). Ці вкорочені та мічені гістидином капсидні білки ORF1 PTTV1а, PTTV1b і PTTV2, були експресовані і очищені в Escherichia coli (E. coli), відповідно, та слугували як рекомбінантні капсидні субодиничні вакцини від інфекції, викликаної PTTV. Чотири штами TTV2, TTV-2p, TTV2#472142, PTTV2b-VA і PTTV2c-VA, містили доступні на даний час повні геномні послідовності. Не зважаючи на те, що з філогенетичної точки зору вони класифікуються в трьох очікуваних підтипах, порівняльний аналіз профілів гідрофільності ORF1, яка кодує амінокислоти з чотирьох PTTV2, показав, що вони містили три гідрофільні області: збагачену аргініном область від а. к. 1-49 на N - кінці і дві особливі області (I і II), розташовані в середині і в з- кінцевій частині, відповідно (Фігура 9 А). С – кінцева область, використана для експресії вкороченої ORF1 PTTV2c-VA та відповідних областей, які містяться в трьох інших штамах PTTV2, показані пунктиром. Вирівнювання амінокислотних послідовностей показало високі рівні консервативності послідовностей областей I (а. к. 322-349) і II (а.к. 536-625) в чотирьох штамах PTTV2 (Фігура 9 В). Оскільки вважають, що гідрофільні області є важливими для наявності антигенності багатьох білків, С – кінцева область (а. к. 310 – 625) в ORF1 PTTV2c-VA, SEQ ID NO:16, яка містить два домени, була обрана для експресії білків і використовувалась як антиген для детектування PTTV2 – специфічного антитіла у свинячій сироватці. Згідно з одним аспектом даного винаходу, експресія вкороченої ORF1 PTTV2c була достатньою для детектування всіх підтипів PTTV2 (2а, 2b і 2с; див. також Фігуру 3А). Згідно з одним з варіантів даного винаходу була отримана та експресована в E. coli С– кінцева область гена ORF1 PTTV2c, злита з 8 х His- мітками. Рекомбінантний білок був нерозчинним та експресувався в тільцях включення бактерій. На Фігурі 10 А показані результати SDS – PAGE неочищених продуктів 2с-ORF1. На Фігурі 10 В представлені результати SDS – PAGE очищених продуктів 2с-ORF1. На Фігурі 10 С представлені результати Вестерн-блоттингу очищених продуктів 2с- ORF1 з використанням анти - His- міченого mAb. Стрілки з вістрям білого кольору показують білок ORF1 очікуваного розміру та його "вкорочений" продукт, тоді як стрілки з чорним вістрям показують здогадні димери очікуваних і вкорочених білків. М: білкові маркери. На Фігурі 10 А показано два важливих поліпептиди (стрілки з вістрям білого кольору), отримані в неочищеному зразку 2с- ORF1, у порівнянні з контрольним образом. Смуга близько 40 кДа узгоджується з очікуваним розміром 2с- ORF1, тоді як поліпептид з м. в. близько 30 кДа був, очевидно, вкороченим на N-кінці продуктом, отриманим з першого продукту. Після очищення на колонці з нікелю SDS – PAGE показав наявність чотирьох поліпептидів, включаючи дві описані смуги (див. Фігуру 10 В). Їх також детектували методом Вестерн блоттингу, використовуючи mAb проти His мітки (див. Фігуру 10 С). Дві смуги, пов'язані з високою молекулярною вагою (стрілки з чорним вістрям), позначають гомодимери, утворені двома поліпептидами з м. в. близько 40 кДа і близько 30 кДа, відповідно, основані на передбачених розмірах (близько 80 кДа і близько 60 кДа). Результати показали, що очищена С кінцева PTTV2c-ORF1 була успішно отримана та могла бути використана для детектування антитіла свинячого TTV2 у свинячий сироватці. Відповідно до іншого варіанту даного винаходу антитіла до свинячого TTV2 в різних зразках сироватки можуть бути виявлені методом Вестерн-блоттингу з використанням очищеної Скінцевої PTTV2c-ORF1. Стрілки з білим вістрям позначають білок ORF1 з очікуваним розміром та його процесований продукт. Варто зазначити, що лише смуги зеленого кольору були визнані 19 UA 108359 C2 5 10 15 20 25 30 35 40 45 50 55 позитивними. У звичайних свиней (здорових та хворих), свиней породи CD/CD і гнотобіотичних свиней з різних джерел були відібрані понад 200 зразків сироватки. Після цього довільно відбирали зразки для детектування імуноглобулінових антитіл до PTTV2c-ORF1 з використанням очищеної С-кінцевої PTTV2c-ORF1 в якості антигена. На Фігурі 11 А представлені результати Вестерн-блоттингу відібраних зразків свинячої сироватки звичайних свиней, на Фігурі 11 В – для свиней CD/CD, а на Фігурі 11 С – для гнотобіотичних свиней. Очищені продукти PTTV2c-ORF1 застосовували як антигени. В більшості зразків звичайних свиней (Фігура 11 А) і свиней CD/CD (Фігура 11 В) були детектовані дві помітні смуги, близько 40 кДа і близько 30 кДа, що вказувало на наявність у цих свиней сильної інфекції, викликаної PTTV2. Однак у всіх гнотобіотичних свиней з двох різних джерел (Blacksburg, VA і Ames, IA) антитіла до PTTV2 виявлені не були (Фігура 11 С). Спостерігались також додаткові смуги з низькою молекулярною вагою. Вони, очевидно, обумовлені неспецифічною реактивністю при проведенні Вестерн-блоттингу. Відповідно до ще одного варіанту даного винаходу PTTV2-специфічний аналіз ELISA може бути використаний як серологічний тест для свинячого TTV2. Для декількох зразків, відібраних у звичайних свиней на фермі в штаті Вісконсін були представлені серонегативні результати (див. Фігуру 12). Ці негативні результати збирали та застосовували як негативний стандарт у развитку PTTV2-специфічного аналізу ELISA. Решта зразків з цього джерела були позитивними (Фігура 12, чотири лінії ліворуч). Крім того, свиняча сироватка з комерційної компанії, що використовується в клітинній культурі (припускається, що у свиней захворювання, зареєстровані в OIE, відсутні), також мала сильну анти-PTTV2 – ORF2 позитивність (Фігура 12) і застосовувалась в якості позитивного контролю для ELISA. Концентрації очищеного антигену 2с- ORF1, свинячої сироватки і кон'югату IgG визначали шляхом "шахматного" титрування для визначення низького сигналу фону та отримання найбільшої різниці OD 405 між позитивним та негативним контрольними зразками. Оптимальна кількість антигену становила 69 нг на комірку, а оптимальні результати ELISA були отримані при використанні розведення 1: 100 зразків сироватки та розведенні 1: 4000 кон'югатів IgG. Критичні величини ELISA знаходились в межах від 0,25 до 0,5 у кожному досліді. На Фігурі 4 представлений репрезентативний результат, що відображає узгодженість результатів Вестерн-блоттингу сироватки та розробленого методу ELISA. 138 зразків сироватки конвенційних свіней із 3 стад були обрані для аналізу кореляції зв`язку вірусного навантаження PTTV2 при проведенні ПЛР в режимі реального часу та рівня IgG антитіл до анти-PTTV2 у сироватці методом ELISA. Результати показали, що свині з 8 невизначеним або вищим вірусним навантаженням PTTV2 (10 копій/мл), більш ймовірно, мають нижчий рівень титру антитіла до PTTV2 у сироватці, ніж свині з середнім значенням вірусного навантаження PTTV2 (Фіг. 13). Зокрема, сироватки від 10 свиней в одному й тому ж стаді були також проаналізовані шляхом порівняння вірусних навантажень PTTV2 та рівня антитіл до анти-PTTV2 у сироватці після їх прибуття в нове господарство після два місяці після прибуття. У 9 з-поміж 10 свиней через 2 міс. були виявлені знижені вірусні навантаження (у трьох тварин вірус взагалі не був виявлений), тоді як титри антитіла до PTTV2 були підвищеними у 9 з-поміж 10 свиней (Фігури 14 А – 14 В). Ці результати дають підставу висловити припущення, що у 10 свиней була присутня інфекція на ранній стадії, викликана PTTV2, яка обумовлювала гуморальну відповідь та прогресивно продукувала IgG антитіла до капсидного білка ORF1. IgG антитіло до PTTV2-ORF1 було здатне нейтралізувати й навіть очищати вірус, що свідчить про те, що ORF1 дійсно кодує капсидний вірусний білок та може містити нейтралізуючі епітопи PTTV2. Згідно з одним з варіантів даного винаходу, С-кінцеві білки PTTV1а – і PTTV1b-ORF1 були експресовані та очищені в системі E.coli, відповідно. Електрофорез SDS-PAGE і Вестернблоттинг з використанням mAb проти His мітки показали, що обидва продукти, 1а- і 1b-ORF1, містили два поліпептиди, один з очікуваним розміром (близько 40 кДа), а інший – у вигляді передбачуваного гомодимера (близько 80 кДа) (див. Фігури 15 А – С). На Фігурі 15 А представлені результати SDS-PAGE неочищених і очищених продуктів 1а - ORF1. На Фігурі 15 В представлені результати очищених продуктів 1b-ORF1 і 1b-ORF1ctruc. На Фігурі 15 С представлені результати Вестерн-блоттингу очищених продуктів 1а - і 1b-ORF1 з використанням mAb проти His мітки. Стрілки з вістрям білого кольору вказують на білок ORF1 очікуваного розміру, тоді як стрілки з вістрям чорного кольору вказують на очікуваний димер білків ORF1. У порівнянні з експресією 2с - ORF1 процесований поліпептид не спостерігали. Як контроль для порівняння проводили експресію С – кінцевої процесованої ділянки 1b-ORF1 (1bORF1ctruc), що призвело до отримання поліпептиду з більш низькою молекулярною вагою у 20 UA 108359 C2 5 10 15 20 25 30 35 40 45 50 55 60 порівнянні з його С – кінцевою непроцесованою протилежною частиною 1b-ORF1 (див. Фігуру 15 В). Відповідно до одного варіанту даного винаходу, очищені білки С – кінцевої ділянки PTTV1а і PTTV1b-ORF1 застосовували для розробки методу генотип – специфічного Вестерн – блоттингу сироватки і методу ELISA, як описано вище для PTTV2. На Фігурі 16 представлені негативні (колонки 1-2) і позитивні (колонки 3-5) результати Вестерн – блоттингу і ELISA зразків сироватки із застосуванням 1а - ORF1 в якості антигена. При проведенні PTTV1а - і PTTV1bспецифічного методу ELISA застосовували ту ж кількість антигену (69 нг), розведення сироватки 1: 100 і розведення кон'югату IgG 1: 4000 (результати не представлені). Крім того, цей винахід передбачає діагностичний реагент для детектування інфекції, викликаної TTV, який включає моноклональне або поліклональне антитіло, очищене від природного господаря, наприклад, шляхом інокуляції свині свинячим TTV або імуногенною композицією за винаходом в імуногенній кількості для продукування вірусної інфекції та виділення антитіла з сироватки інфікованої свині. Або ж у експериментальних тварин можуть бути індуковані антитіла до природних або синтетичних поліпептидів, отриманих або експресованих з амінокислотних послідовностей або імуногенних фрагментів, що кодуються нуклеотидною послідовністю виділеного свинячого TTV. Наприклад, моноклональні антитіла можуть бути отримані з клітин гібридоми, які отримані з мишей, таких як, наприклад, миші лінії Ваlb/c, імунізованих поліпептидним антигеном, отриманим з нуклеотидної послідовності виділеного свинячого TTV. Селекція клітин гібридоми проводиться шляхом вирощування в системі, що містить гіпоксантин, тимідин і амідоптерин, у стандартному культуральному середовищі, наприклад, в модифікованому за способом Дульбекко середовищі Голка (DMEM) або мінімальному підтримуючому середовищі. Клітини гібридоми, які продукують антитіла, можуть бути клоновані з використанням методів, відомих з рівня техніки. Після цього дискретні колонії, що утворились, можуть бути поміщені в окремі комірки культуральних планшетів для культивування в прийнятному культуральному середовищі. Ідентифікація клітин, що секретують антитіла, проводиться звичайними методами скринінгу за допомогою відповідного антигена або імуногена. Культивування клітин гібридоми in vitro або in vivo шляхом отримання асцитної рідини у мишей після введення клітин гібридоми призводить до отримання бажаного моноклонального антитіла відомими методами. Згідно з іншим альтернативним методом капсидний білок свинячого TTV може бути експресований в системі експресії на основі бакуловірусів або в системі на основі E.coli згідно з відомими методиками. Експресований рекомбінантний капсидний білок свинячого TTV може бути використаний як антиген для діагностики методом твердофазного імуноферментного аналізу (ELISA). Наприклад, метод ELISA, оснований на застосуванні рекомбінантного капсидного антигена, може бути використаний для детектування антитіл до свинячого TTV у свиней та інших ссавців. Хоча метод ELISA є кращим, можуть застосовуватись й інші відомі діагностичні тести, такі як імунофлуоресцентний аналіз (IFA), імунопероксидазний метод (IРA) та ін. У випадку, коли це бажано, комерційний діагностичний метод ELISA, відповідно до цього винаходу, може бути застосований для діагностики інфекції у свиней, викликаної свинячим TTV. Приклади ілюструють застосування очищених білків ORF1 і ORF2 свинячого TTV для розробки методу ELISA для детектування антитіл до TTV у свиней. Сироватка, відібрана у свиней, інфікованих свинячим TTV, а також негативні зразки сироватки, відібрані у контрольних тварин, були застосовані для підтвердження цього методу. Було показано, що РTTV2 - специфічні, РTTV1а - специфічні і РTTV1b - специфічні антитіла специфічно розпізнають білки РTTV – ORF. Подальша стандартизація тесту методами, відомими фахівцю в цій галузі, може оптимізувати комерціалізацію методу діагностики свинячого TTV. Іншим аспектом (об'єктом) даного винаходу є унікальна імуногенна композиція, яка містить виділений свинячий TTV або антигенний білок, який кодується виділеним полінуклеотидом, описаним вище, та його застосування для індукції або вироблення антитіл. Композиція містить нетоксичний фізіологічно прийнятний носій і, необов'язково, один або більше ад'ювантів. Прийнятні носії, наприклад, такі як вода, фізіологічний розчин, етанол, етиленгліколь, гліцерин і т.д., обирають серед звичайних ексципієнтів, хоча можна додавати й інші складові. Для того щоб гарантувати фізичну сумісність і стабільність кінцевої композиції, можна проводити стандартні тести. Крім цього, згідно з даним винаходом передбачаються молекулярні вакцини проти свинячого вірусу гепатиту Torque teno (TTV) (infectious molecular) і нуклеотидні молекули свинячого вірусу гепатиту Torque teno (TTV) (віруса тонкого намиста), живі віруси, що продукуються при використанні нуклеотидної молекули, і ветеринарні вакцини для захисту 21 UA 108359 C2 5 10 15 20 25 30 35 40 45 50 55 60 свиней від свинячої TTV вірусної інфекції або захворювання, спричиненого сумісною інфекцією свинячого TTV та інших вірусів. Крім того, винахід передбачає імуногенні поліпептидні продукти експресії, які можна використовувати як вакцини. Молекула нової інфекційної ДНК свинячого TTV являє собою нуклеотидну молекулу, що кодує, принаймні, ділянку інфекційного PTTV1a-VA (SEQ ID NO: 9), PTTV1b-VA (SEQ ID NO: 10), PTTV2c-VA (SEQ ID NO: 11) або PTTV2c-VA (SEQ ID NO: 12) геному. Клон інфекційної PTTV ДНК, переважно, містить, щонайменше, один з ORF1 (відкритою рамкою зчитування), ORF2, ORF1/1 і ORF2/2 генів PTTV1 або PTTV2. Для створення тандемних повторів інфекційних PTTV клонів у поодиноку молекулу ДНК можна включити (інсерція) велику кількість копій PTTV1a-VA (SEQ ID NO: 9), PTTV1b-VA (SEQ ID NO: 10), PTTV2c-VA (SEQ ID NO: 11) або PTTV2c-VA (SEQ ID NO: 12) геному. Показано, що клонована геномна ДНК PTTV, зокрема PTTV1a-VA, PTTV1b-VA, PTTV2c-VA і тандемних повторів PTTV2b-RR, PTTV2c-RR за даним описом є інфекційною in vitro або in vivo, якщо її трансфікувати в клітини PK-15 та давати свиням. Цей новий, легко відтворюваний патогенний агент сам по собі прийнятний для розробки відповідної програми вакцинації для попередження PTTV інфекції у свиней. Згідно з іншим варіантом цього винаходу отримували три PTTV ДНК клони з однією копією PTTV ДНК геному при використанні прототипів ізолятів США PTTV1a-VA, PTTV1b-VA і PTTV2cVA за допомогою fusion ПЛР (ПЛР із зшиванням), відповідно. Кожну з повнорозмірних геномних ДНК вбудовували в клонуючий вектор pSC-B- amp/kan лігуванням на тупих кінцях. Сайт рестрикції BamH I являє собою унікальний сайт на трьох PTTV геномах, який створювали на обох кінцях трьох геномів з метою сприяти утворенню конкатемерів і, тим самим, імітувати TTV кільцевий геном. Поодинокі розщеплення обраної плазмідної ДНК кожного клону за допомогою BamH I призводили чітко до утворення двох різних фрагментів розміром 4.3 т.п.о. і 2.8- т.п.о. (Фіг. 18A). Фрагменти 4.3 т.п.о. являли собою каркасний вектор, тоді як фрагменти 2.8 т.п.о. являли собою вбудовану геномну ДНК PTTV. У випадку "пустого" вектора pSC-B- amp/kan, який розщеплюється тим же самим ферментом, спостерігається лише смуга 4.3 т.п.о. (Фіг. 18A). Отримані в результаті PTTV клони позначають pSC-PTTV1a, pSC-PTTV1b і pSC-PTTV2c, відповідно (Фіг. 17A-C). Далі, дві копії геному повнорозмірного PTTV2c-VA, що утворюється при використанні клона pSC-PTTV2c, лігували в тандемному повторі у вектор pSC-B- amp/kan, отримуючи клон pSC2PTTV2c-RR (Фіг. 17D). Порівняння паттернів поодинокого розщеплення pSC-PTTV2c і pSC2PTTV2c-RR за допомогою Afl II показало, що остання плазміда має додатковий фрагмент 2.8 т.п.о., що являє собою другу копію PTTV2c геному (Фіг. 18B, права панель). Після цього ми використовували ту ж стратегію клонування для отримання тандем-димерної форми PTTV2b ДНК клону, який утворився з TTV клону TTV2-#471942-full (Німеччина). Додатковий фрагмент розміром 2.8 т.п.о., що являє собою другу копію PTTV2b геному, був присутній у цій конструкції, позначеній як pSC-2PTTV2b-RR (Фіг. 17F), яку розщепляли лише за допомогою Hind III, у порівнянні з геномним аналогом з однією копією (Фіг. 18B, ліва панель), що свідчить про успішну конструкцію. Компетентність щодо реплікації конструйованих PTTV інфекційних клонів перевіряли за допомогою трансфекції in vitro клітин PK-15. ІФА (IFA) із застосуванням комерційних кролячих поліклональних антитіл проти PTTV2c ORF1 підтвердив, що конкатемери клонів як TTV2#471942-full, так і pSC-PTTV2c компетентні стосовно реплікації, відповідно (Фіг. 19A і Фіг. 20A). Пасаж (субкультивування) трансфікованих клітин не виключає і не послаблює флуоресцентні сигнали (Фіг. 19B і Фіг. 20B), що свідчить про те, що експресія ORF1 білків обумовлена PTTV2 конкатемерами, які імітують природні PTTV2b або PTTV2c кільцеві молекули. Жодних флуоресцентних сигналів не спостерігали в клітинах із симульованою ДНК-трансфекцією із застосуванням неімунної кролячої сироватки в якості антитіла для ІФА (IFA) виявлення (дані не представлені). Також показано, що конкатемери pSC-PTTV1a клону є компетентними щодо реплікації із застосуванням антитіла проти PTTV1a ORF1 (Фіг. 21). Позитивні флуоресцентні сигнали були локалізовані в ядрі трансфікованих або субкультивованих клітин, що вказує на те, що свинячі TTV, очевидно, реплікуються в ядрах клітин. Це не є несподіваним, оскільки цирковірус свиней (PCV, ЦВС) має аналогічний паттерн експресії in vitro. Пряма трансфекція тандем-димерної форми клону pSC-2PTTV2b-RR або pSC-2PTTV2c-RR в клітинах PK-15 призводить до вірусної реплікації і продукує ORF1 капсидний антиген. ІФА (IFA) із застосуванням антитіл проти PTTV2 ORF1 підтвердив, що обидва клони є компетентними стосовно реплікації, а позитивні ORF1 антигени локалізовані в ядрах (Фіг. 22A і B). Згідно з одним варіантом цього винаходу для інокуляції свиней можна застосовувати інфекційні клони свинячого TTV, які потім виявляють імунну відповідь тварини-господаря і 22 UA 108359 C2 5 10 15 20 25 30 35 40 45 50 55 60 стимулюють продукування нейтралізуючих антитіл. В одному конкретному варіанті цього винаходу два тандем-димерні PTTV2 клони були інфекційними (контагіозними), при введенні їх шляхом ін'єкцій в лімфатичні вузли та м'язи звичайних свиней. Для тестування in vivo інфікуючої здатності (інфекційності) PTTV2 молекулярних клонів звичайних свиней інокулюють клоном pSC-2TTV2b-RR або pSC-2TTV2c-RR. Зразки сироватки беруть у тварин на 0, 7, 14, 21 та 28 дні після інокуляції (ДПІ). PTTV2 ДНК виявляли у pSC2TTV2c-RR- інокульованих свиней, починаючи з 7 ДПІ (#92), 14 ДПІ (#188 і #191) і з 21 ДПІ (#180), відповідно (Фіг. 23A, права панель). PTTV вірусемія з'являлась пізніше у свиней, інокульованих клоном pSC-2TTV2b-RR: у двох свиней вона з'являлась на 14 ДПІ (#189 і #192), у однієї - на 21 ДПІ (#181) і ще у однієї - на 28 ДПІ (#193) (Фіг. 23A, ліва панель). Вірусне навантаження підвищувалось протягом цього курсу у всіх інокульованих свиней, найвище вірусне навантаження спостерігалось у них на 28 ДПІ перед розтином, як було визначено за допомогою PTTV2-специфічної ПЛР в режимі реального часу (Фіг. 23A). Продукти ПЛР в реальному часі, ампліфіковані при використанні обраних свиней, секвенували і виявили, що вони мають послідовності, ідентичні послідовностям відповідних областей pSC-2TTV2b-RR або pSC-2TTV2c-RR (дані не представлені). Всі інокульовані свині були негативними щодо PTTV2 ORF1 антитіл на 0 і 7 ДПІ. На 14 ДПІ всі четверо pSC-2TTV2b-RR-інокульованих свиней сероконвертували (змінили профіль антитіл у сироватці) в анти-PTTV2 ORF1 IgG, в той час як pSC-2TTV2c-RR- інокульована група сероконвертувала на 14 (#92 і #180), 21 (#191) і 28 (#188) ДПІ, відповідно (Фіг. 23B). Результати свідчать про те, що відбулось активне інфікування свинячим вірусом TTV2b або TTV2c. Вакцини інфекційних вірусних та інфекційних молекулярних ДНК клонів і способів їх застосування також включені в обсяг даного винаходу. Інокульованих свиней захищають від вірусної інфекції та спричинених нею захворювань, викликаних TTV2 інфікуванням або коінфікуванням. Новий спосіб захищає свиней, які потребують захисту проти вірусної інфекції, шляхом введення свині імунологічно ефективної кількості вакцини за винаходом, наприклад, такої як вакцина, що містить імуногенну кількість інфекційної PTTV ДНК, плазміди або вірусного вектора, що містить інфекційний клон ДНК PTTV, рекомбінантної ДНК PTTV, продуктів експресії - поліпептидів, очищеного рекомбінантного ORF1 капсидного білка і т.д., що експресується в бактеріях або в бакуловірусах. Для того щоб забезпечити широкий спектр захисту проти вірусних інфекцій, свиням можна одночасно давати інші антигени, такі як PRRSV, PPV, інші інфекційні свинячі агенти та імуностимулятори. Вакцини містять, наприклад, інфекційні вірусні та молекулярні ДНК клони, клонований PTTV інфекційний ДНК геном у прийнятних плазмідах або векторах, наприклад, таких як pSC-B вектор, авірулентний живий вірус, інактивований вірус, субодинична вакцина на основі експресованого рекомбінантного капсидного білка, і т.д. у поєднанні з нетоксическим фізіологічно прийнятним носієм і, необов'язково, з одним або більше ад'ювантами. Вакцина може також містити інфекційний TTV2 молекулярний ДНК клон за даним описом. Інфекційна PTTV ДНК, плазмідна ДНК, що несе інфекційний вірусний геном, та живі віруси є переважними, причому живі віруси є найбільш прийнятними. Авірулентна жива вірусна вакцина за даним винаходом має переваги перед традиційними вірусними вакцинами, в яких використовуються або ослаблені (атенуйовані), живі віруси, які підлягають ризику повернутись до вірулентного стану, або клітини вбитої культури поширять цілий вірус, який може не дати змоги індукувати достатню імунну відповідь антитіла для захисту проти вірусного захворювання. Вакцини та способи їхнього застосування також входять в обсяг цього винаходу. Інокуляція ссавців захищає проти серйозної вірусної інфекції, вона може також забезпечити захист від захворювання, пов'язаного з коінфекцією PTTV, такого як синдром дерматиту і нефропатії поросят (PDNS), мультисистемний синдром виснаження після відлучення поросят від свиноматки (PMWS) та іншого спорідненого захворювання. Вакцини містять, наприклад, інактивований або ослаблений свинячий TTV вірус, нетоксичний, фізіологічно прийнятний носій і, необов'язково, один або більше ад'ювантів. Ад'ювант, який можна вводити у поєднанні з вакциною, за даним винаходом, являє собою речовину, що підвищує імунологічну реакцію свині на вакцину. Ад'ювант можна вводити одночасно з вакциною і в ту ж саму область, що й вакцину, або в різний час, наприклад, як бустер-ін'єкції. Краще також вводити ад'юванти свині способом, що відрізняється від способу введення вакцини, або в іншу область від введення вакцини. Прийнятні ад'юванти включають, але без обмеження, гідроксид алюмінію (квасці), імуностимулюючі комплекси (ISCOMS), неіонні блок-полімери або співполімери, цитокіни (подібні до IL-1, IL-2, IL-7, IFN- α, IFN- β, IFN- γ, і т.д.), сапоніни, монофосфорилліпід A (MLA, МФЛ), мурамілдипептиди (MDP) і т.п. Інші прийнятні ад'юванти включають, наприклад, алюмінію калію сульфат, термолабільний або 23 UA 108359 C2 5 10 15 20 25 30 35 40 45 50 55 60 термостабільний ентеротоксин, виділений з Escherichia coli, холерний токсин або його Всубодиницю, дифтерійний токсин, правцевий токсин, коклюшний токсин, неповний або повний ад'ювант Фрейнда тощо. Ад'юванти на основі токсинів, таких як дифтерійний токсин, правцевий токсин і коклюшний токсин, можна інактивувати перед застосуванням, наприклад, обробкою формальдегідом. Крім цього, вакцини можуть містити додаткові антигени для ініціації імунологічної активності інфекційних PTTV ДНК клонів, наприклад, такі як вірус свинячого репродуктивно-респіраторного синдрому (PRRSV), свинячий парвовірус (PPV), інші інфекційні агенти та імуностимулятори для свиней. Нові вакцини за даним винаходом не обмежуються будь-яким конкретним способом або методом виготовлення. Клоновані вірусні вакцини включають, але без обмеження, інфекційні ДНК вакцини (тобто такі, що використовують плазміди, вектори або інші звичайні носії для безпосередньої ін'єкції ДНК свиням), живі вакцини, модифіковані живі вакцини, інактивовані вакцини, субодиничні вакцини, атенуйовані вакцини, генно-інженерні вакцини тощо. Ці вакцини готують стандартними методами, відомими з рівня техніки. Іншою перевагою є те, що кращий, якому слід надати перевагу, живий вірус, за даним винаходом забезпечує генетично стійку вакцину, яку легше приготувати, зберігати і доставляти, ніж інші типи атенуйованих вакцин. В іншій кращій вакцині даного винаходу використовуються прийнятні плазміди для доставки непатогенного ДНК- клону свиням. На відміну від традиційних вакцин, в яких використовують цілий вірус, що передається культурою живих або вбитих клітин, цей винахід передбачає безпосередню інокуляцію свиней плазмідною ДНК, що містить інфекційний вірусний геном. Додаткові генно-інженерні вакцини, бажані в цьому винаході, отримують методами, відомими з рівня техніки. Такі методи включають, але без обмежень, подальшу маніпуляцію з рекомбінантною ДНК, модифікацію або заміни амінокислотних послідовностей рекомбінантних білків і т.п. Генно-інженерні вакцини на основі рекомбінантної ДНК отримують, наприклад, ідентифікуючи альтернативні ділянки вірусного гену, які кодують білки, що відповідають за індукцію більш сильної імунної або протективної (захисної) відповіді у свиней (наприклад, білки, отримані при використанні ORF1, ORF1/1, ORF2, ORF2/2 і т.д.). Такі ідентифіковані гени або імунодомінантні фрагменти можна клонувати в стандартні вектори для експресії білків, такі як бакуловірусний вектор, і використовувати для інфікування відповідних клітин-господарів (див., наприклад, O'Reilly et al., "Baculovirus Expression Vectors: A Lab Manual, ” Freeman & Co., 1992). Клітини-господарі культивують, для того щоб вони експресували задані вакцинні білки, які можна очистити до потрібного ступеня чистоти і готувати у вигляді прийнятного вакцинного продукту. Рекомбінантні субодиничні вакцини основані на ORF1 капсидних білках PTTV1a, PTTV1b і PTTV2, які експресуються бактеріями (Фіг. 10, Фіг. 15) або бакуловірусами. Якщо клони зберігають будь-які небажані природні властивості викликати захворювання, можна також точно вказати нуклеотидні послідовності у вірусному геномі, які відповідають за будь-яку залишкову вірулентність, і методами генної інженерії зробити вірус авірулентним, наприклад, методом сайт-спрямованого мутагенезу. Сайт- спрямований мутагенез дає змогу додавати, видаляти або проводити заміщення одного або більшої кількості нуклеотидів (див., наприклад, Zoller et al., DNA 3: 479-488, 1984). Синтезують олігонуклеотид, який містить задану мутацію, і гібридизують з ділянкою однониткової (одноланцюгової) вірусної ДНК. Гібридну молекулу, отриману в результаті цієї процедури, використовують для трансформації бактерії. Після цього виділену двохниткову ДНК, яка містить відповідну мутацію, використовують для отримання повнорозмірної ДНК за допомогою лігування до рестрикційного фрагмента останньої, надалі її трансфікують в прийнятну клітинну культуру. Лігування геному до прийнятного вектора для перенесення можна проводити будь-яким стандартним методом, відомим пересічному фахівцеві в даній технічній галузі. Трансфекцію вектора в клітинигосподаря для продукування вірусного потомства можна здійснювати будь-якими звичайними методами, такими как трансфекція, опосередкована фосфатом кальцію або DEAE- декстраном, електропорація, злиття протопластів та з використанням інших загальновідомих методів (див., наприклад, Sambrook et al., "Molecular Cloning: A Laboratory Manual, ” Cold Spring Harbor Laboratory Press, 1989). Далі клонований вірус виявляє заданую мутацію. Або ж ще можна синтезувати два олігонуклеотиди, які містять відповідну мутацію. Їх можна гібридизувати з утворенням двохниткової ДНК, яку можна вбудовувати у вірусну ДНК, отримуючи повнорозмірну ДНК. Імунологічно ефективну кількість вакцин за даним винаходом вводять свині, яка потребує захисту проти вірусної інфекції. Імунологічно ефективну кількість або імуногенну кількість, якою 24 UA 108359 C2 5 10 15 20 25 30 35 40 45 50 55 60 інокулюють свиню, можна легко визначити або просто титрувати за допомогою стандартного тестування. Ефективна кількість являє собою таку кількість, за допомогою якої можна досягти імунологічної відповіді на вакцину, достатньої для захисту свині, яка зазнала дії (експонована до) PTTV віруса. Переважно, свиня захищається тією мірою, в якій значно зменшуються, стають менш інтенсивними або повністю попереджуються від одного до всіх несприятливі фізіологічні симптоми або ефекти вірусного захворювання. Вакцину можна вводити у вигляді разової дози або у вигляді повторних доз. Інтервали доз можуть становити, наприклад, близько 1 – 1000 мікрограмів плазмідної ДНК, що містить інфекційний химерний ДНК-геном (залежно від концентрації імуноактивного компонента вакцини), переважно, 100 – 200 мікрограмів TTV ДНК клону свині, але дози не повинні містити кількість вірусного антигена, достатню для того, щоб викликати побічну (патологічну) реакцію або фізіологічні симптоми вірусної інфекції. З рівня техніки відомі методи визначення або титрування прийнятних доз активного антигенного агента з метою встановити мінімальні ефективні дози з урахуванням ваги свині, концентрації антигена та інших стандартних чинників. Переважно, як вакцину використовують інфекційний вірусний ДНК-клон, або живий інфекційний вірус можна отримувати in vitro, після чого живий вірус можна використовувати як вакцину. У цьому випадку свині можна давати, наприклад, близько 50 – 10000 TCID50 (доза інфікуюча 50 % тканинної культури) живих вірусів. Нові вакцини за винаходом не обмежуються будь-яким конкретним типом або методом отримання. Вакцини включають, але без обмеження, модифіковані живі вакцини, інактивовані вакцини, субодиничні вакцини, атенуйовані вакцини, генно-інженерні вакцини тощо. Перевага живих вакцин полягає в тому, що у реципієнта вакцини активуються всі можливі імунні реакції, включаючи системні, місцеві, гуморальні та клітинні імунні реакції. До недоліків живих вірусних вакцин, які можуть звести нанівець всі переваги, є ймовірність забруднення живими випадковими вірусними агентами або підвищений ризик того, що in situ вірус може поновити вірулентність. Для приготування інактивованих вірусних вакцин ріст вірусів та продукування вірусів може відбуватись у культивованих клітинних лініях свиней, наприклад, але без обмеження, в таких, як клітини PK-15. Потім інактивацію вірусів за допомогою серійних пасажів (субкультивування) оптимізують за протоколами, добре відомими пересічним фахівцям в даній технічній галузі, або, здебільшого, відповідно до методів даного опису. Інактивовані вірусні вакцини можна приготувати за допомогою обробки TTV свині інактивуючими речовинами, такими як формалін або гідрофобні розчинники, кислоти і т.д., опроміненням ультрафіолетовим світлом або рентгенівськими променями, нагріванням і т.д. Інактивацію проводять у спосіб, відомий з рівня техніки. Наприклад, при хімічній інактивації відповідний зразок віруса або зразок сироватки, що містить вірус, протягом достатнього проміжку часу обробляють інактивуючим агентом, в достатній кількості або достатньої концентрації, при достатньо високих (або низьких, залежно від інактивуючого агента) значеннях температури або pH, інактивуючи вірус. Інактивацію при нагріванні проводять при такій температурі та протягом такого проміжку часу, що є достатніми для інактивації віруса. Інактивацію опроміненням проводять, використовуючи довжину світлової хвилі або інше джерело енергії протягом проміжку часу, достатнього для інактивації віруса. Вірус вважають інактивованим, якщо він не здатний інфікувати клітину, сприйнятливу до інфікування. Приготування субодиничних вакцин здебільшого відрізняється від приготування модифікованої живої вакцини або інактивованої вакцини. Перед приготуванням субодиничної вакцини слід ідентифікувати протективні (захисні) або антигенні компоненти вакцини. У цьому винаході антигенні компоненти PTTV були ідентифіковані як ORF1 капсидні білки вірусів PTTV1a, PTTV1b і PTTV2, які в даному винаході експресували та очищали в Escherichia coli (E. coli), та іншій системі експресії, такій як бакуловірусна система експресії, для застосування в якості субодиничних рекомбінантних вакцин на основі капсидних білків. Такі протективні або антигенні компоненти включають деякі амінокислотні сегменти або фрагменти вірусних капсидних білків, які викликають особливо сильну протективну або імунологічну реакцію у свиней; власне поодинокі або множинні вірусні капсидні білки, їх олігомери і вищого порядку асоціації вірусні капсидні білки, які утворюють субструктури вірусів або частини чи ланки таких субструктур; що можуть бути ідентифіковані, олігоглікозиди, гліколіпіди або глікопротеїни, наприклад, такі як ліпопротеїни або ліпідні групи, асоційовані з вірусом, присутні на або поряд з поверхнею віруса або у вірусних субструктурах, і т.д. Переважно, як антигенний компонент субодиничної вакцини використовують ORF1 білок. Можуть також використовуватись інші білки, наприклад, такі, що кодуються нуклеотидною послідовністю в ORF2, ORF1/1 і ORF2/2 гені. Ці імуногенні компоненти легко ідентифікуються методами, відомими з рівня техніки. Після 25 UA 108359 C2 5 10 15 20 25 30 35 40 45 50 55 60 ідентифікації протективні або антигенні ділянки віруса (тобто, "субодиниця") очищаються і/або клонуються методами, відомими з рівня техніки. Субодинична вакцина забезпечує переваги перед іншими вакцинами на основі живого віруса, оскільки субодиниця, така як високоочищені субодиниці віруса, менш токсична, ніж цілий вірус. Якщо субодиничну вакцину отримують методом рекомбінантної ДНК, експресію клонованої субодиниці, такої як гени ORF1, ORF2. ORF1/1 і ORF2/2, можна здійснювати, наприклад, методом, представленим вище, і можна також оптимізувати методами, відомими фахівцям в даній області техніки (див., наприклад, "Molecular Cloning: A Laboratory Manual, ” Cold Spring Harbor Laboratory, Cold Spring Harbor, Mass. (1989)). З іншого боку, якщо субодиниця, що використовується, являє собою інтактну структурну ознаку віруса, таку як цілий капсидний білок, то у цьому випадку метод його виділення з віруса можна оптимізувати. У будь-якому випадку після оптимізації протоколу інактивації протокол очистки субодиниці можна оптимізувати перед отриманням. Для приготування атенуйованих вакцин живий патогенний вірус спочатку атенуюють (роблять непатогенним або нешкідливим) за допомогою методів, відомих з рівня техніки, або, здебільшого, за даним описом. Наприклад, атенуйовані віруси можна приготувати за допомогою методу даного винаходу, який включає новий серійний пасаж на ембріонах свиней. Атенуйовані (ослаблені) віруси можна виявити в природі; вони можуть містити природні генні делеції або, в якості альтернативи, патогенні віруси можна атенуювати, здійснюючи делеції або мутації в генах. Атенуйовані та інактивовані вірусні вакцини містять переважно вакцини за даним винаходом. Генно-інженерні вакцини, також бажані у цьому винаході, отримують методами, відомими з рівня техніки. Такі методи включають, але без обмеження, затосування РНК, рекомбінантної ДНК, рекомбінантних білків, живих вірусів і т.п. Наприклад, після очистки вірус дикого типу можна виділяти з відповідних клінічних біологічних зразків, таких як сироватка, кал, слина, сперма і тканини, методами, відомими з рівня техніки, переважно, методами, що розкриваються в цьому описі, використовуючи інфікованих свиней або інфіковані відповідні лінії клітин. ДНК вилучають з біологічно чистого віруса або інфекційного агента методами, відомими з рівня техніки, і очищають методами, відомими з рівня техніки, переважно, ультрацентрифугуванням в градієнті CsCl. кДНК вірусного геному клонують у відповідного господаря методами, відомими з рівня техніки (Maniatis et al., див. вище), після чого вірусний геном аналізують, визначаючи області геному, суттєві для продукування антигенних ділянок віруса. Далі процедура, як правило, така ж, як для модифікованої живої вакцини або субодиничної вакцини. Генно-інженерні вакцини на основі методу рекомбінантної ДНК отримують, наприклад, ідентифікуючи ділянку вірусного гена, який кодує білки, що відповідають за індукцію більш сильної імунної або протективної відповіді у свиней (наприклад, білки, отримані з використанням ORF1, ORF2, ORF1/1 і ORF2/2, і т.д.). Такі ідентифіковані гени або імунодомінантні фрагменти можна клонувати в стандартні вектори для експресії білків, такі як бакуловірусний вектор, і використовувати для інфікування відповідних клітин-господарів (див., наприклад, O'Reilly et al., "Baculovirus Expression Vectors: A Lab Manual, ” Freeman & Co., 1992). Клітини-господарі культивують, для того щоб вони експресували задані вакцинні білки, які можна очищати до потрібного ступеня чистоти і готувати у вигляді прийнятного вакцинного продукту. Генно-інженерні білки, що застосовуються у вакцинах, можна експресувати, наприклад, в клітинах комах, клітинах дріжджів або клітинах ссавців. Генно-інженерні білки, які можна очищати або виділяти звичайними методами, можна безпосередньо інокулювати свиням або іншим ссавцям, для того щоб захистити від свинячого TTV. Лінію клітин комах (подібну до sf9, sf21 або HIGH-FIVE) можна трансформувати за допомогою вектора для переносу, який містить полінуклеїнові кислоти, отримані при використанні віруса або скопійовані з вірусного геному, який кодує один або більше імунодомінантних білків віруса. Вектор для перенесення включає, наприклад, лінеаризовану ДНК бакуловіруса та плазміду, яка містить задані полінуклеотиди. Лінію клітин-господарів можна сумісно трансфікувати за допомогою лінеаризованої ДНК бакуловіруса і плазміди, для того щоб отримати рекомбінантний бакуловірус. Крім того, ДНК з виділеного свинячого TTV, яка кодує один або більше капсидних білків, можна вбудовувати в живі вектори, такі як поксвірус (вірус групи віспи) або аденовірус, та застосовувати як вакцину. Імунологічно ефективну кількість вакцини за даним винаходом вводять свиням або іншим ссавцям, яким необхідний захист проти вказаної інфекції чи синдрому. "Імунологічно ефективну 26 UA 108359 C2 5 10 15 20 25 30 35 40 45 50 55 60 кількість" можна легко визначити або тестувати за допомогою стандартного тестування. Ефективна кількість означає таку кількість, за допомогою якої можна досягти імунологічної відповіді на вакцину, достатньої для захисту свині або іншого ссавця, який зазнав дії віруса свинячого TTV або коінфекції свинячого TTV, яка може викликати синдром дерматиту і нефропатії у поросят (PDNS), мультисистемний синдром виснаження після відлучення поросят від свиноматки (PMWS) або споріднене захворювання. Здебільшого, свиню або ссавця іншого виду захищають тією мірою, коли значно зменшуються, стають менш інтенсивними або повністю попереджуються від одного до всіх несприятливі фізіологічні симптоми або ефекти вірусного захворювання. Вакцину можна вводити у вигляді разової дози або у вигляді повторних доз. Дози можуть становити, наприклад, близько 1 – 1,000 мікрограмів вірусного антигена (залежно від концентрації імуноактивного компонента вакцини), але не повинні містити кількість вірусного антигену, достатню для того, щоб викликати побічну реакцію або фізіологічні симптоми вірусної інфекції. З рівня техніки відомі методи визначення або титрування прийнятних доз активного антигенного агента з урахуванням ваги птаха або ссавця, концентрації антигена та інших стандартних чинників. Вакцину можна вводити свиням. Вакцину також можна давати людям, таким як свиноводи (працівники свиноферм), що перебувають під підвищеним ризиком інфікування вірусним агентом. Висловлюється припущення, що можна створити вакцину на основі TTV свині, щоб забезпечити загальний захист проти свинячого і TTV людини. Іншими словами, вакцину на основі свинячого TTV можна, переважно, створювати таким чином, щоб захистити від інфікування TTV людини, внаслідок використання так званого "методу Дженнера" (тобто за методом Едварда Дженнера можна використовувати вакцину віруса коров'ячої віспи проти звичайної віспи людини). Бажано, щоб вакцина вводилась безпосередньо свиням або ссавцям іншого виду, які не зазнали впливу TTV віруса. Вакцину зручно вводити перорально, інтрабуккально, інтраназально, трансдермально, парентерально тощо. Парентеральний шлях введення включає, але без обмеження, внутрішньом'язовий, внутрішньовенний, інтраперитонеальний та підшкірний шляхи. Для введення у вигляді рідини вакцину даного винаходу можна приготувати у вигляді водного розчину, сиропу, еліксиру, настоянки тощо. Такі лікарські форми відомі з рівня техніки, здебільшого їх готують, розчиняючи антиген та інші звичайні домішки у відповідних системах носія або розчинника. Прийнятні носії або розчинники включають, але без обмеження, воду, фізіологічний сольовий розчин, етанол, етиленгліколь, гліцерин тощо. Стандартні добавки являють собою, наприклад, дозволені барвники, коригенти (що надають смаку та аромату), підсолоджувачі та протимікробні речовини – консерванти, такі як тимеросал (етилртутьтіосаліцилат натрію). Такі розчини можна стабілізувати, наприклад, додаючи частково гідролізований желатин, сорбіт або клітинне культуральне середовище, і можна забуферити звичайними методами, використовуючи реагенти, відомі з рівня техніки, такі як гідрофосфат натрію, дигідрофосфат натрію, гідрофосфат калію, дигідрофосфат калію, їх суміш і т.п. Рідкі лікарські форми можуть включати суспензії та емульсії, які містять суспендуючі або емульгуючі агенти у поєднанні з іншими стандартними складовими. Ці типи рідких лікарських форм можна приготувати за допомогою звичайних методів. Наприклад, суспензії можна приготувати за допомогою колоїдного млинка. Емульсії можна приготувати, використовуючи, наприклад, гомогенізатор. Парентеральні лікарські форми, створені для введення у вигляді ін'єкції в системи рідин організму, потребують відповідної ізотонічності та буферизації рН до рівнів, що відповідають рівням рідин організму ссавця. За необхідності ізотонічність можна відповідним чином коригувати, застосовуючи хлорид натрію чи інші солі. Для підвищення розчинності інгредієнтів у лікарській формі та стійкості рідкого препарату можна застосовувати відповідні розчинники, такі як етанол або пропіленгліколь. Інші добавки, які можна застосовувати у вакцині за даним винаходом, включають, але без обмеження, декстрозу, звичайні антиоксиданти та звичайні хелатуючі агенти, такі, як етилендіамінтетраоцтова кислота (ЕДТА). Парентеральні лікарські форми необхідно також стерилізувати перед застосуванням. Наведені нижче приклади демонструють деякі аспекти цього винаходу. Однак, зрозуміло, що ці приклади наведені лише для ілюстрації і не претендують на те, щоб бути вичерпними стосовно умов та обсягу даного винаходу. Варто розуміти, що, коли наводяться типові умови реакції (наприклад, температура, тривалість реакції тощо), то можуть також застосовуватись умови, що знаходяться як вище, так і нижче вказаних інтервалів, хоча вони, як правило, є менш прийнятними. Реакції в прикладах проводять при кімнатній температурі (від, близько, 23 °C до, 27 UA 108359 C2 5 10 15 20 25 30 35 40 45 50 55 60 близько, 28 °C) і при атмосферному тиску. Якщо не вказано щось інше, в даному описі частини та відсотки наводяться як вагові частини та вагові відсотки, а всі значення температури наводяться в градусах за Цельсієм. Приклад 1 Екстракція вірусної ДНК, вкладена ПЛР і геномна ПЛР: У дослідженні використовували прийнятні зразки сироватки та насінної рідини від 20 звичайних дорослих кнурів зі свиноферми у Віргинії. Тотальну ДНК виділяли з 20 зразків сироватки і 19 зразків насінної рідини, використовуючи міні-набір для виділення ДНК (QIА.к.mp DNA mini kit (Qiagen)). Для скринінгу на позитивні PTTV-вмісні зразки спочатку здійснювали ампліфікацію консервативних областей в UTR PTTV1 і PTTV2 методом вкладеної ПЛР, використовуючи полімеразу AmpliTag Gold (Applied Biosystems). Дві пари праймерів, які застосовували для ампліфікації фрагменту A в PTTV1, являли собою TTV1-mF (SEQ ID NO: 45)/TTV1-mR (SEQ ID NO: 46) (для першого раунду ПЛР) і TTV1-nF (SEQ ID NO: 47)/TTV1-nR (SEQ ID NO: 48) (для другого раунду ПЛР), тоді як дві пари праймерів, що застосовувались для ампліфікації фрагменту D в PTTV2, являли собою TTV2-mF (SEQ ID NO: 49)/TTV2-mR (SEQ ID NO: 50) (для першого раунду ПЛР) і TTV2-nF (SEQ ID NO: 51)/TTV2-nR (SEQ ID NO: 52) (для другого раунду ПЛР; Фіг. 1A і Таблиця 1). Для того щоб ампліфікувати повнорозмірні геномні послідовності як PTTV1, так і PTTV2, ми спочатку проводили зворотну геномну ПЛР, використовуючи пару консервативних генспецифічних праймерів TTV1-IF (SEQ ID NO: 1)/TTV1-IR (SEQ ID NO: 4), локалізованих в області A для PTTV1, та іншу пару ген-специфічних праймерів TTV2-IF (SEQ ID NO: 5)/TTV2-IR (SEQ ID NO: 8), локалізованих в області D для PTTV2, відповідно, зі злитою Геркулазой II ДНКполімеразою (Stratagene) згідно інструкцій виробника. Жодних ПЛР- продуктів очікуваного розміру виявлено не було. Після цього ми створили нові набори праймерів для ампліфікації двох областей, що охоплюють повні PTTV1 і PTTV2 геноми, у другому раунді ПЛР, відповідно (Фіг. 1A). Пара праймерів, яку використовували для ампліфікації фрагментів B і C в PTTV1, являла собою TTV1-IF (SEQ ID NO: 1)/TTV1-2340R (SEQ ID NO: 2) і TTV1-2311F (SEQ ID NO: 3)/TTV1-IR (SEQ ID NO: 4), відповідно, тоді як пара праймерів, що використовується для ампліфікації фрагментів E і F в PTTV2, являла собою TTV2-IF (SEQ ID NO: 5)/TTV2-2316R (SEQ ID NO: 6) і TTV2-GCF (SEQ ID NO: 7)/TTV2-IR (SEQ ID NO: 8), відповідно (Фіг. 1A і Таблиця 1). Фрагменти C і F містять багаті на вміст GC області PTTV1 і PTTV2, відповідно. Ампліфіковані ПЛР-продукти вирізали окремо, очищали та послідовно клонували у вектор pSC-B-amp/kan (Stratagene), використовуючи набір для ПЛР- клонування StrataClone Blunt PCR cloning kit, відповідно до рекомендацій виробника (Stratagene) з подальшим секвенуванням ДНК. Приклад 2 Скринінг зразків, позитивних щодо свинячого TTV, взяті у свиней на фермі у Віргинії Нещодавно у свиней в різних географічних регіонах детектували ДНК свинячого TTV методом вкладеної ПЛР на основі UTR (нетрансльовааної) послідовності японського PTTV1 штаму Sd-TTV31 (McKeown et al., 2004, див. вище). З урахуванням нещодавньої ідентифікації PTTV2, для ампліфікації області A PTTV1 і області D PTTV2, застосовували два різні набори праймерів для вкладеної ПЛР, відповідно (Фіг. 1A) (Ellis et al., 2008, див. вище; Kekarainen, T., Sibila, M., and Segales, J. (2006). Prevalence of swine Torque teno virus in post-weaning multisystemic wasting syndrome (PMWS)-affected and non-PMWS-affected pigs in Spain. J Gen Virol 87(Pt 4), 833-7; Krakowka et al., 2008, supra). Аналогічний метод виявлення був також застосований в даному дослідженні для ідентифікації штамів PTTV від свиней у Сполучених Штатах. Для того щоб провести скринінг на зразки, позитивні щодо вітчизняних PTTV1- або PTTV2-вірусів, для подальшого використання з метою визначення повнорозмірних геномних послідовностей методом вкладеної ПЛР, проводять аналіз 20 зразків сироватки (SR#1-20) і 19 зразків насінної рідини (SM#1-18 і SM#20), взятих у 20 свиней (кнурів) на фермі у Віргинії. На наш подив, всі 20 зразків сироватки були позитивними щодо PTTV1, а 19 також виявились позитивнми стосовно PTTV2 (за винятком SR#18). На противагу, лише один зразок насінної рідини (SM#6) був PTTV1- позитивним і 3 зразки насінної рідини (SM#8, 9 і 20) виявились PTTV2- позитивними. Цей результат узгоджувався з результатами нещодавнього дослідження, проведеного в Іспанії, в якому було показано, що зразки насінної рідини свиней є позитивними щодо PTTV ДНК (Kekarainen, T., Lopez-Soria, S., and Segales, J. (2007). Detection of swine Torque teno virus genogroups 1 and 2 in boar sera and semen. Theriogenology 68(7), 966-71), і, таким чином, є підстава висловити припущення про можливу вертикальну передачу PTTV. Однак, швидкість розповсюдження як PTTV1, так і PTTV2 в насінній рідині значно нижча, ніж у сироватці, що свідчить про те, що прямий зв'язок між присутністю PTTV ДНК в сироватці та насінній рідині однієї й тієї ж свині відсутній. 28
ДивитисяДодаткова інформація
Назва патенту англійськоюPorcine torgue teno virus vaccines and diagnosis
Автори англійськоюMeng, Xiang-Jin, Huang, Yaowei
Автори російськоюМенг Сян-Цзинь, Хуанг Яовей
МПК / Мітки
МПК: C12Q 1/68, A61K 39/12, C12N 15/63, C12N 15/34, G01N 33/569
Мітки: вірусу, інфекцій, свинячого, torque, вірусом, цим, діагностики, викликаних, основі, вакцина, спосіб
Код посилання
<a href="https://ua.patents.su/86-108359-vakcina-na-osnovi-svinyachogo-virusu-torque-teno-ta-sposib-diagnostiki-infekcijj-viklikanikh-cim-virusom.html" target="_blank" rel="follow" title="База патентів України">Вакцина на основі свинячого вірусу torque teno та спосіб діагностики інфекцій, викликаних цим вірусом</a>
Попередній патент: Mimetик sмас
Наступний патент: Тверда фармацевтична композиція кортикостероїду, що розпадається у ротовій порожнині
Випадковий патент: Спосіб лікування доброякісних епітеліальних захворювань шкіри та слизової оболонки вульви