Спосіб детекції модифікованої житньої транслокації 1rs.1bl в селекційних формах м’якої пшениці
Номер патенту: 59084
Опубліковано: 10.05.2011
Автори: Сиволап Юрій Михайлович, Сударчук Людмила Валеріївна, Чеботар Сабіна Віталіївна
Формула / Реферат
Спосіб детекції модифікованої житньої транслокації 1RS.1BL в селекційних формах м'якої пшениці, що включає ПЛР-аналіз ДНК рослин з молекулярними маркерами до локусів Xrems 1303, w-secalin, Gli-B1, Xgwm18, Xgwm550, який відрізняється тим, що проводять детектування рослин із модифікованою житньою транслокацією 1RS.1BL і виявляють рекомбінантні генотипи за даною транслокацією на будь-якій стадії розвитку рослини, підготовляють зразки до аналізу.
Текст
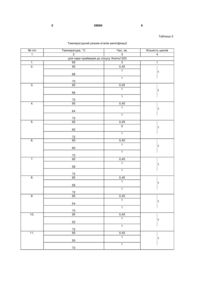
Спосіб детекції модифікованої житньої транслокації 1RS.1BL в селекційних формах м'якої пше 3 59084 кації вихідної форми, так і рекомбінантну модифіковану транслокацію. З метою усунення вказаного недоліку ми пропонуємо систему ДНК-маркерів для детекції модифікованої транслокації 1RS.1BL у генетичному матеріалі на ранній стадії розвитку рослини, що дозволяє проводити відбір рослин з модифікованою 1RS.1BL транслокацією. В основу корисної моделі поставлено ПЛРаналізування ДНК рослин з молекулярними маркерами до локусів Xrems 1303, -secalin, Gli-B1, Xgwm18, Xgwm550. Поставлена задача вирішується тим, що дослідження проводять методом полімеразної ланцюгової реакції з використанням набору молекулярних маркерів для ампліфікації ДНК рослин популяції F4 (та наступних популяцій, що отримано від схрещування донору модифікованої 1RS.1BL транслокації та сорту м’якої пшениці без житньої транслокації), електрофорезу ампліфікованих фрагментів ДНК у 10 % денатуруючому поліакриламідному гелі (ПААГ) з 8 М сечовиною та ідентифікації житньої хромосоми 1RS за результатами електрофорезу. Відмінними від прототипу ознаками в корисній моделі, що заявляється, є: - можливість проводити детекцію у вихідному матеріалі модифікованої житньої транслокації 1RS.1BL на будь-якій стадії розвитку рослини; - можливість виявити рекомбінантні рослини, які виникають при схрещуванні рослини-донора модифікованої транслокації 1RS.1BL із рослиною сорта м'якої пшениці, який не несе даної транслокації; - спосіб підготовки зразків до аналізу. Запропонований спосіб в генетико-селекційних дослідженнях може бути використаний для: - скорочення строків селекційного процесу за рахунок прискорення темпів відбору рослин із модифікованою житньою 1RS.1BL транслокацією; 4 - контроля модифікованої житньої 1RS.1BL транслокації в генетичному матеріалі; - створення нових форм м'якої пшениці з модифікованою транслокацією 1RS.1BL та з будьяким із відомих пшеничних алелів локусу Gli-B1 Спосіб здійснюється таким чином. 1. Виділення ДНК. Сегмент листя розміром 1 см гомогенізують в 1,5 мл пробірці типу "епендорф" (далі епендорф) скляним пестиком до повної мацерації тканин з додаванням 0,5 мл лізуючого буферу (20 мМ Na3EDTA, 100 мМ Трис-НСl рН 8,0 при 25 °С, 1,4 М NaCl, 2,0 % СТАВ). Лізат інкубують 60 хв. при 60 °С. Додають рівний об'єм (0,5 мл) суміші хлороформ-ізоамілового спирту (24:1 за об'ємом), перемішують на вортексі до утворення білої емульсії. Отриману суміш центрифугують 5 хв. при 14000 об/хв. на мікрофузі "Eppendorf 5415". Після центрифугування відбирають водну фазу і переносять в новий епендорф, додають рівний об'єм ізопропилового спирту, витримують при кімнатній температурі протягом 10 хв. Суміш перемішують і осаджають ДНК центрифугуванням при 14000 об/хв. протягом 4 хв. на мікрофузі "Eppendorf 5415". Надосадну рідину зливають, осад промивають двічі 1 мл 70 % етанола - центрифугують при 14000 об/хв. протягом 2 хв. на мікрофузі "Eppendorf 5415", надосадну рідину зливають. Підсушують осад при кімнатній температурі 15-20 хв. і розчиняють в 100 мкл ТЕ-буферу (10 мМ Трис-НСl рН 8,0 (25 °С); 1 мМ Na3EDTA pH 8,0) [11]. Концентрацію виділеної ДНК визначають на флуорометрі "ТКО 100 Dedicated mini Fluorometer" ("Hoefer Scientific Instruments", США) у 1xTNE буфері (3 M NaCl, 50 мМ Na2НРO4) у присутності флуоресцентного барвника Hoechst 332583. 2. Проведення полімеразної ланцюгової реакції (ПЛР). ПЛР проводять із використанням праймерів, вказаних в табл. 1, та згідно режиму ампліфікації (табл. 2). Таблиця 1 Локуси, за якими пропонується детектувати модифіковану житню 1RS.1BL транслокацію, або 1RS.1BL транслокацію без модифікації Локус Xrems1303 -secalin GliB1.1 GliB1.2 Xgwm18 Xgwm550 Хромосомна локалізація 1RS 1BS Послідовність праймерів (5'-3') прямого зворотного tagcaccactcgctctctca tttcccatcagaaaaatcgc ccttcctcatctttgtcctc gctctggtctctggggttgt tgatctggccacaaaggga cattggccaccaattcctgt tgatctggccacaaagggc cattggccaccaattcctgt tggcgccatgattgcattatcttc ggttgctgaagaaccttatttagg cccacaagaacctttgaaga cattgtgtgtgcaaggcac ПЛР проводять у 0,75 мкл епендорфах з використанням ампліфікатора "Терцик" ("ДНКтехнологія", Росія). Реакційна суміш для проведення ПЛР об'ємом 25 мкл містить: 2,5 мкл 10хПЛР-буферу (50 мМ КСl, 20 мМ Трис-НСl рН 8,4 (25 °С), 0,01 % Tween-20), 40 нг ДНК, 2 мМ MgCl2, 10 pmol кожного праймера (прямого та зво ротного) (табл. 1), по 0,2 мМ кожного dАTP, dСTP, dТTP, dGTP, та 1 одиниці ДНК-полімерази Taq або Thermostar ("Yorkshire Bioscience Ltd", UK). ДНКполімеразу Taq-Thermostar використовують для ПЛР-аналізу Gli-B1 локусу. Поверх реакційного розчину нашаровують 20 мкл мінеральної олії. 5 59084 6 Таблиця 2 Температурний режим етапів ампліфікації № п/п 1 1. 2. 3. 4. 5. 6. 7. 8. 9. 10. 11. Температура, °С Час, хв. 2 3 для пари праймерів до локусу Xrems1303 95 3 95 0,45 1 68 1 72 95 0,45 1 66 1 72 95 0,45 1 64 1 72 95 0,45 5 62 1 72 95 0,45 1 60 1 72 95 0,45 1 58 1 72 95 0,45 1 56 1 72 95 0,45 1 54 1 72 95 0,45 1 52 1 72 95 0,45 1 50 1 72 Кількість циклів 4 1 1 1 1 1 1 1 1 1 1 1 7 59084 8 Продовження таблиці 2 12. 1. 2. 0,45 0,30 72 13. 95 47 1 72 10 для праймерів до локусу -secalin 94 3 94 0,45 65 1 72 3. 1. 2 3. 1. 2. 3. 1,5 72 7 для алель-специфічних праймерів до локусу Gli-B1 95 15 95 0,30 56 0,30 72 1 72 5 для праймерів до локусу Xgwm18, Xgwm550 94 3 94 1 55 1 72 2 72 3. Електрофоретичний розподіл продуктів ампліфікації. Продукти ампліфікації фракціонують електрофорезом у 10 % денатуруючому поліакриламідному гелі з 8 М сечовиною розміром 2020 cм ("Hoefer Scientific Instruments", США) в 1xTBE буфері (89,0 мМ Трис, 89,0 мМ борна кислота, 2,0 мМ EDTA) за постійної напруги 550 В 2-3 год., залежно від довжини фрагментів. 4, Візуалізація продуктів ампліфікації. Фрагменти ДНК у ПААГ фарбують сріблом відповідно до методики: поліакриламідний гель занурюють на 510 хв. у 10 % етанол, потім переносять у 1 % HNO3 на 3 хв. Гель двічі промивають дистильованою водою. Витримують протягом 20 хв. у 0,012 М AgNО3 у темряві. Двічі промивають дистильованою водою. Потім заливають гель відновлюючим розчином (0,28 М Na2СО3 (безводний), 0,019 % формаміду), інкубують, перемішують до появи забарвлення фрагментів ампліфікації. Промивають двічі дистильованою водою і переносять у 10 % оцтову кислоту на 2 хв. Потім промивають Н2О. Гель зберігають між двома шарами прозорої поліетиленової плівки. 10 27 1 1 35 1 1 38 1 1 45 1 5. Документування результатів аналізу. Для документування результатів аналізу (табл. 3) та оцінки довжини продуктів ампліфікації використовують систему документації та аналізу гелів "Image Master VDS" ("Amersham Pharmacia Biotech", Австрія) згідно з інструкцією користувача обладнання. Калібрування молекулярної маси здійснюють з використанням стандартів молекулярної маси (pGem, pUC19/MspI), використовуючи програму "Image Master 1D Elite". При аналізі продуктів ПЛР, що отримані при ампліфікації ДНК вивчаємих зразків рослин з праймерами до маркуємих локусів можливо спостерігати різні варіанти результатів ПЛР (табл.. 3). Аналіз таких результатів дає відповідь - присутня звичайна 1В хромосома чи модифікована 1RS.1BL транслокація, або рекомбінантна модифікована транслокація чи звичайне (не модифіковане) 1RS плече хромосоми жита. Варіанти генотипів рослин, що можуть бути отримані від схрещування донорів транслокацій 1RS.1BL модифікованої або не модифікованої із сортом м’якої пшениці зі звичайною 1В хромосомою наведені у табл. 3 9 59084 10 Таблиця 3 Варіанти генотипів рослин з модифікованою 1RS.1BL транс локацією або з 1RS.1BL транслокацією без модифікації за результатами ПЛР-аналізу в селекційному матеріалі Присутність/ відсутність Присутність/відсутність фрагментів ампліфікації з ДНК рослин за даними локуса1RS.1BL модифікованої ми транслокації Gli-Bl (Алелі Xrems1303 Xgwm550 Xgwm18 GliB1.1, GliB 1.2 -secalin або ін.) 1В хромосома пшениці + + + Модифікована 1RS.1BL + + транслокація Модифікована рекомбінантна 1RS.1BL тран+ + + +* слокація Звичайна 1RS.1BL тра+ + нслокація (контроль) Примітки: * - будь-якої з варіантів алелів Gli-B1 локусу, що відрізняється від алеля вихідної форми донора 1RS.1BL модифікованої транслокації. 6. Приклад застосування способу детекції модифікованої житньої транслокації 1RS.1BL в селекційних формах м'якої пшениці. З рослин популяції F4, яка нараховує 61 індивідуальну рослину (матеріал створений д.б.н. О. І. Рибалкою, у відділі генетичних основ селекції Селекційно-генетичного інституту - Національного центру насіннєзнавства та сортовивчення (СП-НЦНС) НААН України) була виділена ДНК. Така популяція отримана від схрещування лінії Pavon MA1 (донор модифікованої 1RS.1BL транслокації) із сортом озимої м'якої пшениці Куяльник. Також у дослідження включено лінії Hostianum 242/97-2-В та Б-16, що мають звичайну 1RS.1BL транслокацю. ПЛР-аналіз виділеної ДНК рослин проводили з праймерами, які представлено в табл. 1. На доріжках гелю після електрофорезу чітко детектовано продукти ПЛР за локусом Xrems1303, що виявляють фрагмент ампліфікації розміром 290 п.н. у зразків із житнім 1RS плечем та не виявили фрагмент ампліфікації у зразків із пшеничним 1BS плечем. З парою праймерів до локусу -secalin - виявляли фрагмент ампліфікації ДНК з молекулярною масою 400 п.н. у зразків із житнім секаліновим локусом (фіг. 1) (такі фрагменти ампліфікації можна детектувати у рослин із звичайною житньою транслокацією). Водночас, дослідження локуса Gli-B1 (фіг. 2) за допомогою алельспецифічної ПЛР показало присутність фрагмента ампліфікації розміром 369 п.н. (тобто алель GliB1.1) або 397 п.н. (алель GliB1.2) у рослин із пшеничною хромосомою 1ВS або із модифікованою 1RS.1BL, транслокацією, у яких локус Gli-B1 замінив секаліновий локус Sec-1, і відсутність продуктів ампліфікації у рослин, які несуть звичайну 1RS.1BL транслокацію. З парою праймерів до локусу Xgwm18 виявлено фрагменти ампліфікації ДНК, розмір яких може варіювати, але у даному випадку становив 186 п.н. - це відповідає апелю, характерному для 1ВS пшениці. У той же час, відсутність фрагментів ампліфікації за Xgwm18 локусом характерна для зразків із житнім 1RS плечем (фіг. 3). Результати дослідження популяції F4, отриманої від схрещування лінії Pavon MA1 із сортом м'якої пшениці Куяльник, представлено в табл. 4. Таблиця 4 Результати ПЛР-аналізу рослин популяції F4 (Куяльник х Pavon MA1 F4: Куяльник х Pavon MA1 Алелі маркерних локусів в п.н. F4: Куяльник х Pavon MA1 Алелі маркерних локусів в п.н. Gli-B1 GliB1 (1BS) № індиві№ інд. Xrems1303 Xgwm18 Xgwm550 (1BS) Xrems1303 Xgwm18 Xgwm550 дуальних рослин (1RS) (1BS) (1BS) (1RS) (1BS) (1BS) Gli Gli Gli Gli рослин B1.1 B1.2 B1.1 B1.2 1 2 3 4 5 6 1 2 3 4 5 6 1/1 290 - 397 8/1 290 397 1/2 290 - 397 8/2 290 369 1/3 290 - 397 8/3 290 195 369 1/4 290 - 397 8/4 290 397 Продовження таблиці 4 11 2/1 2/2 2/3 2/4 2/5 3/1 3/2 3/3 3/4 3/5 4/1 4/2 4/3 4/4 4/5 5/1 5/2 5/3 5/4 5/5 6/1 6/2 6/3 6/4 6/5 7/1 7/2 7/3 7/4 7/5 290 290 290 290 290 290 290 290 290 290 59084 186 186 186 186 186 186 186 186 186 186 186 186 186 186 186 186 186 186 186 186 195 195 195 195 195 195 195 195 195 195 195 195 195 195 195 195 195 195 195 195 369 369 369 369 369 369 369 369 369 369 369 369 369 369 369 369 369 369 369 397 397 397 397 397 397 397 397 397 397 397 9/1 9/2 9/3 9/4 9/5 10/1 10/2 10/3 10/4 10/5 11/1 11/2 11/3 11/4 П/5 12/1 12/2 12/3 12/4 13/1 13/2 13/3 13/4 12 290 290 290 290 290 290 290 290 290 290 290 290 290 290 290 186 186 186 186 186 186 186 186 195 195 195 195 195 195 195 195 369 369 369 369 369 369 369 369 369 397 397 397 397 397 397 397 397 397 397 397 397 397 397 Примітки: « - » - не детектовано продуктів ампліфікації ДНК За результатами ПЛР-аналізу у дослідженому матеріалі ідентифіковано рослини з модифікованою 1RS.1BL транслокацією (аналогічною транслокації в батьківській лінії Pavon MA1) під номерами: 1/1 - 1/4, 4/1 - 4/5, 5/1 - 5/5, 8/1, 8/4, 9/1 - 9/5, 10/1, 10/2, 10/4, 10/5, 11/1 - 11/5. Визначено рослини, що мають звичайну 1В хромосому пшениці: 2/1 - 2/5, 3/1 - 3/5, 6/1 - 6/5, 7/1 - 7/5, 12/1 - 12/4, 13/1 - 13/4. Також ідентифіковано чотири рослини, в яких відбулись рекомбінаційні події у короткому 1RS плечі модифікованої житньо-пшеничної транслокації та плечі 1BS пшениці. Так, рослина під номером 3/4 має звичайну 1В хромосому, але в результаті рекомбінації в локусі Gli-B1 успадкувала батьківський алель GliB1.2 (характерний для лінії Pavon MA1). Рослини під номерами 8/2 та 10/3 несуть модифіковану транслокацію, але, в результаті рекомбінації в їх генотипах, алель GliB1.2, характерний для вихідної батьківської форми - лінії Pavon MA1, замістив алель GliB1.1, присутній у сорту Куяльник. У рослини під номером 8/3 рекомбінація зачепила більш широкий регіон. Ця рослина має модифіковану житньо-пшеничну транслокацію 1RS.1BL, а вихідний алель GliB1.2, характерний для батьківської форми - лінії Pavon MA1, заміщений алелем GliB1.1 сорту Куяльник та за локусом Xgwm550 детектується алель розміром 195 п.н., що також детектується у сорті Куяльник. Нами пропонується метод використання ПЛР із системою ДНК-маркерів (Xrems1303, -secalin, GliB1.1, GliB1.2, Xgwm18, Xgwm550), яка дозволяє детектувати присутність модифікованої житньої 1RS.1BL транслокації, а також 1RS.1BL транслокації без модифікації у досліджуваному генетичному матеріалі, а саме: - Xrems1303 - свідчить про наявність житньої хромосоми 1RS; - -secalin - показує присутність локусу Sec-1 на хромосомі 1RS (у випадку детекції звичайної транслокації 1RS.1BL); - якщо рослина має модифіковану 1RS.1BL транслокацію, з вилученим Sec-1 локусом, при ПЛР-аналізі з парою праймерів до -secalin локусу детекція фрагменту ампліфікації з молекулярною масою 400 п.н. не відбувається, водночас, у такої рослини детектуються специфічні для жита фрагменти за локусом Xrems1303 та один із алелів локусу Gli-B1 а також фрагмент ампліфікації за локусом Xgwm18, що є характерним для проксимального регіону 1BS хромосоми; - алель-специфічні праймери GliB1.1 і GliB1.2 визначають алелі Gli-B1 локусу на 1BS хромосомі, якщо така присутня у рослин або рослина має фрагмент хромосоми з цим локусом; - молекулярні маркери Xgwm18 та Xgwm550 детектують присутність фрагментів ампліфікації, 13 що є характерними для проксимального і дистального регіону 1BS хромосоми пшениці, відповідно. Таким чином, ПЛР-аналізування з системою запропонованих маркерів до локусів Xrems1303, -secalin і Gli-B1, Xgwm18, Xgwm550 може застосовуватись в селекційно-генетичних дослідженнях для детекції модифікованої житньої 1RS.1BL транслокації або 1RS.1BL транслокації без модифікації в селекційних формах м'якої пшениці. 7. Перелік фігур креслення Фіг. 1. Електрофореграма розподілу продуктів ампліфікації ДНК за -secalin локусом у 10 % денатуруючому ПААГ. М - маркер молекулярної маси pUC19/MspI, 1 - лінія Б-16, 2 - лінія Н242/97-2-В, 3 лінія Pavon MAI, 4 - сорт Куяльник Фіг. 2. Електрофореграма розподілу продуктів ампліфікації ДНК популяції F4 і батьківських форм у 10 % денатуруючому ПААГ, що отримані з алель-специфічними праймерами: a. GliB1.1; б. GliB1.2. M - маркер молекулярної маси pUC19/MspI, 4 - сорт Куяльник, 3 - лінія Pavon MAI, 5 - 12 - індивідуальні рослини популяції F4 (2/1, 3/1, 6/1, 1/1, 7/1, 8/1, 9/1, 10/1, відповідно), 1 - лінія Б16, 2 - лінія Н242/97-2-В Фіг. 3. Електрофореграма розподілу продуктів ампліфікації ДНК популяції F4 і батьківських форм за Xgwm18 локусом у 10 % денатуруючому ПААГ. М - маркер молекулярної маси pUC19/MspI, 3 лінія Pavon MAI, 4 - сорт Куяльник; 5, 13, 6, 14, 7, 15, 9, 16, 17, 8, 18, 10, 11, 12, 19 -індивідуальні рослини популяції F4 (2/1, 2/2, 3/1, 4/1, 6/1, 6/2, 7/1, 12/1, 13/1, 1/1, 5/1, 8/1, 9/1,10/1, 11/1, відповідно) Джерела інформації: 1. Rabinovich S. V. Importance of wheat-rye translocations for breeding modern cultivars of Triticum aestivum L. // Euphytica. - 1998. - V. 100. - P. 323-340. 2. Mclntosh R. A., Hart G. E., Devos K. M., Gale M. D., Rogers W. J. Catalogue of gene symbols for wheat // Proc. 9th Intern. Wheat Genetics Symp. 1998, Sascatoon, Sascatchewan, Canada, V. 5. - P. 123-145. 59084 14 3. Zeller F. J., Gunzel G., Fischbeck G., Gersterncorn P., Weipert D. Verfhderung der Backeigenschaften der Weizen-Roggen ChromosomenTranslocation 1B/1R. - Getreide Mchl. Brot., 1982. V. 36. - P. 141-143. 4. Lukaszewski A. Manipulation of the 1RS.1BL translocation in wheat by induced homoelogus recombination // Crop. Sci. - 2000. - V. 40. - P. 216-225. 5. Рибалка О. І., Литвиненко М. А. Використання в селекції пшениці транслокації 1RS.1BL // Вісник аграрної науки. - К., 2009. - № 4. - С. 36-40). 6. Villareal R. L., Rajaram S., MuJeeb-Kazi A., Del-Того E. The effect of chromosome 1B/1R translocation on the yield potential of certain spring wheats (Triticum aestivum L.) // Plant Breed. - 1991. - V. 106. - P. 77-81. 7. Козуб Н. А., Созинов И. А., Колючий В. Т., Созинов А. А. Сорта мягкой пшеницы украинской селекции с ржаными 1BL/1RS и 1AL/1RS транслокациями // Фактори експериментальної еволюції організмів: 36. наук, пр. / Укр. т-во генетиків і селекціонерів ім. М. І. Вавилова / За ред. М. В. Роїка. К.: Логос, 2006. - Т. 3. - С. 216-220. 8. Созинов А. А., Попереля Ф. А. Электрофорез глиадина как метод идентификации пшениц, у которых 1B-хромосома полностью или частично замещена 1R-хромосомой ржи // Докл. ВАСХНИЛ. - 1977. - № 2. - С. 2-4. 9. Sozinov A. A., Poperela F. A. Genetic classification of prolamines and its use for plant breeding // Ann. Technol. Agric. - 1980. - 29. - P. 229-245. 10. Сударчук Л. В., Чеботар С. В., Рибалко О. І., Сиволап Ю. М. Детекція модифікованої транслокації 1RS.1BL за допомогою молекулярних маркерів у селекційному матеріалі м'якої пшениці // Вісник ОНУ. - Т. 15, Вип. 6. - 2010. - С. 39-48. 11. Использование ПЦР-анализа в генетикоселекционных исследованиях / Научнометодическое руководство. Под ред. Сиволапа Ю. М. - К.: Аграрна наука, 1998. - С. 34-40. 15 Комп’ютерна верстка Г. Паяльніков 59084 Підписне 16 Тираж 24 прим. Міністерство освіти і науки України Державний департамент інтелектуальної власності, вул. Урицького, 45, м. Київ, МСП, 03680, Україна ДП “Український інститут промислової власності”, вул. Глазунова, 1, м. Київ – 42, 01601
ДивитисяДодаткова інформація
Назва патенту англійськоюMethod for detecting modified ryetranslocation 1rs.1bl in breeding forms of soft wheat
Автори англійськоюSyvolap Yurii Mykhailovych, Chebotar Sabina Vitaliivna, Sudarchuk Liudmyla Valeriivna
Назва патенту російськоюСпособ детекции модифицированной ржаной транслокации 1rs.1bl в селекционных формах мягкой пшеницы
Автори російськоюСиволап Юрий Михайлович, Чеботар Сабина Витальевна, Сударчук Людмила Валерьевна
МПК / Мітки
МПК: A01H 1/06
Мітки: формах, детекції, житньої, пшениці, м'якої, селекційних, модифікованої, спосіб, 1rs.1bl, транслокації
Код посилання
<a href="https://ua.patents.su/8-59084-sposib-detekci-modifikovano-zhitno-translokaci-1rs1bl-v-selekcijjnikh-formakh-myako-pshenici.html" target="_blank" rel="follow" title="База патентів України">Спосіб детекції модифікованої житньої транслокації 1rs.1bl в селекційних формах м’якої пшениці</a>
Попередній патент: Високоефективний акумулятор постійного струму
Наступний патент: Спосіб прогнозування загрози переривання вагітності в 1 триместрі
Випадковий патент: Пристрій для відбору проб матеріалу, що транспортується на конвеєрі