Спосіб одночасного виявлення мікобактерій туберкульозного комплексу та ідентифікації мутацій днк мікобактерій, що призводять до стійкості мікроорганізмів до рифампіцину та ізоніазиду, на біологічних мікрочипах,
Номер патенту: 92720
Опубліковано: 10.12.2010
Автори: Засєдатєлєв Алєксандр Сєргєєвіч, Соболєв Алєксандр Юрьєвіч, Міхайловіч Владімір Міхайловіч, Лапа Сєргєй Анатольєвіч, Грядунов Дмітрій Алєксандровіч, Мірзабєков Андрєй Дарьєвіч
Формула / Реферат
1. Спосіб одночасного виявлення мікобактерій туберкульозного комплексу та ідентифікації мутацій ДНК мікобактерій, які приводять до стійкості мікроорганізмів до рифампіцину й ізоніазиду, у клінічних зразках, який включає:
(A) - мультиплексну ампліфікацію фрагментів генів rроВ, katG, inhA, аhрС, мобільного елемента IS6110 із використанням набору пар специфічних праймерів для першої стадії ПЦР; послідовності яких представлені SEQ ID NO: 70, 71, 74, 75, 77, 78, 80, 81, 83, 84;
(Б) - мультиплексну ампліфікацію фрагментів генів rроВ, katG, inhA, ahpC, мобільного елемента IS6110 із використанням як матриці продукту ПЦР, отриманого на стадії (А), і набору пар специфічних праймерів для другої стадії ПЦР, послідовності яких представлені SEQ ID NO: 72, 73, 74, 76, 77, 79, 80, 82, 83, 85, причому один праймер у кожній парі праймерів є флуоресцентно міченим, з одержанням переважно одноланцюгових флуоресцентно мічених фрагментів;
(B) - забезпечення біочипа для одночасного виявлення мікобактерій туберкульозного комплексу й ідентифікації мутацій, які приводять до стійкості до рифампіцину й ізоніазиду, що являє собою підкладку з гелевими елементами, у кожному з яких іммобілізований унікальний олігонуклеотидний зонд, що має послідовність, вибрану із групи, яка включає послідовності: а) відповідні послідовності фрагмента гена rроВ дикого типу; б) відповідні послідовності фрагмента мутантного варіанта гена rроВ, що приводить до стійкості мікроорганізмів до рифампіцину; в) комплементарні до послідовностей, охарактеризованих в а) і б); г) відповідні послідовності фрагмента гена katG дикого типу; д) відповідні послідовності фрагмента мутантного варіанта гена katG, що приводить до стійкості мікроорганізмів до ізоніазиду; є) комплементарні до послідовностей, охарактеризованих у г) і д); ж) відповідні послідовності фрагмента гена inhA дикого типу; з) відповідні послідовності фрагмента мутантного варіанта гена inhA, що приводить до стійкості мікроорганізмів до ізоніазиду; і) комплементарні до послідовностей, охарактеризованих у ж) і з); к) відповідні послідовності фрагмента гена ahpC дикого типу; л) відповідні послідовності фрагмента мутантного варіанта гена ahpC, що приводить до стійкості мікроорганізмів до ізоніазиду; м) комплементарні до послідовностей, охарактеризованих у к) і л); н) відповідні послідовності фрагмента мобільного елемента IS6110; о) комплементарні до послідовностей, охарактеризованих у н);
(Г) - гібридизацію ампліфікованих мічених продуктів, отриманих на стадії (Б), на біочипі в умовах, які забезпечують розрізнення в один нуклеотид між довершеними і недовершеними дуплексами, які утворюються в результаті гібридизації;
(Д) - реєстрацію й інтерпретацію результатів гібридизації.
2. Спосіб за п. 1, який відрізняється тим, що на другій стадії мультиплексної ПЦР (Б) з метою одержання переважно одноланцюгових флуоресцентно мічених фрагментів для всіх використаних пар праймерів флуоресцентно мічений праймер використовують у молярному надлишку відносно іншого праймера, який входить до тієї ж пари.
3. Спосіб за п. 1, який відрізняється тим, що ампліфікацію фрагментів генів і мобільного елемента IS6110 проводять, використовуючи безпосередньо матеріал клінічного зразка (мокротиння, ексудат, змивки, бронхоальвеолярний лаваж) або попередньо вирощену культуру мікроорганізмів.
4. Спосіб за п. 1, який відрізняється тим, що з метою забезпечення розрізнення в один нуклеотид між довершеними і недовершеними дуплексами, які утворюються в результаті гібридизації, використовують гібридизаційний буфер, який дозволяє проводити гібридизацію в розширеному інтервалі температур.
5. Спосіб за п. 1, який відрізняється тим, що реєстрацію результатів на стадії (Д) проводять за допомогою портативного аналізатора флуоресценції й програмного забезпечення, що дозволяє використовувати програмну обробку інтенсивностей сигналів з наступною інтерпретацією результатів.
6. Спосіб за п. 1, який відрізняється тим, що інтерпретацію зареєстрованих результатів на стадії (Д) виконують шляхом порівняння інтенсивності флуоресценції сигналів комірок, у яких утворилися довершені й недовершені гібридизаційні дуплекси.
7. Спосіб за п. 1, який відрізняється тим, що інтерпретовані результати можуть бути застосовані для підтвердження клінічного діагнозу - туберкульоз, і цілей епідеміологічного генотипування, використовуючи як маркер наявність тієї або іншої мутації.
8. Набір специфічних пар праймерів для здійснення способу одночасного виявлення мікобактерій туберкульозного комплексу й ідентифікації мутацій ДНК мікобактерій, які приводять до стійкості їх до рифампіцину й ізоніазиду, у клінічних зразках за п. 1, причому послідовності праймерів представлені SEQ ID NO: 70-85.
9. Біочип, використовуваний у способі одночасного виявлення мікобактерій туберкульозного комплексу й ідентифікації мутацій ДНК мікобактерій, які приводять до стійкості до рифампіцину й ізоніазиду, у клінічних зразках за п. 1, який є підкладкою з гелевими елементами, у кожному з яких іммобілізований унікальний олігонуклеотидний зонд, причому послідовності зондів представлені SEQ ID NO: 1-69.
10. Набір олігонуклеотидних зондів, використовуваний для одержання біочипа, що його використовують в способі одночасного виявлення мікобактерій туберкульозного комплексу й ідентифікації мутацій ДНК мікобактерій, які приводять до стійкості до рифампіцину й ізоніазиду, у клінічних зразках за п. 1, причому зонди мають послідовності SEQ ID NO: 1-69.
Текст
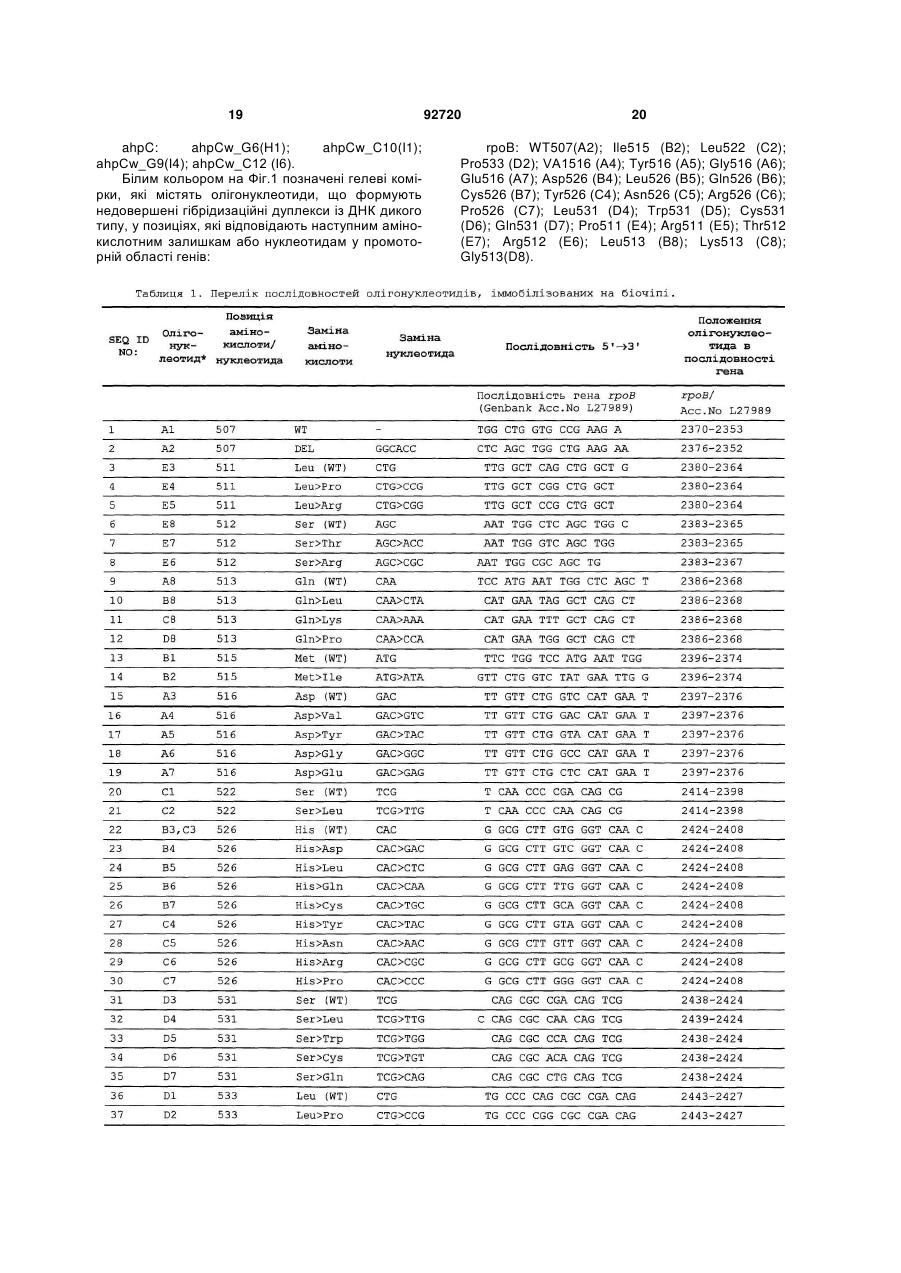
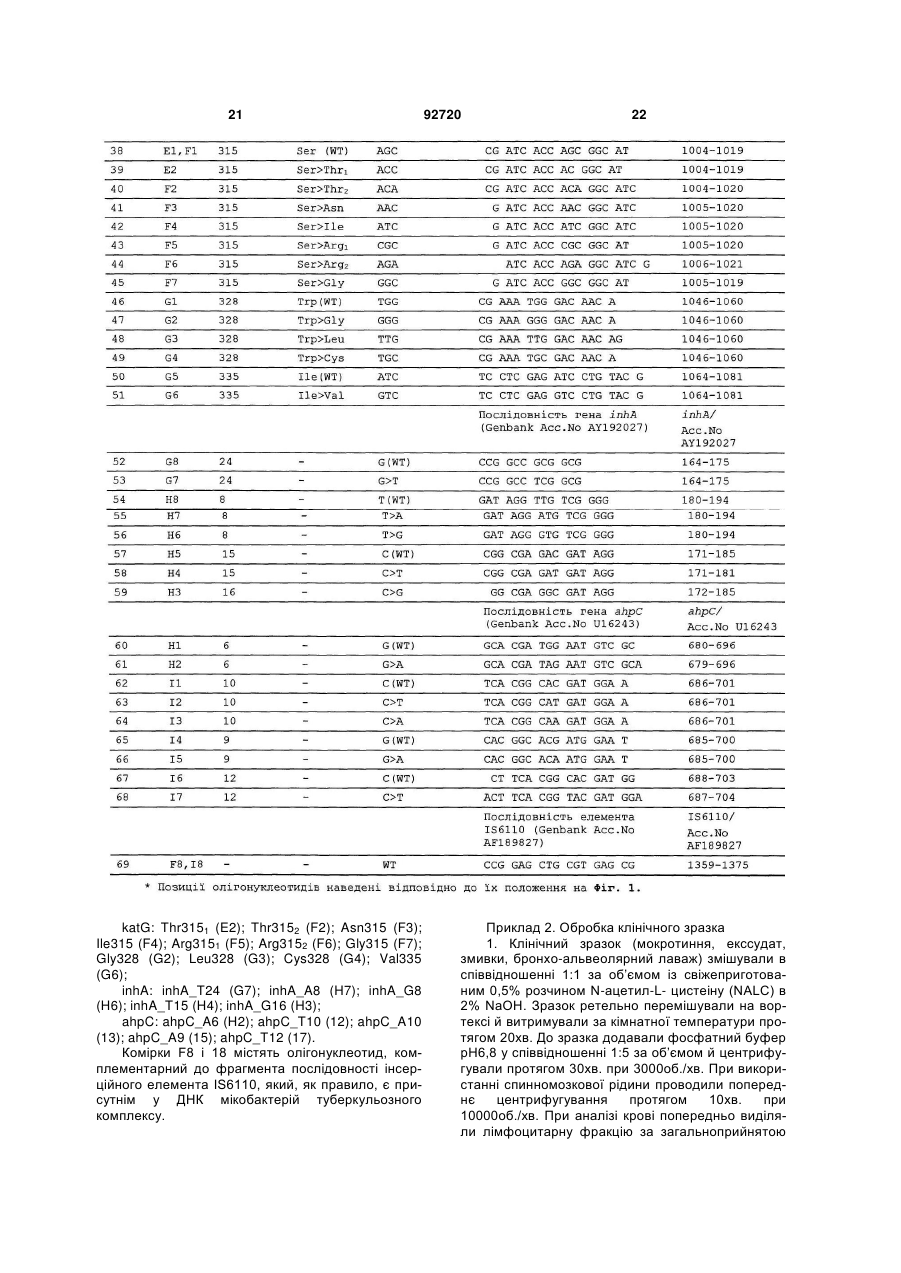
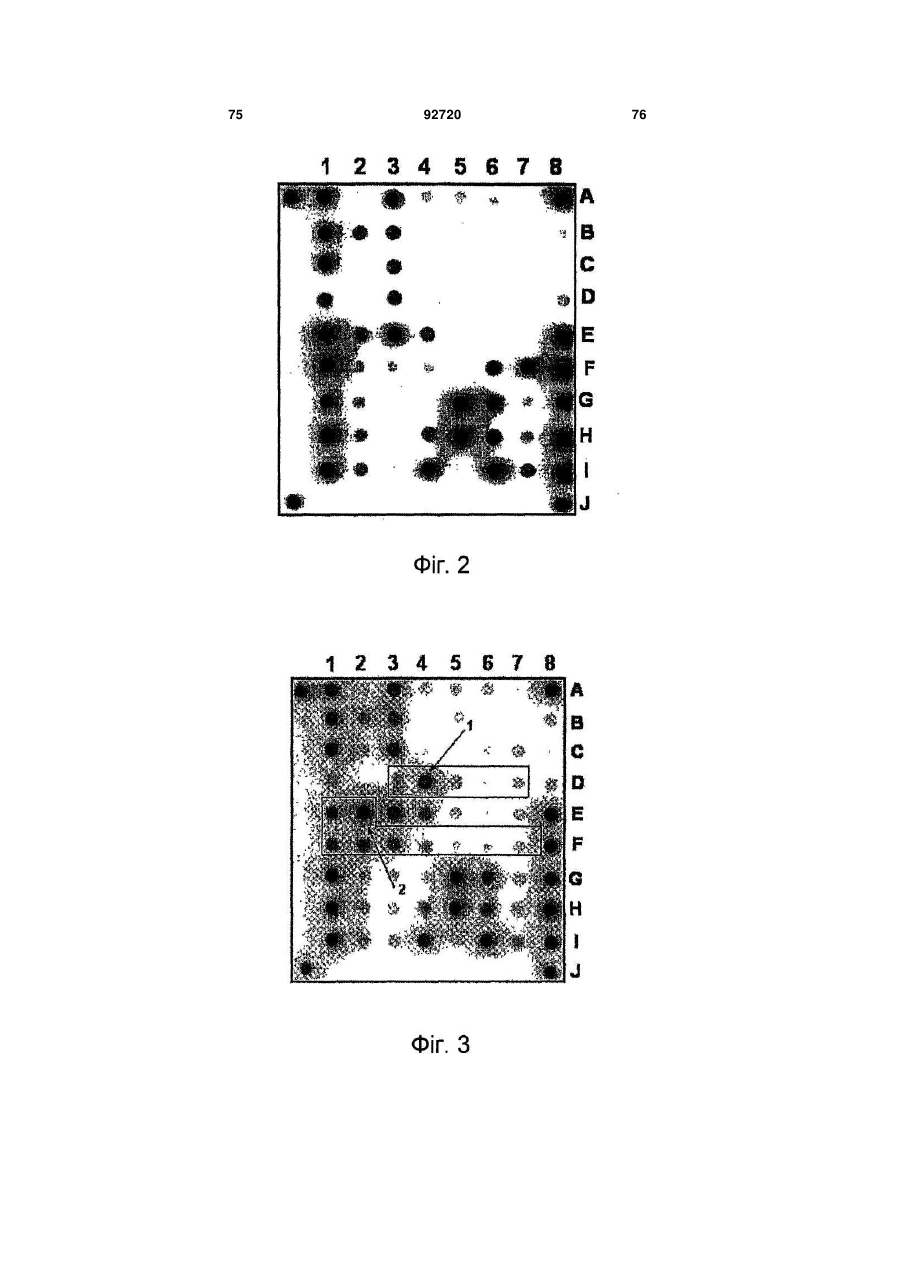
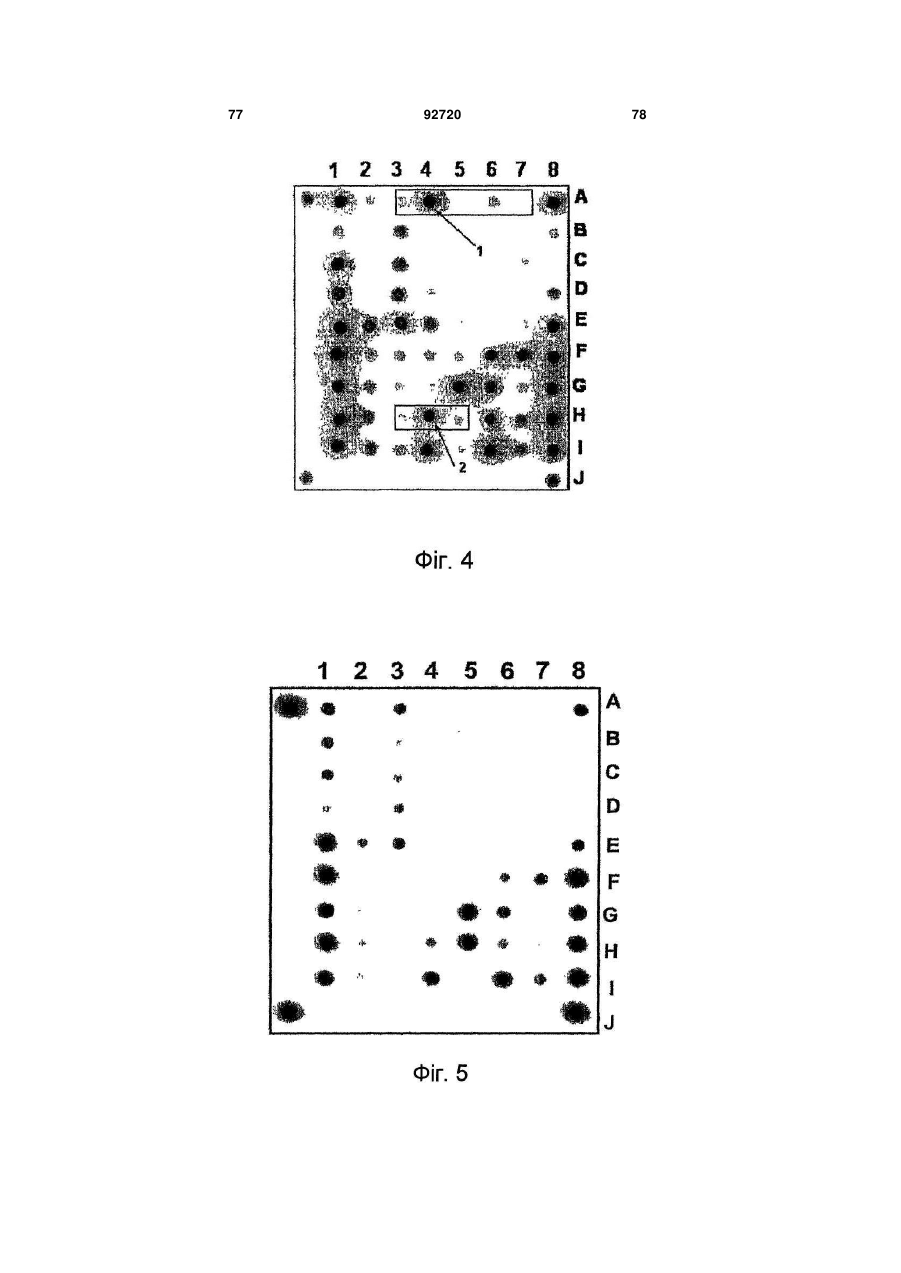
1. Спосіб одночасного виявлення мікобактерій туберкульозного комплексу та ідентифікації мутацій ДНК мікобактерій, які приводять до стійкості мікроорганізмів до рифампіцину й ізоніазиду, у клінічних зразках, який включає: (A) - мультиплексну ампліфікацію фрагментів генів rроВ, katG, inhA, аhрС, мобільного елемента IS6110 із використанням набору пар специфічних праймерів для першої стадії ПЦР; послідовності яких представлені SEQ ID NO: 70, 71, 74, 75, 77, 78, 80, 81, 83, 84; (Б) - мультиплексну ампліфікацію фрагментів генів rроВ, katG, inhA, ahpC, мобільного елемента IS6110 із використанням як матриці продукту ПЦР, отриманого на стадії (А), і набору пар специфічних праймерів для другої стадії ПЦР, послідовності яких представлені SEQ ID NO: 72, 73, 74, 76, 77, 79, 80, 82, 83, 85, причому один праймер у кожній парі праймерів є флуоресцентно міченим, з одержанням переважно одноланцюгових флуоресцентно мічених фрагментів; (B) - забезпечення біочипа для одночасного виявлення мікобактерій туберкульозного комплексу й ідентифікації мутацій, які приводять до стійкості до рифампіцину й ізоніазиду, що являє собою підкладку з гелевими елементами, у кожному з яких іммобілізований унікальний олігонуклеотидний зонд, що має послідовність, вибрану із групи, яка включає послідовності: а) відповідні послідовності фрагмента гена rроВ дикого типу; б) відповідні послідовності фрагмента мутантного варіанта гена rроВ, що приводить до стійкості мікроорганізмів до рифампіцину; в) комплементарні до послідовностей, охарактеризованих в а) і б); г) відповідні послідовності фрагмента гена katG дикого типу; д) відповідні послідовності фрагмента мутантного варіанта гена katG, що приводить до стійкості мікроорганізмів до ізоніазиду; є) комплементарні до UA (21) a200613828 (22) 26.12.2006 (24) 10.12.2010 (31) 2005140679 (32) 26.12.2005 (33) RU (46) 10.12.2010, Бюл.№ 23, 2010 р. (72) ЗАСЄДАТЄЛЄВ АЛЄКСАНДР СЄРГЄЄВІЧ, RU, СОБОЛЄВ АЛЄКСАНДР ЮРЬЄВІЧ, RU, ГРЯДУНОВ ДМІТРІЙ АЛЄКСАНДРОВІЧ, RU, ЛАПА СЄРГЄЙ АНАТОЛЬЄВІЧ, RU, МІХАЙЛОВІЧ ВЛАДІМІР МІХАЙЛОВІЧ, RU, МІРЗАБЄКОВ АНДРЄЙ ДАРЬЄВІЧ, RU (73) УЧРЄЖДЄНІЄ РОССІЙСКОЙ АКАДЄМІІ НАУК ІНСТІТУТ МОЛЄКУЛЯРНОЙ БІОЛОГІЇ ІМ. В.А.ЕНГЕЛЬГАРДТА РАН (ІМБ РАН), RU (56) WO A1 0036142, 22.06.2000. WO A1 0222872, 21.03.2002. US B1 6242584, 05.06.2001. US A 5643723, 01.07.1997. KR A 20050009052, 24.01.2005. KR A 20030008620, 29.01.2003. KR A 20000000849, 15.01.2000. JP A 2004154125, 03.06.2004. JP A 2005087132, 07.04.2005. CN A 1341752, 27.03.2002. RU C2 2204608, 20.05.2003. RU C1 2200323, 10.03.2003. RU C1 2175015, 20.20.2001. RU C 2000331, 07.09.1993. Grace Lin S.Y. :”Rapid detection of isoniazid and rifampin resistance mutations in Mycobacterium tuberculosis complex from cultures or smear-positive sputa by use of molecular beacons”, Journal of clinical microbiology, Sept. 2004, Vol. 42, No. 9, p. 4204-4208. Mercedes Marin et al.:”Rapid direct detection of multiple rifampin and isoniazid resistance mutations in Mycobacteriu tuberculosis in respiratory samples by real-time PCR”, Antimicrobial agents and chemotherapy, Nov. 2004, Vol. 48, No. 11, p. 42934300. 2 (19) 1 3 92720 4 послідовностей, охарактеризованих у г) і д); ж) відповідні послідовності фрагмента гена inhA дикого типу; з) відповідні послідовності фрагмента мутантного варіанта гена inhA, що приводить до стійкості мікроорганізмів до ізоніазиду; і) комплементарні до послідовностей, охарактеризованих у ж) і з); к) відповідні послідовності фрагмента гена ahpC дикого типу; л) відповідні послідовності фрагмента мутантного варіанта гена ahpC, що приводить до стійкості мікроорганізмів до ізоніазиду; м) комплементарні до послідовностей, охарактеризованих у к) і л); н) відповідні послідовності фрагмента мобільного елемента IS6110; о) комплементарні до послідовностей, охарактеризованих у н); (Г) - гібридизацію ампліфікованих мічених продуктів, отриманих на стадії (Б), на біочипі в умовах, які забезпечують розрізнення в один нуклеотид між довершеними і недовершеними дуплексами, які утворюються в результаті гібридизації; (Д) - реєстрацію й інтерпретацію результатів гібридизації. 2. Спосіб за п. 1, який відрізняється тим, що на другій стадії мультиплексної ПЦР (Б) з метою одержання переважно одноланцюгових флуоресцентно мічених фрагментів для всіх використаних пар праймерів флуоресцентно мічений праймер використовують у молярному надлишку відносно іншого праймера, який входить до тієї ж пари. 3. Спосіб за п. 1, який відрізняється тим, що ампліфікацію фрагментів генів і мобільного елемента IS6110 проводять, використовуючи безпосередньо матеріал клінічного зразка (мокротиння, ексудат, змивки, бронхоальвеолярний лаваж) або попередньо вирощену культуру мікроорганізмів. 4. Спосіб за п. 1, який відрізняється тим, що з метою забезпечення розрізнення в один нуклеотид між довершеними і недовершеними дуплексами, які утворюються в результаті гібридизації, використовують гібридизаційний буфер, який дозволяє проводити гібридизацію в розширеному інтервалі температур. 5. Спосіб за п. 1, який відрізняється тим, що реєстрацію результатів на стадії (Д) проводять за допомогою портативного аналізатора флуоресценції й програмного забезпечення, що дозволяє використовувати програмну обробку інтенсивностей сигналів з наступною інтерпретацією результатів. 6. Спосіб за п. 1, який відрізняється тим, що інтерпретацію зареєстрованих результатів на стадії (Д) виконують шляхом порівняння інтенсивності флуоресценції сигналів комірок, у яких утворилися довершені й недовершені гібридизаційні дуплекси. 7. Спосіб за п. 1, який відрізняється тим, що інтерпретовані результати можуть бути застосовані для підтвердження клінічного діагнозу - туберкульоз, і цілей епідеміологічного генотипування, використовуючи як маркер наявність тієї або іншої мутації. 8. Набір специфічних пар праймерів для здійснення способу одночасного виявлення мікобактерій туберкульозного комплексу й ідентифікації мутацій ДНК мікобактерій, які приводять до стійкості їх до рифампіцину й ізоніазиду, у клінічних зразках за п. 1, причому послідовності праймерів представлені SEQ ID NO: 70-85. 9. Біочип, використовуваний у способі одночасного виявлення мікобактерій туберкульозного комплексу й ідентифікації мутацій ДНК мікобактерій, які приводять до стійкості до рифампіцину й ізоніазиду, у клінічних зразках за п. 1, який є підкладкою з гелевими елементами, у кожному з яких іммобілізований унікальний олігонуклеотидний зонд, причому послідовності зондів представлені SEQ ID NO: 1-69. 10. Набір олігонуклеотидних зондів, використовуваний для одержання біочипа, що його використовують в способі одночасного виявлення мікобактерій туберкульозного комплексу й ідентифікації мутацій ДНК мікобактерій, які приводять до стійкості до рифампіцину й ізоніазиду, у клінічних зразках за п. 1, причому зонди мають послідовності SEQ ID NO: 1-69. Винахід відноситься до молекулярної біології, мікробіології й медицини та забезпечує спосіб виявлення мікобактерій туберкульозного комплексу (Mycobacterium tuberculosis, M. bovis, M. bovis BCG, M. africanum i M. microti) з одночасним визначенням чутливості штамів до рифампіцину й ізоніазиду в клінічному зразку на диференціюючому біочіпі. Для виявлення мутацій, відповідальних за стійкість до лікарських засобів, у цей час застосовуються наступні методи: I. Екстанція ланцюга міченим дідезоксирибонуклеозидтри фосфатом (SNP - single nucleotide polymorphism); Dubiley S., Kirillov E and A. Mirzabekov. 1999. Polymorphism analysis and gene detection by minisequencing on an array of gel-immobilized primers. Nucleic Acids Research, Vol.27, No.18 (e19). Marth GT, Korf I, Yandell MD et al. 1999. A general approach to single-nucleotide polymorphism discovery. Nat Genet., Dec/23 (4):452-456. II. Алель-Специфічна ПЦР (Allele specific PCR); De los Monteras L.E.E., J.C.Galan, M.Gutierrez, S.Samper, J.F.G.Marin, C.Martin, L.Doninguez, L. de Rafael, F.Baquero, E.Gomez-Mampaso, and J.Blazquez. 1998. Allele-Specific PCR Method Based on pncA and oxyR Sequences for Distinguishing Mycobacterium bovis from Mycobacterium tuberculosis: Intraspecific M.bovis pncA Sequence Polymorphism. J. Clin. Microbiol. 36:, 239-242. III. Вивчення поліморфізму фрагментів після рестрикції (RFLP - restriction fragment length polymorphism); 5 PCR-RFLP Detection of point Mutations in the Catalase-Peroxidase Gene (katG) of Mycobacterium tuberculosis Associated with Isoniazid Resistance. In: PCR Protocols for Emerging Infectious Diseases (D.H.Persing, Ed.) (1996) ASM Press, Washington, pp.144-149. IV. Аналіз конформаційного поліморфізму одно- і дволанцюгової ДНК (SSCP і DSCP - single(double)-strand conformation polymorphism); Delgado M.B. and A.Telenti. Detection of Fluoroquinolone Resistance Mutations in Mycobacterium tuberculosis. In: PCR Protocols for Emerging Infectious Diseases (D.H.Persing, Ed.) (1996) ASM Press, Washington, pp.138-143. Pretorius G.S., P.D. van Helden, F.Sirgel, K.D.Eisenach and T.C.Victor. 1995. Mutations in katG Gene Sequencesin Isoniazid-Resistant Clinical Isolatesof Mycobacterium tuberculosis Are Rare. Antimicrob. Agents Chemother., 39, 10, 2276-2281. V. Гібридизація на олігонуклеотидних мікро матрицях високої щільності (Hybridization on highdensity microarray); Troesch A, Nguyen H, Miyada CG et al. Mycobacterium species identification and rifampin resistance testing with high-density DNA probe arrays. J Clin Microbiol 1999; 37: 49-55. W. Sougakoff, M. Rodrigue, C. Truffot-Pernot, M. Renard, N. Durin, M. Szpytma, R. Vachon, A. Troesch and V. Jarlier. Use of a high-density DNA probe array for detecting mutations involved in rifampicin resistance in Mycobacterium tuberculosis. Clin Microbiol Infect. 2004;10(4) : 289-94. VI. Гібридизація на спеціалізованих олігонуклеотидних мікрочіпах (Hybridization on specialized microarray); Jun Yue, Wei Shi, Jingping Xie, Yao Li, Erliang Zeng and Honghai Wang. Detection of rifampinresistant Mycobacterium tuberculosis strains by using a specialized oligonucleotide microarray. Diagn Microbiol Infect Dis 2004; 48: 47-54. Грядунов Д.Α., В.M. Михайловіч, С.А. Лапа, Η.І. Рудінскій, В.E. Панський, А.В. Чудінов, А.С. Засєдатєлєв, А.Д. Мірзабеков. Ідентифікація штамів Mycobacterium tuberculosis з одночасним визначенням їхньої стійкості до ліків методом гібридизації на олігонуклеотидних мікрочіпах. Молекулярна генетика, мікробіологія, вірусологія 2003; (4): 24-7. VII. Методи, засновані на секвенуванні (sequencing); Kapur, V., L.-L. Li, S. Iordanescu, M. R. Hamrick, A. Wanger, B. N. Kre-iswirth, and J. M. Musser. 1994. Characterization by automated DNA sequencing of mutations in the gene (rpoB) encoding the RNA polymerase b subunit in rifampin-resistant Mycobacterium tuberculosis strains from New York City and Texas. J. Clin. Microbiol. 32:1095-1098. Cavusoglu С, Karaca-Derici Y, Bilgiç A. In-vitro activity of rifabutin against rifampicin-resistant Mycobacterium tuberculosis isolates with known rpoB mutations. Clin Microbiol Infect. 2004; 10(7): 662-5. VIІІ. Дідезоксифінгерпринтінг (ddF-method); Rys P.N. and T.A.Felmlee. Detection of Rifampicin Resistance Mutations in Mycobacterium tuberculosis by Dideoxy Fingerprinting. In: PCR Protocols for Emerging Infectious Diseases 92720 6 (D.H.Persing, Ed.) (1996) ASM Press, Washington, pp.112-121. IX. ПЦР-Гетеродуплексний аналіз (PCRheteroduplex analysis); Williams D.L., C.W.Limbers, L.Spring, S.Jayachandra, and T.P.Gillis. PCR-Heteroduplex Detection of Rifampicin-Resistant Mycobacterium tuberculosis. In: PCR Protocols for Emerging Infectious Diseases (D.H.Persing, Ed.) (1996) ASM Press, Washington, pp.122-129. X. Метод недовершеного дуплекса із РНК (RNA mismatch analysis); Dracopoli, N.C. Ed. "Dеtection of Mutations by RNase Cleavage". Current Protocols in Human Genetics. 1998. John Wiley & Sons, Inc. Marth GT, Korf I, Yandell MD et al. 1999. A general approach to single-nucleotide polymorphism discovery. Nat Genet., Dec;23(4): 452-456. XI. Метод структурно-специфічного розщеплення (Structure-specific endonuclease cleavage); Brow M.A.D., M.C.Oldenburg, V.Lyamichev, L.M.Heisler, N.Lyamicheva, J.G.Hall, N.J.Eagan, D.M.Olive, L.M.Smith, Լ L.Fors, and J.E.Dahlberg. 1996. Differentiation of Bacterial 16S rRNA Genes and Intergenic Regions and Mycobacterium tuberculosis katG Genes by Structure-Specific Endonuclease Cleavage. J. Clin. Microbiol., 34: 31293137. XII. Метод Зондів (Line Probe assay (LiPA)); Rossau R, Traore H, De Beenhouwer H, Mijs W, Jannes G, De Rijk Ρ, Portaels F. Evaluation of the INNO-LiPA Rif. ТВ assay, a reverse hybridization assay for the simultaneous detection of Mycobacterium tuberculosis complex and its resistance to rifampin. Antimicrob Agents Chemother 1997; 41: 2093-2098. Drobniewski FA, Watterson SA, Wilson SM, Harris GS. A clinical, microbiological and economic analysis of a national UK service for the rapid molecular diagnosis of tuberculosis and rifampin resistance in Mycobacterium tuberculosis. J Med Microbiol 2000; 49: 271-278. XIII. Метод із застосуванням фагів (PhaB assay). Wilson, S. M., Ζ. Al-Suwaidi, R. McNerney, J. Porter, and F. Drobniewski. 1997. Evaluation of a new rapid bacteriophage-based method for the drug susceptibility testing of Mycobacterium tuberculosis. Nat. Med. 3: 465-468. XIV. Метод розщеплення клівазою (CFLP Cleavase fragment length polymorphism). Sreevatsan S, Bookout JB, Ringpis FM, Mogazeh SL, Kreiswirth BN, Pottathil RR, Barathur RR. 1998. Comparative Evaluation of Cleavase Fragment Length Polymorphism With PCR-SSCP and PCRRFLP to Detect Antimicrobial Agent Resistance in Mycobacterium tuberculosis. Mol Diagn. 1998 Jun; 3(2): 81-91. XV. Методи, засновані на лігуванні (Ligationbased assays). Mikhailovich V., Lapa S., Gryadunov D., Sobolev Α., Strizhkov В., Chernyh Ν., Skotnikova O., Irtuganova O., Moroz Α., Litvinov V., Vladimirskii M., Perelman M., Chernousova L., Erokhin V., Zasedatelev Α., and Mirzabekov A. 2001. 7 Identification of rifampin-resistant Mycobacterium tuberculosis strains by hybridization, PCR, and ligase detection reaction on oligonucleotide microchips. J. Clin. Microbiol. 39: 2531-2540. XVI. Метод ПЦР із детекцією у режимі реального часу (Real-time PCR). Marin M, Garcia de Viedma D, Ruiz-Serrano MJ, Bouza E. Rapid direct detection of multiple rifampin and isoniazid resistance mutations in Mycobacterium tuberculosis in respiratory samples by real-time PCR. Antimicrob Agents Chemother. 2004 Nov; 48(11): 4293-300. XVII. Mac-спектрометричні методи (Differential sequencing with mass spectrometry). Griffin, T.J. and L.M. Smith. 2000. Single nucleotide polymorphism analysis by MALDI - TOF mass spectrometry. Trends Biotechnol. 18: 77-84. XVIII. Метод резонансного переносу енергії на флуорофор (Fluorescence Resonance Energy Transfer). Viedma G. D., del Sol Diaz Infantes M, Lasala F, Chaves F, Alcala L, Bouza E. 2002. New Real-Time PCR Able To Detect in a Single Tube Multiple Rifampin Resistance Mutations and High-Level Isoniazid Resistance Mutations in Mycobacterium tuberculosis. J Clin Microbiol. 40(3): 988-95. XIX. ПЦР на олігонуклеотидних мікрочіпах (PCR on oligonucleotide microchips). Mikhailovich V., Lapa S., Gryadunov D., Sobolev A., Strizhkov В., Chernyh N., Skotnikova O., Irtuganova O., Moroz A., Litvinov V., Vladimirskii M., Perelman M., Chernousova L., Erokhin V., Zasedatelev A., and Mirzabekov A. 2001. Identification of rifampin-resistant Mycobacterium tuberculosis strains by hybridization, PCR, and ligase detection reaction on oligonucleotide microchips. J. Clin. Microbiol. 39: 2531-2540. Tillib SV, Strizhkov BN, Mirzabekov AD. Integration of multiple PCR amplifications and DNA mutation analyses by using oligonucleotide microchip. Anal Biochem. 2001 May 1; 292(1): 155-60. Для виявлення мікобактерій туберкульозного комплексу застосовуються наступні методи: XX. Установлення наявності мобільного генетичного елемента IS 6110 або білка теплового шоку (65кДа) методом ПЦР (Polymerase Chain Reaction for IS 6110 or 65kD heat shock protein); Gunisha P, Madhavan HN, Jayanthi U, Therese KL. 2001. Polymerase chain reaction using IS6110 primer to detect Mycobacterium tuberculosis in clinical samples. Indian J Pathol Microbiol. 44(2): 97102. XXI. Установлення наявності гена рибосомного спейсера методом ПЦР (Ribosomal intergenic spacer detection by Polymerase Chain Reaction); Glennon M, Smith T, Cormican M, Noone D, Barry T, Maher M, Dawson M, Gilmartin JJ, Gannon F. 1994. The ribosomal intergenic spacer region: a target for the PCR based diagnosis of tuberculosis. Tuber Lung Dis 75(5): 353-60. Kraus G, Cleary T, Miller N, Seivright R, Young AK, Spruill G, Hnatyszyn HJ. 2001. Rapid and specific detection of the Mycobacterium tuberculosis complex using fluorogenic probes andreal-time PCR. Моl Cell Probes 15(6): 375-83. 92720 8 XXII. Автоматизована ПЦР на приладі Амплікор (Рош) (Amplicor M. tuberculosis test (ROCHE) for amplifying part of the 16S rRNA gene.) J. Schirm, L. A. B. Oostendorp, and J. G. Mulder. 1995. Comparison of Amplicor, In-House PCR, and Conventional Culture for Detection of Mycobacterium tuberculosis in Clinical Samples. J. Clin. Microbiol. 33: 3221-3224. XXIII. Лігазна ланцюгова реакція (Ligase Chain Reaction). Viinanen AH, Soini H, Marjamaki M, Liippo K, Viljanen MK. 2000. Ligase chain reaction assay is clinically useful in the discrimination of smear-positive pulmonary tuberculosis from atypical mycobacterioses. Ann Med 32(4): 279-83. Lin IJ, Che MJ, Yeh A, Hwang JJ, Wei CY, Tsao WL, Lee CP. 1999. Comparison of the sensitivity and specificity of an automatic ligase chain reaction assay system with a one-step polymerase chain reaction assay in the diagnosis of Mycobacterium tuberculosis complex. Changgeng Yi Xue Za Zhi 22(2): 204-11. XXIV. Метод ПЦР із детекцією у режимі реального часу (Real-time PCR). Shrestha NK, Tuohy MJ, Hall GS, Reischl U, Gordon SM, Procop GW. Detection and differentiation of Mycobacterium tuberculosis and nontuberculous mycobacterial isolates by real-time PCR. J Clin Microbiol. 2003 Nov; 41(11): 5121-6. Для перерахованих вище методів установлення мутацій відзначені наступні недоліки: Методи екстенції ланцюга й алельспецифічної ПЦР у пробірці (І, II) вимагають постановки незалежних реакцій відповідно до числа досліджуваних мутацій (тобто більше 30 у випадку гена rроВ) і, відповідно, великої кількості досліджуваного зразка; Методи вивчення поліморфізму (III, IV), ПЦРгетеродуплексний аналіз (IX) і метод недовершеного дуплекса із РНК (X) трудомісткі, потребують великої кількості часу, дають непрямий висновок про тип мутації й вимагають типових стандартів (на кожну мутацію); крім того, для методу недовершеного дуплекса пред‟являються підвищені вимоги щодо відсутності РНК-ази, він трудомісткий і неприйнятний для детекції дуплекса G-U (Nash, K.A., A. Gaytan, and С.В. Inderlied. 1997. Detection of rifampin resistance in Mycobacterium tuberculosis by means of a rapid, simple, and specific RNA/RNA mismatch assay. J. Infect. Dis. 176: 533-536; Telenti, A., P. Imboden, F. Marchesi, D. Lowrie, S. Cole, M.J. Colston, L. Matter, K. Schopfer, and T. Bodmer. 1993. Detection of rifampin resistance mutations in Mycobacterium tuberculosis. Lancet 341: 647-650); Гібридизація на олігонуклеотидних мікроматрицях високої щільності (V) вимагає складного комп‟ютерного обчислення отриманих результатів і дорогих видаткових матеріалів (зокрема мікроматриця); у цей час розроблені мікрочіпи високої щільності тільки для аналізу мутацій у гені rроВ, що приводять до стійкості до рифампіцину; Відомі методи гібридизації на спеціалізованих мікрочіпах (VI) не можуть бути застосованими для аналізу клінічних зразків, оскільки як досліджуваний матеріал використовується ДНК, виділена з культур штамів, що вимагає додаткового культи 9 вування й очищення клітин, отриманих із клінічного матеріалу (Д.А. Грядунов, В.М. Михайловіч, С.А. Лапа, Н.І. Рудинський, В.Е. Панський, А.В. Чудінов, А.С. Засєдатєлєв, А.Д. Мірзабеков. Ідентифікація штамів Mycobacterium tuberculosis з одночасним визначенням їхньої стійкості до ліків методом гібридизації на олігонуклеотидних мікрочіпах. Молекулярна генетика, мікробіологія, вірусологія 2003; (4): 24-7; Yue Jue,Wei Shi, Jingping Xie,Yao Li, Erliang Zeng and Honghai Wang. Detection of rifampin-resistant Mycobacterium tuberculosis strains by using a specialized oligonucleotide microarray. Diagn Microbiol Infect Dis 2004; 48: 47-54); Пряме визначення нуклеотидної послідовності (VII) вимагає виділення чистої культури, постановки реакцій ампліфікації й секвенування й додаткового очищення продукту для аналізу на автоматичному секвенаторі; Методи дидезоксифінгерпринтінга (VIII) і структурно-специфічного розщеплення (XI) вимагають попередньої стандартизації (підбора умов), що збільшує трудомісткість, а також наявності типових стандартів. Крім того, дидезоксифінгерпринтінг вимагає використання радіоактивної мітки, що перешкоджає його широкому використанню в умовах клінічної лабораторії; Комерційний набір INNO-LiPA (XII) відрізняється високою собівартістю й ідентифікує обмежену кількість мутацій у гені rpoB (Rossau R, Traore H, De Beenhouwer H, Mijs W, Jannes G, De Rijk P, Portaels F. Evaluation of the INNO-LiPA Rif. ТВ assay, a reverse hybridization assay for the simultaneous detection of Mycobacterium tuberculosis complex and its resistance to rifampin. Antimicrob Agents Chemother 1997; 41: 2093-2098); Метод із застосуванням фагів (XIII) потребує великої кількості часу, тому що пов‟язаний з реплікацією фагів і наступною реєстрацією лізису на культурі М. smegmatis, a також він трудомісткий; Методи розщеплення клівазою (XIV) і масспектрометрії (XVII) вимагають гомогенного високо очищеного зразка ДНК; стадія підготовки зразка й вартість устаткування також є обмежуючими факторами для широкого застосування масспектрометричного аналізу (XVII); Методи, засновані на літуванні (XV), застосовуються дуже обмежено через складність створення набору зондів на велику кількість детектованих мішеней, використовують дорогий фермент - термостабільну ДНК-лігазу; ПЦР із детекцією у режимі реального часу (XVI) виявляє наявність тільки найпоширеніших мутацій у досліджуваному фрагменті генома, але не дозволяє точно ідентифікувати нуклеотидну заміну й при цьому є досить дорогим для рутинного аналізу; Метод резонансного переносу енергії на флуорофор (XVIII) має два серйозні недоліки: 1) здатний визначати обмежену кількість мутацій і 2) не здатний диференціювати функціонально значимі мутації від мутацій, які фенотипічно не проявляються; Методи ПЦР і літування на олігонуклеотидних мікрочіпах (XIX) вимагають подальшого вдоскона 92720 10 лювання й складні для виконання в практичних лабораторіях. Всі перераховані вище методи визначення мікобактерій туберкульозного комплексу, які ґрунтуються на ампліфікації IS-елемента (XX), білка теплового шоку (XX), гена рибосомного спейсера (XXI), а також методи автоматизованої ПЦР (XXII), лігазної ланцюгової реакції (XXIII) і ПЦР із детекцією у режимі реального часу (XXIV) здатні виявити присутність у досліджуваному зразку збудника туберкульозу, але не визначити чутливість останніх до антимікобактеріальних препаратів. Переваги способу виявлення штамів мікобактерій туберкульозного комплексу, стійких до рифампіцину й ізоніазиду, на мініатюрних біологічних чіпах. Спосіб виявлення в клінічних зразках штамів мікобактерій туберкульозного комплексу, стійких до рифампіцину й ізоніазиду, на мініатюрних біологічних чіпах вигідно відрізняється від відомих з рівня техніки методів можливістю ідентифікації збудника безпосередньо в клінічному матеріалі з одночасним визначенням стійкості до двох антимікобактеріальних препаратів першої черги; можливістю встановлення типу мутації, що визначає резистентність, а також низькою собівартістю, коротким проміжком часу, необхідного для одержання результату. Метод не вимагає дорогого устаткування й висококваліфікованого персоналу. Дані, отримані за допомогою методу гібридизації, можуть бути використані для визначення терапевтичної дози лікарського препарату й епідеміологічного генотипування. У своєму першому аспекті даний винахід забезпечує прискорений спосіб одночасного виявлення мікобактерій туберкульозного комплексу в клінічному матеріалі й визначення їхньої стійкості до рифампіцину й ізоніазиду на підставі ідентифікації мутацій ДНК мікобактерій, що приводять до стійкості мікроорганізмів до рифампіцину й ізоніазиду, з використанням мініатюрного біочіпа. Спосіб заснований на двохстадійній мультиплексній ПЦР із одержанням одноланцюгових флуоресцентно мічених фрагментів ДНК із наступною гібридизацією цих фрагментів на чіпі, що містить набір диференціюючих олігонуклеотидів. Спосіб передбачає наступні стадії: (A) - мультиплексну ампліфікацію фрагментів генів rроВ, katG, inhA, ahpC, мобільного елемента IS6110 з використанням набору пар специфічних праймерів для першої стадії ПЦР; (Б) - мультиплексну ампліфікацію фрагментів генів rроВ, katG, inhA, ahpC, мобільного елемента IS6110 з використанням як матриці продукту ПЦР, отриманого на стадії (А), і набору пар специфічних праймерів для другої стадії ПЦР, причому один праймер у кожній парі праймерів є флуоресцентно міченим, з одержанням переважно одноланцюгових флуоресцентно мічених фрагментів; (B) - забезпечення біочіпа для одночасного виявлення мікобактерій туберкульозного комплексу й ідентифікації мутацій, що приводять до стійкості до рифампіцину й ізоніазиду, який є підкладкою з гелевими елементами, у кожному з яких іммобілізований унікальний олігонуклеотидний 11 зонд, який має послідовність, обрану із групи, яка включає послідовності: а) відповідні до послідовності фрагмента гена rроВ дикого типу; б) відповідні до послідовності фрагмента мутантного варіанта гена rроВ, який приводить до стійкості мікроорганізмів до рифампіцину; в) комплементарного до послідовностей, охарактеризованих в а) і б); г) відповідні до послідовності фрагмента гена katG дикого типу; д) відповідні до послідовності фрагмента мутантного варіанта гена katG, який приводить до стійкості мікроорганізмів до ізоніазиду; e) комплементарного до послідовностей, охарактеризованих у г) і д); ж) відповідні до послідовності фрагмента гена inhA дикого типу; з) відповідні до послідовності фрагмента мутантного варіанта гена inhA, який приводить до стійкості мікроорганізмів до ізоніазиду; і) комплементарного до послідовностей, охарактеризованих у ж) і з); к) відповідні до послідовності фрагмента гена ahpC дикого типу; л) відповідні до послідовності фрагмента мутантного варіанта гена ahpC, який приводить до стійкості мікроорганізмів до ізоніазиду; м) комплементарного до послідовностей, охарактеризованих у к) і л); н) відповідні до послідовності фрагмента мобільного елемента IS6110; о) комплементарні до послідовності, охарактеризованої в н); (Г) - гібридизацію ампліфікованних мічених продуктів, отриманих на стадії (Б), на біочіпі в умовах, що забезпечують розрізнення в один нуклеотид між утвореними в результаті гібридизації довершеними й недовершеними дуплексами; (Д) - реєстрацію й інтерпретацію результатів гібридизації. В одному зі своїх втілень спосіб характеризується тим, що на першій стадії мультиплексної ПЦР (А) використовують набір пар специфічних праймерів, послідовності яких представлені SEQ ID NO: 70, 71, 74, 75, 77, 78, 80, 81, 83, 84. У ще одному із втілень спосіб характеризується тим, що на другій стадії мультиплексної ПЦР (Б) використовують набір пар специфічних праймерів, послідовності яких представлені SEQ ID NO: 72, 73, 74, 76, 77, 79, 80, 82, 83, 85. У наступному втіленні спосіб характеризується тим, що на другій стадії мультиплексної ПЦР (Б) з метою одержання переважно одноланцюгових флуоресцентно мічених фрагментів для всіх використовуваних пар праймерів флуоресцентно мічений праймер використовують у молярному надлишку відносно іншого праймеру, який утворює ту ж пару. У наступному втіленні спосіб характеризується тим, що ампліфікацію фрагментів генів і мобільного елемента IS6110 проводять, використовуючи безпосередньо матеріал клінічного зразка (мокротиння, ексудат, змив, бронхо-альвеолярний лаваж) або попередньо вирощену культуру мікроорганізмів. У ще одному із втілень спосіб характеризується тим, що біочіп містить набір іммобілізованих олігонуклеотидів, послідовності яких представлені SEQ ID NO: 1-69. У наступному втіленні спосіб характеризується тим, що з метою забезпечення розрізнення в один 92720 12 нуклеотид між довершеними й недовершеними дуплексами, які утворюються в результаті гібридизації, використовують гібридизаційний буфер, який дозволяє проводити гібридизацію в розширеному інтервалі температур. В одному із втілень спосіб характеризується тим, що реєстрацію результатів на стадії (Д) проводять за допомогою портативного аналізатора флуоресценції й програмного забезпечення, що дозволяє використовувати програмну обробку інтенсивностей сигналів з наступною інтерпретацією результатів. У ще одному із втілень спосіб характеризується тим, що інтерпретацію зареєстрованих результатів на стадії (Д) виконують шляхом порівняння інтенсивності флуоресценції сигналів комірок, у яких утворилися довершені й недовершені гібрідизаційні дуплекси. Нарешті в ще одному із втілень спосіб характеризується тим, що інтерпретовані результати можуть бути застосовані для підтвердження клінічного діагнозу захворювання на туберкульоз і з метою епідеміологічного генотипування, використовуючи як маркер наявність тієї або іншої мутації. У своєму наступному аспекті даний винахід забезпечує набір специфічних пар праймерів для здійснення способу, який є першим аспектом даного винаходу, одночасного виявлення мікобактерій туберкульозного комплексу й ідентифікації мутацій ДНК мікобактерій, що приводять до їх стійкості до рифампіцину й ізоніазиду, у клінічних зразках, причому послідовності праймерів представлені SEQ ID NO: 70-85. У ще одному аспекті даний винахід відноситься до біочіпу, що його використовують у способі, який є першим аспектом даного винаходу, одночасного виявлення мікобактерій туберкульозного комплексу й ідентифікації мутацій ДНК мікобактерій, що приводять до стійкості до рифампіцину й ізоніазиду, у клінічних зразках, де біочіп є підкладкою з гелевими елементами, у кожному з яких іммобілізований унікальний олігонуклеотидний зонд, причому послідовності зондів представлені SEQ ID NO: 1-69. Нарешті, ще одним аспектом даного винаходу є набір олігонуклеотидних зондів, які мають послідовності SEQ ID NO: 1-69, який використовують для одержання біочіпа, що його використовують в способі одночасного виявлення мікобактерій туберкульозного комплексу й ідентифікації мутацій ДНК мікобактерій, що приводять до стійкості до рифампіцину й ізоніазиду, у клінічних зразках, що є першим аспетктом даного винаходу. Інші аспекти даного винаходу стануть зрозумілими із прикладених фігур, докладного опису й формули винаходу. Для більш ясного розуміння сутності заявленого винаходу, а також для демонстрації його характерних рис і переваг далі приводиться докладний опис винаходу з посиланнями на фігури креслень, на яких Фіг.1. представляє схему розміщення дискримінуючих олігонуклеотидів на біочіпі. Фіг.2. представляє результати гібридизації зразка ДНК М. tuberculosis дикого типу. 13 Фіг.3. представляє результати гібридизації зразка ДНК М. tuberculosis, що містить мутації, які приводять до амінокислотних замін Ser531>Leu у гені rроВ і Ser315>Thr у гені katG. Фіг.4. представляє результати гібридизації зразка ДНК М. tuberculosis, що містить мутації, які приводять до амінокислотної заміни Asp516>Val у гені rроВ і нуклеотидної заміни C>Т15 у промоторній області гена inhA. Фіг.5. представляє результати гібридизації зразка ДНК М. bovis BCG, яка не містить замін у генах rроВ і katG, і регуляторних областях генів inhA і аhрС. Фіг.6. представляє результати гібридизації зразка ДНК М. avium S58. Завдання даного винаходу полягає у створенні прискореного способу виявлення мікобактерій туберкульозного комплексу в клінічному зразку з одночасним визначенням стійкості мікобактерій до рифампіцину й ізоніазиду, за допомогою виявлення мінорного поліморфізму (крапкових нуклеотидних замін, делецій, інсерцій) ДНК мікобактерій, що приводить до виникнення резистентності. У заявленому методі запропоноване використання: мультиплексної полімеразної ланцюгової реакції для одночасної ампліфікації послідовностей мобільного елемента IS 6110, генів rpoB, katG, inhA, ahpC, одержання із напрацьованих продуктів одноланцюгових флуоресцентно мічених фрагментів зазначених генів, при цьому як вихідний зразок може бути використаний клінічний матеріал, лізат клітинної культури, інші фізіологічні рідини. Заявлений спосіб також передбачає використання оригінального олігонуклеотидного біочіпа з іммобілізованими специфічними зондами, процедури гібридизації, реєстрації й інтерпретації результатів. Принципова схема виявлення рифампіцин- і ізоніазид-стійких штамів мікобактерій туберкульозного комплексу на біочіпі. Клінічний зразок (у випадку легеневої форми захворювання - мокротиння) піддають деконтамінації й лізису клітин з метою забезпечення доступу до геномної ДНК. Одним із придатних способів є розрідження в лужному середовищі у присутності N-ацетил-L-цистеіну й кип‟ятіння з детергентом для забезпечення доступу до ДНК і деконтамінації зразка. Для цих цілей також можуть бути використані інші способи, відомі фахівцям у даній галузі, такі як руйнування клітин за допомогою ультразвуку (Padilla E, Gonzalez V, Manterola JM, et al. Evaluation of two different cell lysis methods for releasing mycobacterial nucleic acids in the INNOLiPA mycobacteria test. Diagn Microbiol Infect Dis. 2003 May;46(1): 19-23), лізис за допомогою гуанідину тіоціанату - саркозіну (Kotlowski R., Martin A., Ablordey A. et al. One-tube cell lysis and DNA extraction procedure for PCR-based detection of Mycobacterium ulcerans in aquatic insects, molluscs and fish. J Med Microbiol. 2004 Sep; 53(Pt9): 927-33) і т.д. Специфічну ампліфікацію фрагментів функціонально значимих генів виконують на першій стадії за допомогою мультиплексної ПЦР. На другому етапі одержують переважно одноланцюгові флуо 92720 14 ресцентно мічені продукти методом асиметричної мультиплексної ПЦР із використанням праймерів, які містять на 5‟-кінці флуоресцентний барвник. Праймери для проведення першої стадії ампліфікації вибирають таким чином, щоб вони фланкували регіон гена або регуляторної області, де знаходяться мутації, які найчастіше зустрічаються, і які що приводять до стійкості мікроорганізму до хіміопрепаратів. Використовуючи спеціалізоване програмне забезпечення, наприклад Oligo v.6.3 (Molecular Biology Insights Inc., США) або Fast PCR (http://www.biocenter.helsinki.fi/bi/Programs/fastpcr.ht m) або інші комерційно доступні програми, або програми, вільно доступні в мережі Internet, розраховують температури плавлення праймерів і, варіюючи їхню довжину, домагаються того, щоб розкид температур відпалу праймерів усередині набору не перевищував 3-4°С. При підборі праймерів уникають таких послідовностей, які здатні формувати вторинні структури типу шпильки з високими температурами плавлення. При виборі праймерів для мультиплексної ПЦР уникають таких послідовностей, які утворюють між собою дуплекси, що складаються більш ніж із трьох-п‟яти нуклеотидів. Кожний обраний праймер повинен мати унікальну специфічність відносно аналізованої ділянки послідовності нуклеїнових кислот генома мікобактерій туберкульозного комплексу. Специфічність праймерів перевіряється за допомогою програмного забезпечення, яке використовує пошук у базах нуклеотидних послідовностей за алгоритмом BLAST (наприклад, www.ncbi.nlm.nih.gov/BLAST). Праймери для одночасної ампліфікації декількох ділянок генома вибирають при можливості таким чином, щоб довжини ампліфікованих фрагментів різнилися на 30-50 пар основ (п.о), що дозволяє візуально контролювати наявність/відсутність специфічної смуги, яка відповідає продукту ампліфікації, електрофорезом в 2% агарозному гелі. Праймери для проведення другої стадії ампліфікації вибирають із урахуванням вимог, викладених вище, з тією відмінністю, що принаймні один із праймерів обирається усередині ПЦРфрагмента, що його одержують на першій стадії, яка підвищує специфічність реакції. Праймери вибирають таким чином, щоб розмір ампліфікованих фрагментів на другій стадії становила 70400п.о. Більша довжина ПЦР-продуктів, отриманих на другій стадії, утруднює ефективну дифузію аналізованих фрагментів генома в гелевих елементах біочіпа при гібридизації, що в кінцевому підсумку може привести до зменшення кількості гібридизаційних дуплексів, що утворилися і, як наслідок, падіння флуоресцентного сигналу в комірках. При підборі праймерів варто враховувати той факт, що на виході реакції виходять переважно одноланцюгові флуоресцентно мічені продукти, комплементарні до олігонуклеотидів, іммобілізованих на біочіпі. Тому в кожній парі праймер, який містить флуоресцентну мітку і який застосовується у надлишку («ведучий праймер»), обирається з того ланцюга, послідовність якого комплементарна до послідовностей іммобілізованих у комірках біочіпа олігонуклеотидів. Тобто, у випадку, якщо олі 15 гонуклеотиди для іммобілізації обрані зі значеннєвого ланцюга, для формування гібридизаційних дуплексів в комірках біочіпа необхідна переважна ампліфікація антизначеннєвого ланцюга, тим самим «ведучий праймер» обирається з ланцюга, комплементарного до послідовності гена (антизначеннєвий ланцюг), і навпаки. Як флуоресцентний барвник може бути використаний будь-який флуоресцентний барвник, який може бути хімічно приєднаний до 5‟-кінця олігонуклеотидного праймера і який не буде істотно перешкоджати проходженню полімеразної ланцюгової реакції. Спектр таких барвників добре відомий фахівцеві в даній галузі техніки й включає, наприклад, такі барвники флуоресцеинового (TAMRA®, ROX®, JOE®) , родамінового (Texas Red®), поліметинового (Су3®, Су5®, Су5.5®, Су7®) рядів (Ranasinghe R. and Brown T. Fluorescence based strategies for genetic analysis. Chem. Covmun., 2005, 5487-5502). Флуоресцентні барвники комерційно доступні, зокрема виробництва фірми Molecular Probes, США. Найкращими є барвники, спектр збудження яких лежить у довгохвильовій (червоній) області спектра, що дозволяє використовувати для збудження недорогі джерела збудливого випромінювання типу напівпровідникових лазерів. Флуоресцентний барвник може бути приєднаний до 5‟-кінця праймера як безпосередньо, так і через проміжний спейсер, наприклад, 5‟-AminoModifier фірми Glen Research. Для флуоресцентного мічення праймера флуоресцентний барвник використовують у вигляді реакційноздатної похідної, наприклад, сукцинімідного ефіру. Ковалентне приєднання флуоресцентного барвника може здійснюватися в ручному режимі після закінчення синтезу праймера й відділення його у вільному вигляді від твердої фази (CPG), на якій проводився синтез. Для очищення флуоресцентно міченого праймера від барвника, що не вступив в реакцію та інших побічних продуктів реакції використовується високоефективна рідинна хроматографія (ВЕРХ). При виборі дискримінуючих олігонуклеотидів для іммобілізації на біочіпі з урахуванням розміру й складності аналізованої послідовності й, зокрема, наявності повторів і протяжних гомополімерних послідовностей визначають довжину дискримінуючих олігонуклеотидів, яка забезпечує їх специфічність відносно аналізованої послідовності. Для кожної позиції, для якої відомі мутації, поліморфізм або алельні варіанти, підбирають набір специфічних дискримінуючих олігонуклеотидів, здатний виявляти відомі заміни, делеції й інсерції. Використовуючи програмне забезпечення, наприклад Oligo v.6.3 (Molecular Biology Insights Inc., США), розраховують температури плавлення олігонуклеотидів і, варіюючи їхню довжину, домагаються того, щоб розкид температур плавлення олігонуклеотидів становив не більше 2-3°С. Уникають таких олігонуклеотидів, які здатні формувати вторинні структури типу шпильки з високими температурами плавлення. Положення тих варіабельних нуклеотидів, визначення яких проводять, та інших нуклеотидних перебудов обирають при 92720 16 можливості не далі 1-4 нуклеотида від середини відповідного дискримінуючого олігонуклеотида. Дискримінуючі олігонуклеотиди іммобілізують у гелевих елементах, які наносяться на матрицю у вигляді краплин діаметром від 80 до 300мкм з періодом 150-500мкм, без використання спеціальних пристосувань, наприклад, кварцових масок. Як матриця може використовуватися як скляна підкладка (предметне або покривне скло), так і більш доступні матеріали, такі як пластик. Для іммобілізації олігонуклеотидів у біочіпах використовується їх спільна полімеризація з основними компонентами гелю. У результаті цієї одностадійної реакції молекули, які іммобілізуються незворотньо, ковалентно приєднуються до тих або інших мономерів зростаючого полімерного ланцюга й рівномірно розподіляються в повному об‟ємі гелю с високим виходом (порядку 50% для олігонуклеотидів) (Rubina A.Y., Pan‟kov S.V., Dementieva E.I. et al. Hydrogel drop microchips with immobilized DNA: properties and methods for large-scale production. Anal Biochem 2004; 325: 92-106). Концентрація іммобілізованих олігонуклеотидних зондів може бути оцінена зафарбовуванням гелевих елементів біочіпа барвником з низькою специфічністю до нуклеотидної послідовності ДНК (А.Л. Міхєйкін, А.В. Чудінов, А.И. Ярощук, А.Ю. Рубіна, С.В. Паньков, А.С. Крилов, А.С. Засєдатєлєв, А.Д. Мірзабеков. Барвник з низькою специфічністю до нуклеотидної послідовності ДНК: застосування для оцінки кількості олігонуклеотидів, іммобілізованих в комірках біологічних мікрочіпів. Молекулярна біологія 2003; 37(6): 1061-70). ПЦР-продукти, отримані на другій стадії ампліфікації, гібридизують на диференціюючому біочіпі з іммобілізованими олігонуклеотидами, комплементарними до послідовностей досліджуваних на предмет наявності мутацій генів. Гібридизацію проводять у розчині, який містить буферний компонент для підтримки рН, сіль для створення іонної сили й хаотропний (дестабілізуючий водневі зв‟язки) агент, у герметичнії гібридизаційній камері за температури, яка залежить від температури плавлення іммобілізованих на мікрочіпі дискримінуючих олігонуклеотидів. Як дестабілізуючі водневі зв‟язки агенти можуть бути використані, наприклад, гуанідин тіоціанат, сечовина або формамід. Вибір оптимальної температури гібридизації проводять із урахуванням зручності практичного застосування системи. Дискримінуючі олігонуклеотиди, заявлені в даному винаході, мають температуру плавлення в інтервалі від 42 до 44°С, що дозволяє проводити гібридизацію при 37°С з використанням хаотропного агента. Температура 37°С зручна тим, що більшість клінічних лабораторій оснащені термостатами, які підтримують цю температуру. У випадку, якщо розроблювальний метод може бути орієнтований на можливість застосування системи в польових умовах, інтервал використовуваних температур гібридизації може бути розширений до 20-37°С, з відповідною зміною іонної сили гібридизаційного буфера від 0,3 до 1М (Lapa S., Mikheev М., Shchelkunov S. et al., Specieslevel identification of orthopoxviruses with an 17 oligonucleotide microchip. J Clin Microbiol. 2002 Mar; 40 (3) :753-7). Досліджувані фрагменти ДНК утворюють довершені гібрідизаційні дуплекси тільки з відповідними (повністю комплементарними) олігонуклеотидами. З усіма іншими олігонуклеотидами досліджувані фрагменти ДНК дають недовершений дуплекс. Дискримінацію довершених і недовершених дуплексів виконують шляхом порівняння інтенсивностей флуоресценції комірок, у яких утворилися дуплекси. Інтенсивність сигналу в комірці, у якій утворився довершений гібрідизаційний дуплекс (Ідов) вища, ніж у такій, де утворився недовершений дуплекс (Інедов). Проведення гібридизації за оптимальних умов (температура, підібрана концентрація хаотропного агента й іонна сила гібридизаційного буферу) дозволяє досягти співвідношення Ідов/Інедов 2 між двома комірками, які містять зонди, що належать одній групі, і різняться на один нуклеотид. Використовуючи схему розташування олігонуклеотидів на біочіпі, визначають мутації, властиві досліджуваній послідовності ДНК, і роблять висновок про стійкість (або чутливість) досліджуваного зразка до рифампіцину й/або ізоніазиду. Інтенсивності флуоресцентних сигналів в комірках, які належать одній групі, порівнюють між собою. Максимальний сигнал флуоресценції свідчить про утворення довершеного гібридизаційного дуплекса в комірці, де даний сигнал зареєстрований. Якщо максимальний сигнал зареєстрований в комірці, яка відповідає ДНК без мутацій (тобто, тій, що належить мікроорганізму, чутливому до лікарського препарату), то вважають, що за даною амінокислотною позицією (групою комірок) досліджуваний зразок мутацій не має. Якщо максимальний сигнал зареєстрований в комірці, яка відповідає ДНК із мутацією (мутаціями) (тобто, тій, що належить мікроорганізму, стійкому до лікарського препарату), то вважають, що за даною амінокислотною позицією (групою комірок) досліджуваний зразок має амінокислотну заміну, що приводить до виникнення резистентності. Досліджуваний зразок ДНК визнається таким, що належить до чутливого штаму мікобактерій, якщо за кожною амінокислотною позицією (групою комірок) зразок охарактеризований як чутливий. Досліджуваний зразок ДНК визнається таким, що належить до стійкого штаму мікобактерій, якщо, як мінімум, за однією з амінокислотних позицій (групі комірок) зразок охарактеризований як такий, що має мутацію, яка приводить до виникнення резистентності. Для таких зразків додатково з‟ясовують тип препарату, до якого виявлена стійкість, установивши групу, за якою зразок віднесений до резистентного типу. Окрему групу із двох комірок утворює система для виявлення ДНК мікобактерій туберкульозного комплексу. Інтерпретація результату в даній системі ґрунтується на реєстрації більш інтенсивного флуоресцентного сигналу в детектуючих комірках відносно комірок порівняння. Як комірки порівняння використовуються комірки гелю, які не містять іммобілізованих олігонуклеотидів. Якщо в детектуючих комірках спостерігаються більш інтенсивні сигнали, то робиться висновок, що в досліджува 92720 18 ному зразку виявлена ДНК, яка належить мікобактеріям туберкульозного комплексу. Далі винахід буде проілюстровано прикладами, які покликанні забезпечити краще розуміння сутності заявленого винаходу, але не повинні розглядатися як обмежуючі даний винахід. Приклади. Приклад 1. Біочіп для виявлення мікобактерій туберкульозного комплексу й визначення їх чутливості до рифампіцину й ізоніазиду. 1. Олігонуклеотиди для іммобілізації на біочіпі й праймери для ампліфікації були синтезовані на автоматичному синтезаторі 394 DNA/RNA synthesizer (Applied Biosystems, США) і містили спейсер з вільної аміногрупою 3‟-Amino-Modifier C7 CPG 500 (Glen Research, США) для наступної іммобілізації в гель або 5‟-Amino-Modifier С6 (Glen Research, США) для введення флуоресцентної мітки, відповідно. Введення індодикарбоціанінової флуоресцентної мітки («Биочип-ИМБ», Росія) здійснювали відповідно до рекомендацій виробника. Виготовлення біочипів проводили відповідно до процедури, описаної раніше (Rubina AY, Pan‟kov SV, Dementieva El et al. Hydrogel drop microchips with immobilized DNA: properties and methods for large-scale production. Anal Biochem 2004; 325: 92106). Біочіпи містили напівсферичні комірки діаметром 100мкм і були віддалені одні від одних на відстань 300мкм. Рівномірність розміщення комірок і їх діаметр оцінювали за допомогою програмного забезпечення „Тест-чіп‟ («Биочип-ИМБ», Росія). Контроль якості мікрочіпів виконували вимірюванням концентрацій іммобілізованих олігонуклеотидів. Біочіпи забарвлювали флуоресцентним барвником ImD-310, концентрацію іммобілізованих зондів оцінювали так, як це було описано раніше (А.Л. Міхейкін, А.В. Чудінов, А.І. Ярощук, А.Ю. Рубіна, С.В. Паньков, А.С. Крилов, А.С Засєдатєлєв, А.Д. Мірзабеков. Барвник з низькою специфічністю до нуклеотидної послідовності ДНК: застосування для оцінки кількості олігонуклеотидів, іммобілізованих в комірках біологічних мікрочіпів. Молекулярна біологія 2003; 37(6): 1061-70). Структура біочіпа Біочіп містить 69 іммобілізованих олігонуклеотидів, список яких представлений у таблиці 1, три маркерні точки для правильного позиційонування (захоплення зображення), що виконується комп‟ютерною програмою, і 7 резервних комірок порожнього гелю. Розташування іммобілізованих на мікрочіпі олігонуклеотидів показано на Фіг.1. Сірим кольором на Фіг.1 позначені гелеві комірки, які містять олігонуклеотиди (Таблиця 1, SEQ ID NO 1-69), здатні формувати довершений гібридизаційний дуплекс із ДНК, що не має мутацій (тобто із ДНК дикого типу (WT)), у позиціях, які відповідають наступним амінокислотним залишкам або нуклеотидам у промоторній області генів: rpoB: (Gly/Thr)WT507(A1); Met515(B1); Ser522(C1); Leu533(D1); Asp516(A3); His(B3, C3); Ser531(D3); Leu511(E3); Gln513 (A8); Ser512 (E8); katG: Ser315(El,Fl); Trp328 (Gl); Ile335 (G5); inhA: inhAw_G24(G8); inhAw_AT8 (H8); inhAw_C15(H5); 19 ahpC: ahpCw_G6(H1); ahpCw_C10(I1); ahpCw_G9(І4); ahpCw_C12 (І6). Білим кольором на Фіг.1 позначені гелеві комірки, які містять олігонуклеотиди, що формують недовершені гібрідизаційні дуплекси із ДНК дикого типу, у позиціях, які відповідають наступним амінокислотним залишкам або нуклеотидам у промоторній області генів: 92720 20 rpoB: WT507(A2); Ile515 (B2); Leu522 (C2); Pro533 (D2); VA1516 (А4); Tyr516 (A5); Gly516 (А6); Glu516 (A7); Asp526 (В4); Leu526 (В5); Gln526 (В6); Cys526 (В7); Туr526 (С4); Asn526 (С5); Arg526 (С6); Рrо526 (С7); Leu531 (D4); Тгр531 (D5); Cys531 (D6); Gln531 (D7); Pro511 (E4); Arg511 (E5); Thr512 (E7); Arg512 (E6); Leu513 (B8); Lys513 (C8); Gly513(D8). 21 katG: Thr3151 (E2); Thr3152 (F2); Asn315 (F3); Ile315 (F4); Arg3151 (F5); Arg3152 (F6); Gly315 (F7); Gly328 (G2); Leu328 (G3); Cys328 (G4); Val335 (G6); inhA: inhA_T24 (G7); inhA_A8 (H7); inhA_G8 (H6); inhA_T15 (H4); inhA_G16 (H3); ahpC: ahpC_A6 (H2); ahpC_T10 (12); ahpC_A10 (13); ahpC_A9 (15); ahpC_T12 (17). Комірки F8 і 18 містять олігонуклеотид, комплементарний до фрагмента послідовності інсерційного елемента IS6110, який, як правило, є присутнім у ДНК мікобактерій туберкульозного комплексу. 92720 22 Приклад 2. Обробка клінічного зразка 1. Клінічний зразок (мокротиння, екссудат, змивки, бронхо-альвеолярний лаваж) змішували в співвідношенні 1:1 за об‟ємом із свіжеприготованим 0,5% розчином N-ацетил-L- цистеіну (NALC) в 2% NaOH. Зразок ретельно перемішували на вортексі й витримували за кімнатної температури протягом 20хв. До зразка додавали фосфатний буфер рН6,8 у співвідношенні 1:5 за об‟ємом й центрифугували протягом 30хв. при 3000об./хв. При використанні спинномозкової рідини проводили попереднє центрифугування протягом 10хв. при 10000об./хв. При аналізі крові попередньо виділяли лімфоцитарну фракцію за загальноприйнятою 23 методикою з використанням фіколу. Подальша обробка зразків проводилася однаково. 2. Осад клітин суспендували в 1,5мл ТЕ буфера (10мм Тріс-НСI, 1мм ЕДТА), рН8,0, і осаджували при 3000об./хв. протягом 30хв. Відмивання повторювали ще раз. 3. До отриманого осаду додавали 30мкл ТЕ буфера, рН8,0, який містив 1% (об./об.) Тритон Х100, і витримували в сухому термостаті при 95°С протягом 30хв. 4. Зразок центрифугували при 10000об./хв протягом 10хв., надосадову рідину (3мкл) використовували для проведення ПЦР. Приклад 3. Ампліфікація фрагментів мобільного елемента IS6110, генів rpoB, katG, inhA, ahpC; одержання одноланцюгових флуоресцентно мічених фрагментів методом мультиплексної ПЦР. На першій стадії проводили мультиплексну ампліфікацію фрагментів генів rроВ (212п.о.), katG (166п.о.), inhA (133п.о.), ahpC (126п.о.) і інсерційного елемента IS6110 (309п.о). В 25мкл ПЦР-суміші вносили 3мкл зразка, отриманого в п.4 Приклада 2. Склад ПЦР-суміші: - 1Х ПЦР-буфер: 10мМ KC1, 10мМ Тріс-НСl (рН8,3) (Сілєкс, Росія); - 1,5мМ МgСl2; - 200мкМ кожного dATP, dCTP, dGTP, dUTP (Сілєкс); - Суміш праймерів (послідовності представлені в Таблиці 2) у наступній концентрації: p105f, p293r, katG_f, katG_r1, IS_f, IS_r1; InhA_f, InhA_rl, ahpc_f, ahpC_r1 - 100нM; - 5од. термостабільної Taq ДНК-полімерази (Сілєкс); - 0,5од. урацил-ДНК-глікозилази (Сілєкс). Ампліфікацію проводили на програмувальному термостаті MiniCycler (MJ Research, США) з наступним режимом: 95°С - 30с, 67°С - 30с, 72°С 30с; 36 циклів (у першому циклі час денатурації збільшували до 5хв., в останньому - час добудовування до 5хв.). 1мкл реакційної суміші, отриманої після першої стадії ПЦР, використовували як матрицю при проведенні другої стадії. 92720 24 Другу стадію ПЦР проводили в із праймерами, специфічними до фрагмента гена rроВ (126п.о.), katG (140п.о.), inhA (93п.о.), ahpC (96п.о.), і IS6110 (110п.о.). Склад ПЦР-Суміші (50мкл): - 1Х ПЦР - буфер: 10мМ КС1, 10мМ Тріс-НСІ (рН 8,3) (СілєкС, Росія); - 1,5м МgСl2; - 200мкМ кожного dNTP (Сілєкс); - Суміш праймерів (послідовності представлені в Таблиці 2) у наступній концентрації: p1272f*/p1398r, 100нМ; katG_f/katG_r2*, 100нМ; inhA_f/inhA_r2*, 100нМ; ahpC_f/ahpC_r2*, 100нМ; IS_f/IS_r2*, 100нМ; - 10од. термостабільної Taq ДНК-полімерази (Сілєкс). 5‟-кінець праймерів, відзначених індексом*, містив лінкер С6 aminomodifier (Glen Research Corp., США), до аміногрупи якого було приєднано індодикарбоціаніновий барвник («Биочип-ИМБ», Росія). Ампліфікацію проводили на програмувальному термостаті MiniCycler (MJ Research, США) з наступним режимом: 95°С - 30с, 65°С - 30с, 72°С 20с; 46 циклів (у першому циклі час денатурації збільшували до 5хв., в останньому - час добудовування до 5хв.). 12мкл отриманого продукту використовувалися в гібридизації на біочіпі. Приклад 4. Гібридизація ампліфікованих мічених продуктів на біочіпі Дo 12мкл реакційної суміші, отриманої після другої стадії ПЦР, яка містила переважно одноланцюгові флуоресцентно мічені фрагменти ДНК, що відповідали п‟яти аналізованим фрагментам, додавали концентрований розчин гібридизаційного буфера так, щоб кінцева концентрація гуанідину тіоціанату становила 1М, HEPES - 50мМ, рН7,5, ЕДТА - 5мМ. Отриману суміш (28мкл) поміщували в гібридизаційную камеру мікрочіпа. Камери були виготовлені ТОВ «Биочип-ИМБ». Гібридизацію проводили при 37°С протягом 6-12 годин. По закінченні гібридизації мікрочіп тричі промивали дистильованою водою при 37°С. 25 Приклад 5. Реєстрація й інтерпретація результатів гібридизації Реєстрація флуоресцентного зображення біочіпа виконувалася на портативному флуоресцентному аналізаторі (ТОВ «Биочип-ИМБ», Росія) з використанням програмного забезпечення ImageWare®, розробленого тією ж компанією. Інтерпретацію результатів проводили в такий спосіб. Гелеві комірки, які містять олігонуклеотиди, об‟єднані в 22 групи (розмежовані на Фіг.1 товстими лініями) таким чином, що порівняння інтенсивностей флуоресцентних сигналів комірок усередині кожної групи дозволяє зробити висновок про наявність/відсутність мутації (мінорного поліморфізму), що приводить до заміни одного амінокислотного залишку або делеції, або заміни нуклеотида в промоторній області генів inhA і ahpC. Приклад 6. Визначення в клінічному зразку мікобактерій туберкульозного комплексу, чутливих до рифампіцину й ізоніазиду (ДНК дикого типу), методом гібридизації на біочіпі. Зразок мокротиння, отриманий від пацієнта, з підтвердженою мікроскопією наявністю кислотостійких бактерій (БК+), був розділений на 2 частини, одна йз яких після деконтамінації (N-ацетил-Lцистеін і NaOH) і нейтралізації була посіяна на середовище Левенштейна-Йенсена. Зразок інкубували при 37°С протягом б тижнів із щотижневою перевіркою утворення бактеріальних колоній. При виявленні колоній присутність кислотостійких бактерій у зразку виявляли мікроскопією з фарбуванням за Цілем-Нільсеном. Ідентифікацію мікобактерій туберкульозного комплексу проводили за результатами біохімічних тестів (Kent PT, Kubica GP. Public health mycobacteriology. A guide for level III laboratory. Atlanta, GA: Centers for Disease Control and Prevention, 1985). Тести на стійкість до ліків проводили методом пропорцій на середовищах Левенштейна-Йенсена, які містять рифампіцин і ізоніазид у концентраціях 40мг/л і 0,2мг/л, відповідно. Ізолят вважався стійким, якщо на середовищі із препаратом виростали колонії, які становили більше 1% від числа тих колоній, що виросли на середовищі без препарату. Друга частина зразка мокротиння використовувалась для аналізу способом запропонованим винаходом, як описано в Прикладах 2-5. На Фіг.2 представлена картина гібридизації зразка ДНК, виділеного із чутливого до рифампіцину й ізоніазиду штаму мікобактерій туберкульозного комплексу. Відповідно до запропонованого алгоритму інтерпретації результатів, комірки з олігонуклеоти 92720 26 дами, комплементарними до ДНК дикого типу, мають більшу інтенсивність флуоресцентного сигналу, ніж інші комірки всередині кожної групи. Таким чином, ДНК досліджуваного об‟єкта утворює довершені гібрідизаційні дуплекси з олігонуклеотидами, комплементарними до послідовності ДНК дикого типу (чутливого до рифампіцину й ізоніазиду штаму мікобактерій туберкульозу). Приналежність даного штаму до мікобактерій туберкульозного комплексу встановлюється шляхом порівняння інтенсивності флуоресцентного сигналу в комірках з іммобілізованим олігонуклеотидом на IS6110 (F8, 18) і в елементах порожнього гелю, які виконують роль негативного контролю. Із цього роблять висновок, що у вивченому клінічному зразку присутні мікобактерії туберкульозного комплексу, причому стійких до рифампіцину й ізоніазиду мікобактерій не виявлено. Методом культивування на твердих середовищах і біхімічними тестами була підтверджена наявність М. tuberculosis у зразку мокротиння. Ріст колоній на середовищах з хіміопрепаратом був відсутній. Таким чином, культуральний спосіб зафіксував наявність у клінічному зразку М. tuberculosis дикого типу, що повністю збігається з результатами, отриманими способом даного винаходу. Приклад 7. Визначення в клінічному зразку мікобактерій туберкульозного комплексу, резистентних до рифампіцину й ізоніазиду (ДНК, що містить мутації), методом гібридизації на біочіпі. Зразок мокротиння, отриманий від пацієнта, з підтвердженою мікроскопією наявністю кислотостійких бактерій (БК+), був розділений на 2 частини, одна із яких після деконтамінації (N-ацетил-Lцистеін і NaOH) і нейтралізації була посіяна на середовище Левенштейна-Йенсена. Зразок інкубували при 37°С протягом 6 тижнів із щотижневою перевіркою утворення бактеріальних колоній. При виявленні колоній присутність кислотостійких бактерій у зразку виявляли мікроскопією з фарбуванням за Цілем-Нільсеном. Ідентифікацію мікобактерій туберкульозного комплексу проводили за результатами біохімічних тестів (Kent PT, Kubica GP. Public health mycobacteriology. A guide for level III laboratory. Atlanta, GA: Centers for Disease Control and Prevention, 1985). Тести на стійкість до ліків проводили методом пропорцій на середовищах Левенштейна-Йенсена, що містять рифампіцин і ізоніазид у концентраціях 40мг/л і 0,2мг/л, відповідно. Ізолят вважався стійким, якщо на середовищі із препаратом виростали колонії, які ста 27 новили більше 1% від числа тих колоній, що виросли на середовищі без препарату. Друга частина зразка мокротиння використовувалась для аналізу способом запропонованим винаходом, як описано в Прикладах 2-5. На Фіг.3 представлена картина гібридизації зразка ДНК, виділеного з резистентного до рифампіцину й ізоніазиду штаму мікобактерій туберкульозного комплексу. У кожній із груп елементів біочіпа інтенсивність флуоресцентного сигналу в комірці з олігонуклеотидом, комплементарним до ДНК дикого типу, вища інтенсивності сигналу інших комірок. Виключення становлять групи елементів D3-D7 і Е1, Е2, F1-F7 (Фіг.3). Максимальну інтенсивність флуоресценції всередині групи D3-D7 має елемент D4. Отже, ДНК досліджуваного об‟єкта має точкову нуклеотидну заміну А>Т у положенні 2431 гена rроВ (Genbank Ace.No L27989), що приводить до заміни амінокислоти Ser на Leu у позиції №531 і веде до виникнення резистентності досліджуваного штаму до рифампіцину. Максимальну інтенсивність флуоресценції всередині групи Е1, Е2, F1-F7 має елемент Е2. У такий спосіб визначається наявність у досліджуваній ДНК нуклеотидної заміни G на С у положенні 1013 гена katG (Genbank Ace.No U06262), що приводить до заміни амінокислоти Ser 315 на Thr у позиції №315 і веде до виникнення резистентності досліджуваного штаму до ізоніазиду. Приналежність даного штаму до мікобактерій туберкульозного комплексу встановлюється шляхом порівняння інтенсивності флуоресцентного сигналу в комірках з іммобілізованим олігонуклеотидом на IS6110 (F8, 18) і в елементах порожнього гелю, які виконують роль негативного контролю. Таким чином, робиться висновок, що у вивченому клінічному зразку виявлені мікобактерії туберкульозного комплексу, резистентні до рифампіцину й ізоніазиду. Резистентність до рифампіцину обумовлена заміною амінокислоти Ser на Leu у позиції №531 гена кроВ. Стійкість до ізоніазиду визначається мутацією Ser315>Leu у гені katG. Методом культивування на твердих середовищах і біохімічними тестами була підтверджена наявність М. tuberculosis у зразку мокротиння. Стійкий ріст колоній на середовищах з рифампіцином та ізоніазидом підтвердив наявність у зразку М. tuberculosis, стійкого до вищевказаних препаратів, що повністю збігається з результатами, отриманими способом запропонованим даним винаходом. Приклад 8. Визначення в клінічному зразку мікобактерій туберкульозного комплексу, резистентних до рифампіцину й ізоніазиду (ДНК, що містить мутації), методом гібридизації на біочіпі. Зразок мокротиння, отриманий від пацієнта, з підтвердженою мікроскопією наявністю кислотостійких бактерій (БК+), був розділений на 2 частини, одна із яких після деконтамінації (N-ацетил-Lцистеін і NaOH) і нейтралізації була посіяна на середовище Левенштейна-Йенсена. Зразок інкубували при 37°С протягом б тижнів із щотижневою перевіркою утворення бактеріальних колоній. При 92720 28 виявленні колоній присутність кислотостійких бактерій у зразку виявляли мікроскопією з фарбуванням за Цілем-Нільсеном. Ідентифікацію мікобактерій туберкульозного комплексу проводили за результатами біохімічних тестів (Kent PT, Kubica GP. Public health mycobacteriology. A guide for level III laboratory. Atlanta, GA: Centers for Disease Control and Prevention, 1985). Тести на стійкість до ліків проводили методом пропорцій на середовищах Левенштейна-Йенсена, що містять рифампіцин і ізоніазид у концентраціях 40мг/л і 0,2мг/л, відповідно. Ізолят уважався стійким, якщо на середовищі із препаратом виростали колонії, які становили більше 1% від числа тих колоній, що виросли на середовищі без препарату. Друга частина зразка мокротиння використовувалась для аналізу способом запропонованим винаходом, як описано в Прикладах 2-5. На Фіг.4 представлена картина гібридизації зразка ДНК, виділеного з резистентного до рифампіцину й ізоніазиду штаму мікобактерій туберкульозного комплексу. У кожній із груп елементів біочіпа інтенсивність флуоресцентного сигналу в комірці з олігонуклеотидом, комплементарним до ДНК дикого типу вище інтенсивності сигналу інших комірок. Виключення становлять групи елементів А3-А7 і Н3-Н5 (Фіг.4). Максимальну інтенсивність флуоресценції усередині групи А3-А7 має елемент А4. Отже, ДНК досліджуваного об‟єкта має точкову нуклеотидну заміну OG у положенні 2384 гена rроВ (Genbank Ace.No L27989), що приводить до заміни амінокислоти Asp на Val у позиції №516 і веде до виникнення резистентності досліджуваного штаму до рифампіцину. Максимальну інтенсивність флуоресценції усередині групи Н3-Н5 має елемент Н4. Отже, ДНК досліджуваного об‟єкта має нуклеотидну заміну C>Т у положенні -15 відносно сайту ініціації трансляції в промоторній області гена inhA. Приналежність даного штаму до мікобактерій туберкульозного комплексу встановлюється шляхом порівняння інтенсивності флуоресцентного сигналу в комірках з іммобілізованим олігонуклеотидом на IS6110 (F8, І8) і в елементах порожнього гелю, які виконують роль негативного контролю. У такий спосіб робиться висновок, що у дослідному клінічному зразку виявлені мікобактерії туберкульозного комплексу, резистентні до рифампіцину й ізоніазиду. Резистентність до рифампіцину обумовлена заміною амінокислоти Asp на Val у позиції №516 гена rроВ. Стійкість до ізоніазиду визначається нуклеотидними замінами C>Т (-15) у промоторній області гена inhA. Методом культивування на твердих середовищах і біохімічними тестами була підтверджена наявність М. tuberculosis у зразку мокротиння. Стійкий ріст колоній на середовищах з рифампіцином і ізоніазидом підтвердив наявність у зразку М. tuberculosis, стійкого до вищевказаних препаратів, що повністю збігається з результатами, отриманими способом запропонованим даним винаходом. 29 Приклад 9. Виявлення ДНК, виділеної зі штаму М. bovis BCG, чутливого до рифампіцину й ізоніазиду, методом гібридизації на біочіпі. Культура охарактеризованого штаму М. bovis BCG, який належить до мікобактерій туберкульозного комплексу, була проаналізована способом запропонованим даним винаходом, як описано в Прикладах 2-5. На Фіг.5 представлена картина гібридизації зразка ДНК, виділеного із чутливого до рифампіцину й ізоніазиду штаму мікобактерій туберкульозного комплексу. Відповідно до запропонованого алгоритму інтерпретації результатів, комірки з олігонуклеотидами, комплементарними до ДНК дикого типу, мають більшу інтенсивність флуоресцентного сигналу, ніж інші комірки всередині кожної групи. Таким чином, ДНК досліджуваного об‟єкта утворює довершені гібрідизаційні дуплекси з олігонуклеотидами, комплементарними до послідовності ДНК дикого типу (чутливого до рифампіцину й ізоніазиду штаму мікобактерій туберкульозного комплексу). Належність даного штаму до мікобактерій туберкульозного комплексу встановлюється шляхом порівняння інтенсивності флуоресцентного сигналу в комірках з іммобілізованим олігонуклеотидом на IS6110 (F8, I8) і в елементах порожнього гелю, що виконують роль негативного контролю. Із цього можна зробити висновок, що аналізована ДНК відноситься до мікобактерій туберкульозного комплексу, чутливих до рифампіцину й ізоніазиду. При цьому гібридизаційна картина ДНК штаму М. bovis BCG збігається з гібридизаційною картиною штаму М. tuberculosis дикого типу. Приклад 10. Аналіз ДНК, виділеної зі штаму М. avium S58 методом гібридизації на біочіпі. Культура охарактеризованого штаму М. avium S58, який не належить до мікобактерій туберкульозного комплексу, була проаналізована способом запропонованим даним винаходом, як описано в Прикладах 2-5. 92720 30 На Фіг.6 представлена картина гібридизації зразка ДНК, виділеного зі штаму, що не належить до мікобактерій туберкульозного комплексу. Відповідно до запропонованого алгоритму інтерпретації результатів, належність аналізованого штаму до мікобактерій туберкульозного комплексу встановлюється шляхом порівняння інтенсивності флуоресцентного сигналу в комірках з іммобілізованим олігонуклеотидом на IS6110 (F8, l8) і в елементах порожнього гелю, які виконують роль негативного контролю. Як видно з Фіг.6, в отриманій гібридизаційній картині сигнали в комірках F8 і I8 відсутні. Це означає, що геном досліджуваного мікроорганізму не містить інсерційного елемента IS6110. Отже, аналізована ДНК виділена з мікроорганізму, який не належить до мікобактерій туберкульозного комплексу, а отже, аналіз стійкості до ліків не має сенсу. Таким чином, представлений винахід дозволяє в короткий термін визначати стійкі до рифампіцину й ізоніазиду форми мікобактерій туберкульозу, безпосередньо використовуючи матеріал клінічного зразка. Спосіб даного винаходу вигідно відрізняється від існуючих на даний час аналогів простотою виконання й низькою собівартістю. Оптимізована двостадійна мультиплексна ПЦР із використанням оригінального набору праймерів дозволяє досягти високої чутливості методу (не менше 500 геном-еквівалентів мішені на зразок). Застосування біочіпа, який містить оригінальний набір диференціюючих олігонуклеотидів, дозволяє також виконувати типування (маркування) мікобактерій туберкульозу на генотипічному рівні. Хоча найкращі втілення даного винаходу та їхні переваги було докладно описано вище, фахівець у даній галузі зможе внести різні зміни, доповнення або, навпаки, щось опустити, не виходячи при цьому за рамки даного винаходу, які визначаються прикладеною нижче формулою винаходу. 31 92720 32 33 92720 34 35 92720 36 37 92720 38 39 92720 40 41 92720 42 43 92720 44 45 92720 46 47 92720 48 49 92720 50 51 92720 52 53 92720 54 55 92720 56 57 92720 58 59 92720 60
ДивитисяДодаткова інформація
Назва патенту англійськоюMethod for simulteneous identification of mycobacteria of tuberculosis complex and identification of mutations of dna of mycobacteria, leading to resistance of microorganisms towards rifampicin and isoniazid, on biological microchips, set of primers, biochip, and set of oligonucleotide probes, used in a method
Автори англійськоюZasiedatieliev Alieksandr Sierhieievich, Soboliev Alieksandr Yurievich, Hriadunov Dmitrii Alieksandrovuch, Lapa Sierhiei Anatolievich, Mikhailovich Vladimir Mikhailovich, Mirzabiekov Andriei Darievich
Назва патенту російськоюСпособ одновременного обнаружения микобактерий туберкулезного комплекса и идентификации мутаций днк микобактерий, которые приводят к стойкости микроорганизмов к рифампицину и изониазиду, на биологических микрочипах, набор праймеров, биочип и набор олигонуклеотидных зондив, которые применяются в способе
Автори російськоюЗаседателев Александр Сергеевич, Соболев Александр Юрьевич, Грядунов Дмитрий Александрович, Лапа Сергей Анатольевич, Михайлович Владимир Михайлович, Мирзабеков Андрей Дарьевич
МПК / Мітки
МПК: C12Q 1/68, C12R 1/32, C07H 21/00
Мітки: мікрочипах, рифампіцину, ізоніазиду, виявлення, днк, мікроорганізмів, одночасного, ідентифікації, мутацій, призводять, туберкульозного, комплексу, спосіб, стійкості, біологічних, мікобактерій
Код посилання
<a href="https://ua.patents.su/40-92720-sposib-odnochasnogo-viyavlennya-mikobakterijj-tuberkuloznogo-kompleksu-ta-identifikaci-mutacijj-dnk-mikobakterijj-shho-prizvodyat-do-stijjkosti-mikroorganizmiv-do-rifampicinu-ta-iz.html" target="_blank" rel="follow" title="База патентів України">Спосіб одночасного виявлення мікобактерій туберкульозного комплексу та ідентифікації мутацій днк мікобактерій, що призводять до стійкості мікроорганізмів до рифампіцину та ізоніазиду, на біологічних мікрочипах,</a>
Попередній патент: Форма з випуском газу
Випадковий патент: Адаптивний супутниковий спектрофотометр