Спосіб визначення днк культури lactococcus lactis subsp. cremoris методом полімеразної ланцюгової реакції
Номер патенту: 107547
Опубліковано: 12.01.2015
Автори: Семенівська Олена Анатоліївна, Вакуленко Микола Михайлович, Малова Валерія Всеволодівна
Формула / Реферат
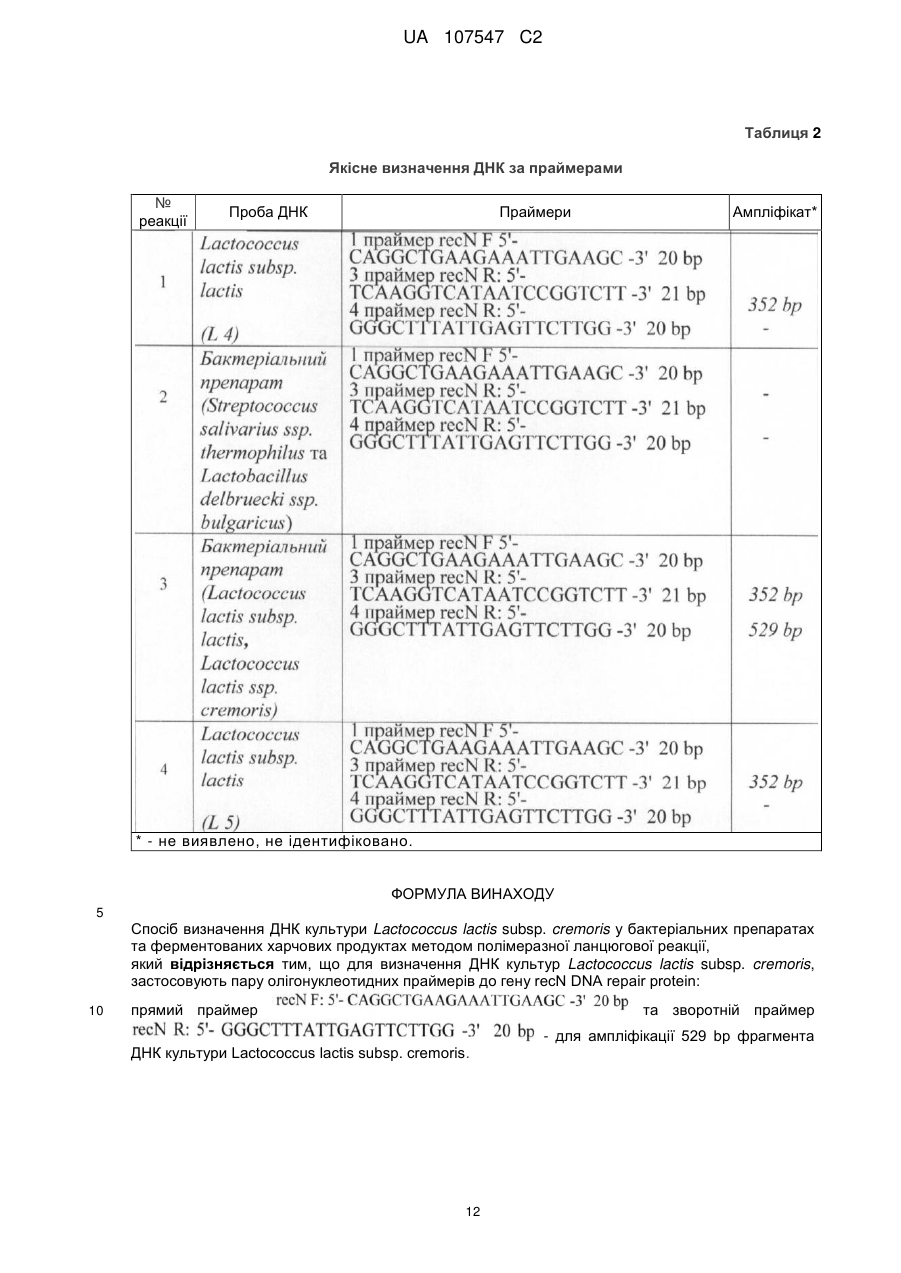
Спосіб визначення ДНК культури Lactococcus lactis subsp. cremoris у бактеріальних препаратах та ферментованих харчових продуктах методом полімеразної ланцюгової реакції,
який відрізняється тим, що для визначення ДНК культур Lactococcus lactis subsp. cremoris, застосовують пару олігонуклеотидних праймерів до гену recN DNA repair protein:
прямий праймер ![]() та зворотній праймер
та зворотній праймер ![]() - для ампліфікації 529 bp фрагмента ДНК культури Lactococcus lactis subsp. cremoris.
- для ампліфікації 529 bp фрагмента ДНК культури Lactococcus lactis subsp. cremoris.
Текст
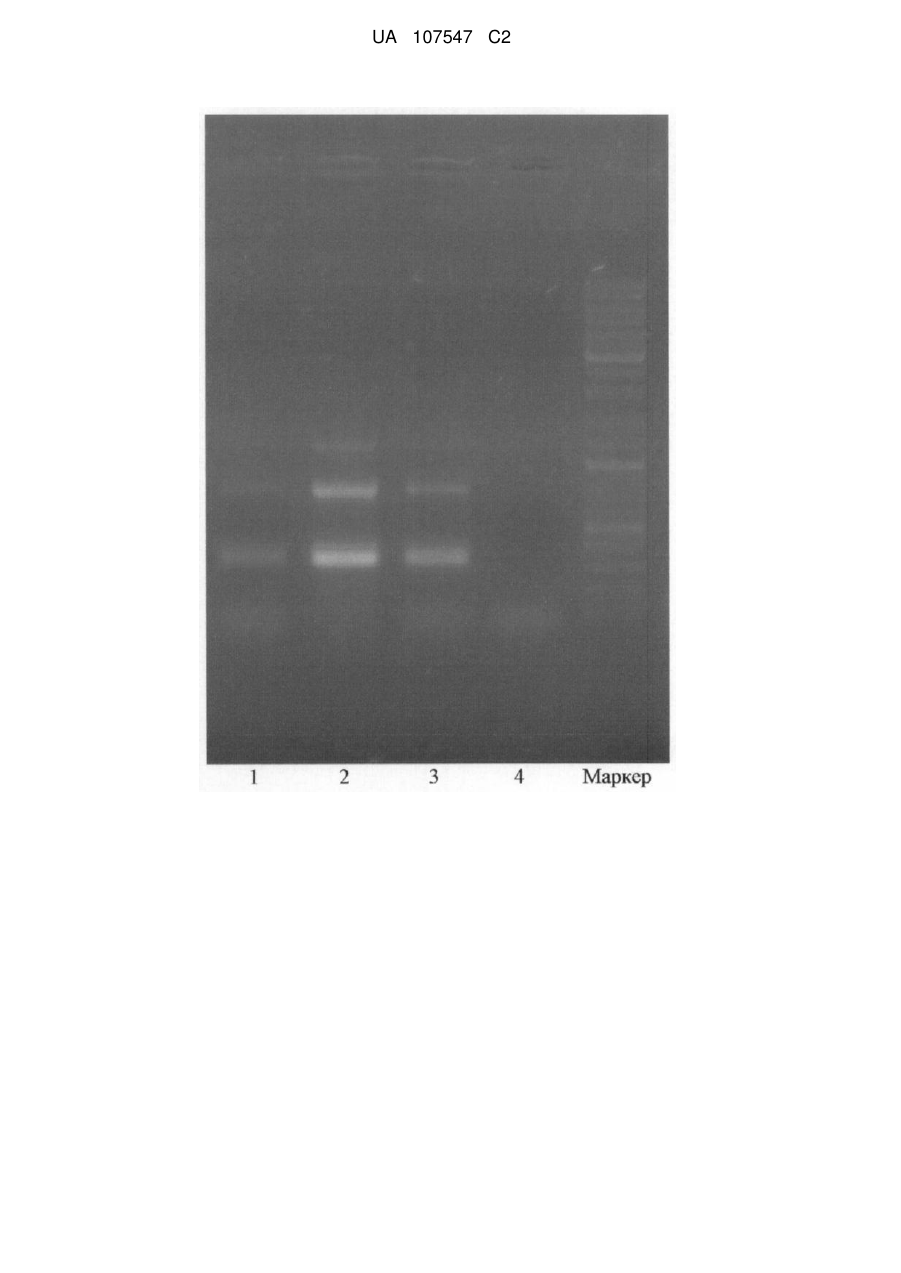
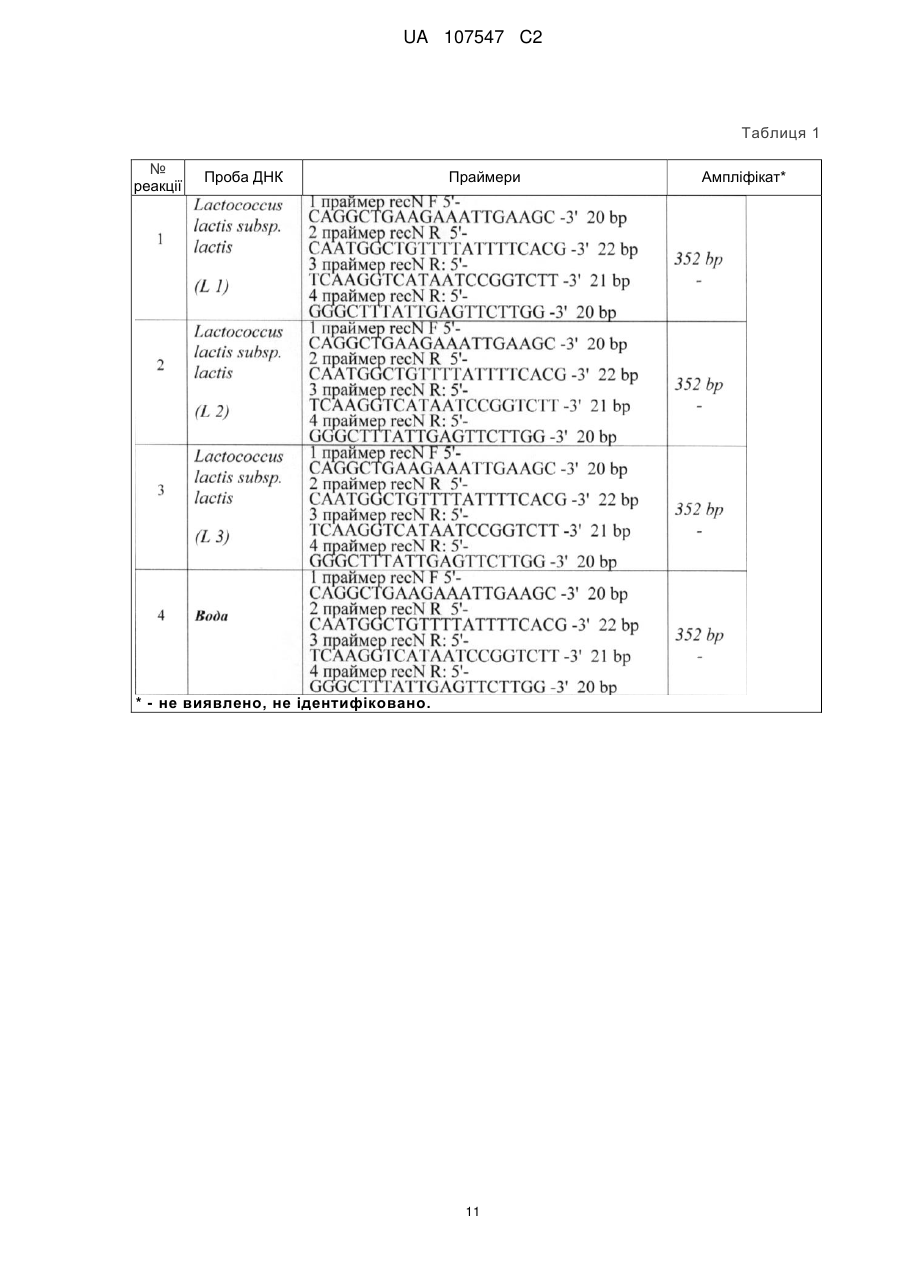
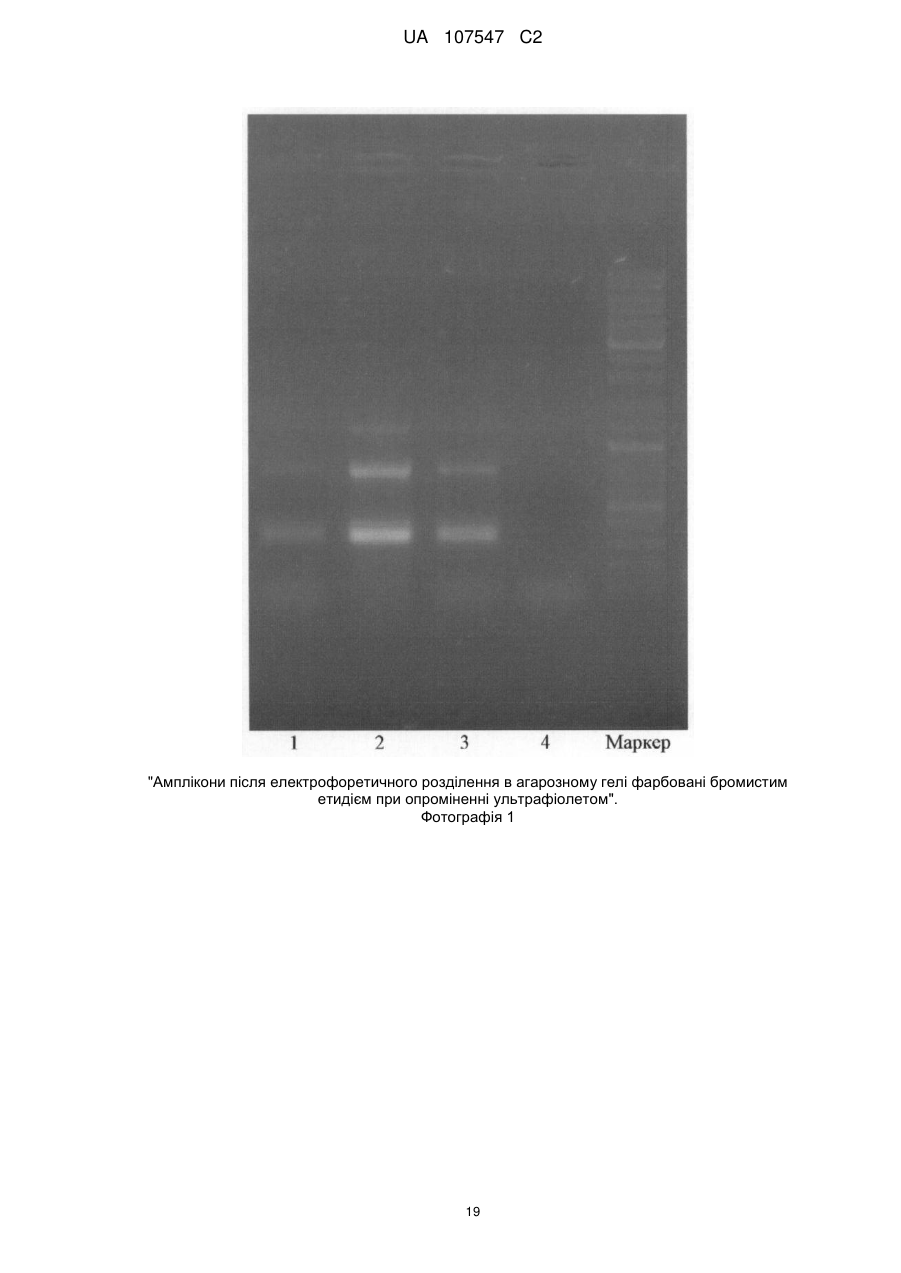
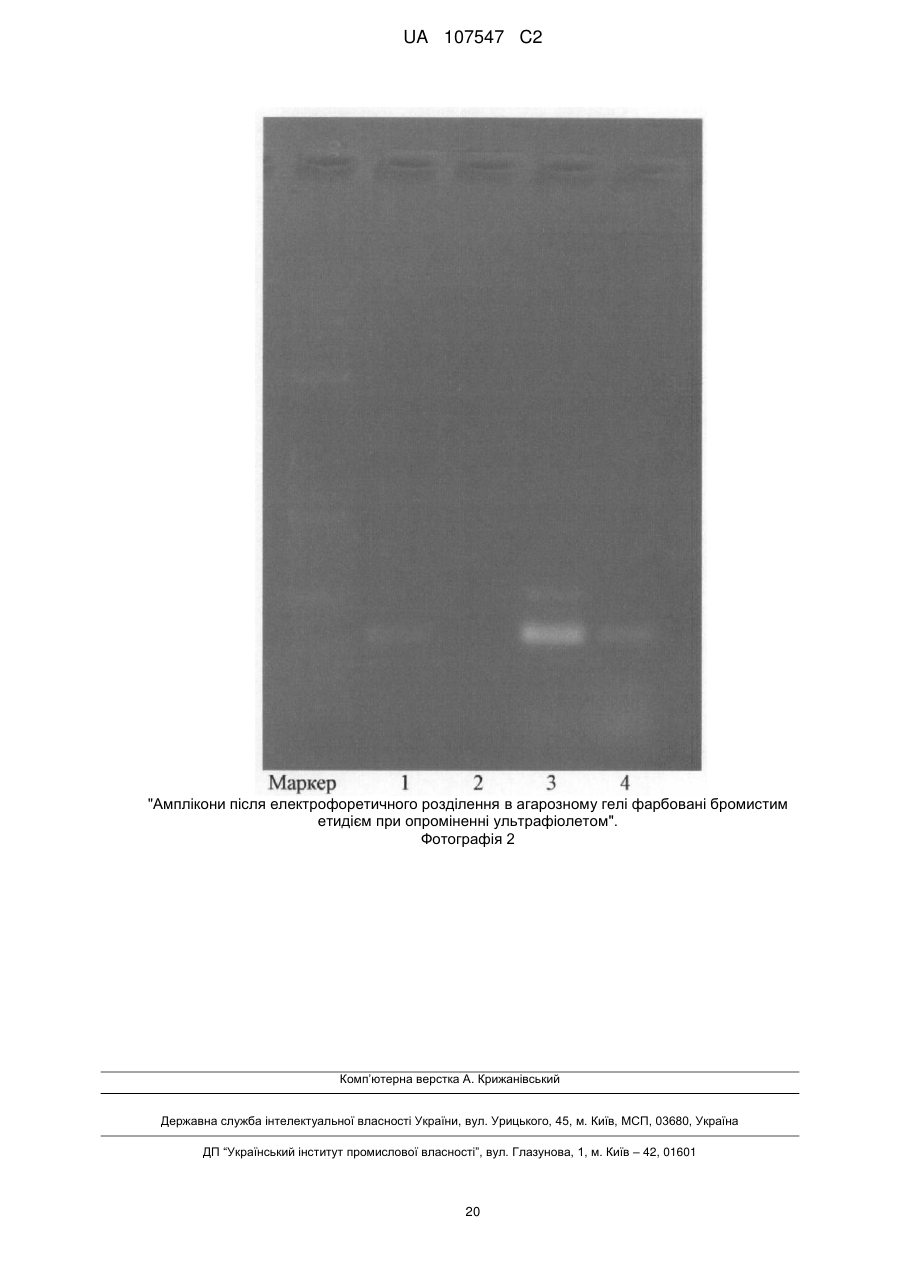
Реферат: Винахід належить до способу визначення ДНК культури Lactococcus lactis subsp. cremoris у бактеріальних препаратах та ферментованих харчових продуктах методом полімеразної ланцюгової реакції. UA 107547 C2 (12) UA 107547 C2 UA 107547 C2 5 10 15 20 25 30 35 40 45 50 55 60 До недавнього часу при відборі штамів для створення бактеріальних заквасок використовувалися лише стандартні мікробіологічні підходи, такі як виділення культур, ідентифікація їх таксономічного положення на основі вивчення морфологічних, фізіологобіохімічних властивостей, визначення умов культивування. Однак, застосування тільки цих традиційних підходів не завжди дозволяє ефективно відбирати безпечні і технологічні штами молочнокислих бактерій для виробництва бактеріальних препаратів та ферментованих харчових продуктів. Альтернативним методом, заснованим на детекції та ідентифікації послідовностей нуклеїнових кислот є аналіз їх ДНК з використанням полімеразної ланцюгової реакції, скорочено - ПЛР. Полімеразна ланцюгова реакція дозволяє ефективно проводити ідентифікацію та детекцію видоспецифічних генів культур за допомогою підібраних праймерів. Праймери використовують у молекулярній біології та біотехнології для ідентифікації та детекції видоспецифічних генів. Праймери - синтетичні олігонуклеотиди - короткі фрагменти нуклеїнової кислоти комплементарні необхідні послідовностям ДНК або РНК, які ініціюють синтез комплементарного ланцюга за участі ДНК-полімерази, а також під час реплікації ДНК. Праймери для ініціації синтезу ДНК-полімеразою нового ланцюга починаючи з 3'-кінця праймера. Праймерів має бути два - прямий та зворотній, які обмежують ділянку ДНК, що піддається ампліфікації. Полімеразна ланцюгова реакція, скорочено - ПЛР, передбачає ампліфікацію - чисельне копіювання ділянки ДНК, яка обмежена двома різноспрямованими олігонуклеотидними праймерами, за допомогою термостабільної ДНК-полімерази [1]. Використання термостабільних ДНК-полімераз зробило метод ПЛР зручним та загальновживаним [2]. На сьогодні метод ПЛР широко використовується для ідентифікації та детекції видоспецифічних генів [3]. Для гену або ділянки ДНК, яка цікавить дослідників, підбирається пара праймерів, за допомогою якої можна провести ампліфікацію ділянки характерної довжини і отримати амплікони, зручні для ідентифікації після розділення методом електрофорезу в агарозному гелі [4]. Відомі праймери для ідентифікації та детекції нуклеїнової кислоти штамів культур Lactococcus lactis subsp. lactis та Lactococcus lactis subsp. cremoris не є високоспецифічними, оскільки наявний високий рівень подібності між послідовностями нуклеїнових кислот цих штамів. Крім того, ці праймери були підібрані для аналізу полімеразної ланцюгової реакції із проксимальної ділянки фрагмента гену 16S рибонуклеїнової кислоти, скорочено - 16S РНК [5]. Аналіз ДНК методом ПЛР може бути використаний для визначення генотипу культур Lactococcus lactis subsp. lactis та Lactococcus lactis subsp. cremoris до рівня підвидів. Такий підхід не забезпечує технічну простоту і точність диференціації культур і відомі праймери для гену gadB не дали чіткого визначення окремих підвидів Lactococcus lactis [6]. Аналіз ДНК за шістьма парами праймерів до генів Lactococcus lactis , використаний для визначення генотипу до рівня підвидів, є громіздким, не забезпечує точність ідентифікації культур, не дає чіткого визначення окремих підвидів культур Lactococcus lactis subsp. lactis та Lactococcus lactis subsp. cremoris [7]. Штами молочнокислих бактерій - важливий біотехнологічний об'єкт, оскільки вони широко застосовуються при виробництві ферментованих харчових продуктів: йогуртів, просто к ваш і, ряжанки, м'яких, розсільних сирів, тощо; деякі штами молочнокислих бактерій мають пробіотичні властивості і використовуються в складі лікарських препаратів, БАД, продуктів функціонального харчування для людини і кормових добавок для тварин. Оскільки за морфологічними та біохімічними ознаками культури Lactococcus lactis можуть мати однакові властивості за ними складно провести їх підвидову належність. Для точної диференціації культур методом полімеразної ланцюгової реакції використовують пари олігонуклеотидних праймерів. Праймери для визначення культур Lactococcus lactis subsp. lactis та Lactococcus lactis subsp. cremoris методом полімеразної ланцюгової реакції є об'єктом даного винаходу. Запропоновані праймери можуть бути застосовані при дослідних та виробничих роботах з ідентифікації молочнокислих бактерій Lactococcus lactis subsp. lactis та/або Lactococcus lactis subsp. cremoris для ідентифікації ДНК культур Lactococcus lactis subsp. lactis та/або Lactococcus lactis subsp. cremoris . у бактеріальнихпрепаратах та ферментованих харчових продуктах методом полімеразної ланцюгової реакції. Праймери для визначення ДНК культур Lactococcus lactis subsp. lactic та Lactococcus lactis subsp. cremoris у бактеріальних препаратах та ферментованих харчових продуктах методом 1 UA 107547 C2 полімеразної ланцюгової реакції, для визначення ДНК культур Lactococcus lactis subsp. lactis та Lactococcus lactis subsp. cremoris , застосовують пари олігонуклеотидних праймерів до гену recN DNA repair protein: прямий праймер 5 та зворотній праймер - для ампліфікації 790 bp фрагмента ДНК культур Lactococcus lactis subsp. lactis та Lactococcus lactis subsp. cremoris ; прямий праймер та зворотній - для ампліфікації 352 bp фрагмента ДНК культури Lactococcus lactic subsp. lactis ; 10 прямий праймер та зворотній праймер - для ампліфікації 529 bp фрагмента ДНК культури Lactococcus lactis subsp. cremoris . 15 20 25 При цьому прямий праймер підібраний таким чином, що є уніфікованим для визначення кожного фрагмента ДНК гену recN repair protein RecN Lactococcus, а зворотні - визначають фрагмент ДНК характерної довжини для кожного підвиду. Запропоновані праймери для визначення культур Lactococcus lactis subsp. lactis та Lactococcus lactis subsp. cremoris методом полімеразної ланцюгової реакції застосовують при дослідних та виробничих роботах з ідентифікації молочнокислих бактерій Lactococcus lactis subsp. lactis та/або Lactococcus lactis subsp. cremoris для ідентифікації ДНК культур Lactococcus lactis subsp. lactis та/або Lactococcus lactis subsp. cremoris у бактеріальних препаратах та ферментованих харчових продуктах методом полімеразної ланцюгової реакції. Для визначення ДНК культур Lactococcus lactis subsp. lactis та/або Lactococcus lactis subsp. cremoris , застосовують пари олігонуклеотидних праймерів: прямий праймер та зворотній праймер - для ампліфікації 790 bp фрагмента ДНК культур Lactococcus lactis subsp. lactis та Lactococcus lactis subsp. cremoris ; прямий праймер та зворотній праймер - для ампліфікації 352 bp фрагмента ДНК 30 культури Lactococcus lactis subsp. lactis ; прямий праймер 35 40 45 50 55 та зворотній праймер - для ампліфікації 529 bp фрагмента ДНК культури Lactococcus lactis subsp. cremoris . Підбір праймерів проводили згідно правил молекулярного дизайну. Для підбору праймерів використовувались програми Primer, Oligo, або їх аналоги, які дозволяють здійснювати багатофакторний аналіз вибраних послідовностей ДНК. Порівняльний аналіз вибраних олігонуклеотидних праймерів здійснювали з допомогою програми Blast (http://www.ncbi.nlm.nih.gov/blast) для виключення фрагментів гомологічних до ДНК споріднених та неспоріднених видів. Геномний аналіз проводився за повними послідовностями геномів бактерій, доступних в GenBank, на основі гомології до нуклеотидної послідовності генів молочнокислих бактерій за допомогою програми Blast. Досліджувані організми відносяться до: cellular organisms; Bacteria; Firmicutes; Bacilli; Lactobacillales; Streptococcaceae; Lactococcus; Lactococcus lactis . Для визначення культур Lactococcus laclis subsp. lactis та Lactococcus lactis subsp. cremoris обрали ген репарації ДНК білків recN (англ. - DNA repair protein recN): Lactococcus lactis subsp. lactis ІІ1403 chromosome, complete genome за даними GenBank per. № NC_002662.1 (фрагмент від 883095 р до 884762 р довжиною 1668 bp), див. Фігура 1. "Послідовність recN DNA repair protein Lactococcus lactis subsp. lactis II1403 chromosome"; Lactococcus lactis subsp. cremoris MG1363 chromosome, complete genome за даними GenBank per. № NC_009004.1 (фрагмент від 1660156 р до 1661823 р довжиною 1668 п.н. bp), див. Фігура 2. "Послідовність recN DNA repair protein Lactococcus lactis subsp. cremoris MG1363 chromosome". Нуклеотидні послідовності гену recN (англ. DNA repair protein) підвидів Lactococcus lactis subsp. lactic та Lactococcus lactis subsp. cremoris відрізняються на певних ділянках 2 UA 107547 C2 послідовностей ДНК [http://www.ncbi.nlm.nih.gov/blast/Blast.cgil. Таким чином, за цими ділянками гену recN DNA repair protein можна точно встановити приналежність до підвиду lactis або cremoris культури всередині виду Lactococcus lactis . 5 3 UA 107547 C2 4 UA 107547 C2 5 10 15 Для ідентифікації ДНК Lactococcus lactis subsp. lactis або Lactococcus lactis subsp. cremoris , культури яких використовують при дослідних та виробничих роботах з ідентифікації молочнокислих бактерій Lactococcus lactis subsp. lactis та/або Lactococcus lactis subsp. cremoris для ідентифікації ДНК культур Lactococcus lactis subsp. lactis та/або Lactococcus lactis subsp. cremoris у бактеріальних препаратах та ферментованих харчових продуктах методом полімеразної ланцюгової реакції застосовують пари праймерів. Праймери для гену recN DNA repair protein культур Lactococcus lactis subsp. lactis 111403 chromosome, complete genome та/або Lactococcus lactis subsp. cremoris MG1363 chromosome, complete genome - для ампліфікації 790 bp фрагмента ДНК Lactococcus lactic subsp. lactic та Lactococcus lactis subsp. cremoris : прямий праймер діє з 586 р, зворотній праймер діє з 1375 р. Розмір послідовності: 1668 bp. 5 UA 107547 C2 Розмір фрагмента: 790 bp 5 Праймери для гену recN DNA repair protein Lactococcus lactis subsp. lactis 111403 chromosome, complete genome, для ампліфікації 352 bp фрагмента ДНК прямий праймер діє праймер Розмір послідовності: 1668 bp. з 586 20 bp діє з 937 р. 10 Розмір фрагмента: 352 bp 6 р, зворотній UA 107547 C2 Праймери для гену recN DNA repair protein Lactococcus lactis subsp. cremoris MG1363 chromosome, для ампліфікації 529 bp фрагмента ДНК прямий праймер 5 діє з 586 р, зворотній праймер Розмір послідовності: 1668 bp. діє з 1114 р. Розмір фрагмента: 529 bp 10 Для перевірки запропонованих пар праймерів: прямий праймер 15 20 та зворотній праймер - для ампліфікації 790 bp фрагмента ДНК культур Lactococcus lactic subsp. lactis та/або Lactococcus lactis subsp. сremoris; прямий праймер та зворотній праймер - для ампліфікації 352 bp фрагмента ДНК культури Lactococcus lactis subsp. lactis 111403 chromosome; прямий праймер та зворотній праймер , комплементарні до гену recN DNA repair protein для ампліфікації 529 bp фрагмента ДНК культури Lactococcus lactis subsp. cremoris MG1363 chromosome проводили полімеразну ланцюгову реакцію. Приклад 1 7 UA 107547 C2 Для проведення паралельних полімеразних ланцюгових реакцій використовували термостабільну Taq ДНК полімеразу, відповідний десятикратний ПЛР-буфер, 25мМ MgCl2, розчини чотирьох дезоксирибонуклеотидтрифосфатів в об'ємі: Н2О 10Х буфер MgCl2 25 мМ dNTP 2 мМ Taq Праймер 1 F (10 пкМ/мкл) праймер 2 R (10 пкМ/мкл) праймер 3 R (10 пкМ/мкл) праймер 4 R (10 пкМ/мкл) проба ДНК 5 мкл 2,5 мкл 4 мкл 3,5 мкл 0,5 мкл 2,5 мкл, 2,5 мкл, 2,5 мкл, 2,5 мкл, 2 мкл, 25 мкл. 5 (- праймер 1 мкл, праймер 2 мкл, 10 15 20 25 30 35 40 45 (10пкМ/мкл) - 2,5 (10пкМ/мкл) - 2,5 праймер 3 (10пкМ/мкл) - 2,5 мкл, праймер 4 (10пкМ/мкл) - 2,5 мкл, проба ДНК - 2 мкл кожної). Для проведення полімеразної ланцюгової реакції з запропонованими праймерами використовували проби ДНК окремих культур та бактеріальних препаратів на їх основі. ПЛР 4 аліквоти проводили у апараті "GeneAmp PCR System 9600" з відповідним програмним забезпеченням. Параметри: початкова стадія денатурації ДНК при 95 °C впродовж 2 хвилини; 30 циклів, які складаються з: етапу при 95 °C впродовж 30 секунд, етапу при 56 °C впродовж 30 секунд, етапу при 72 °C впродовж 60 секунд; кінцева стадія елонгації при 72 °C впродовж 5 хвилин. Результати полімеразної ланцюгової реакції наведені в таблиці 1 "Якісне визначення ДНК за праймерами" та Фотографія 1. У кожній реакції використовували праймери з різними пробами ДНК: реакція № 1-3 ампліфікація ДНК чистих культур Lactococcus lactis subsp. lactis , та реакція № 4 при повній відсутності ДНК у пробі - вода; маркер. З кожної реакції відбирали по 10 мкл кінцевого продукту ПЛР і аналізували шляхом електрофорезу в агарозному гелі. Для розділення фрагментів ДНК отриманих під час проведення ПЛР використовується 1 % агарозний гель, який містить бромистий етидій для фарбування ампліконів і подальшої візуалізації результатів під дією ультрафіолетових променів. Продукти ПЛP являють собою амплікони фрагментів вибраних генів характерної довжини, що дозволяє розрізнити їх після електрофоретичного розділення в агарозному гелі: 790 bp специфічного фрагмента ДНК гену recN DNA repair protein культур Lactococcus lactis subsp. fact is; 352 bp специфічного фрагмента ДНК гену rеcN DNA repair protein культури Lactococcus lactis subsp. lactis . Результати електрофорезу наведені на Фотографії 1 "Амплікони після електрофоретичного розділення в агарозному гелі фарбовані бромистим етидієм при опроміненні ультрафіолетом". Результатом підбору праймерів є ампліфікація фрагментів 790 bp специфічного фрагмента ДНК гену recN DNA repair protein культур Lactococcus lactis subsp. lactis , 352 bp специфічного фрагмента ДНК гену recN DNA repair protein культури Lactococcus lactis subsp. lactis , що дозволяє визначити присутність геномної ДНК культури Lactococcus lactis subsp. lactis . Приклад 2 Для проведення паралельних полімеразних ланцюгових реакцій використовували термостабільну Taq ДНК полімеразу, відповідний десятикратний ПЛР-буфер, 25мМ MgCl2, розчини чотирьох дезоксирибонуклеотидтрифосфатів в об'ємі: 8 UA 107547 C2 Н2О 10Х буфер MgCl2 25 мМ dNTP 2 мМ Taq праймер F (10 пкМ/мкл) праймер R 2 (10 пкМ/мкл) праймер R 3 (10 пкМ/мкл) праймер R 4 (10 пкМ/мкл) проба ДНK 5 мкл, 2,5 мкл, 4 мкл, 3,5 мкл, 0,5 мкл, 2,5 мкл, 2,5 мкл, 2,5 мкл, 2,5 мкл, 2 мкл. 25 мкл. (- праймер 1 мкл, 5 10 15 20 25 30 35 40 45 (10пкМ/мкл) - 2,5 праймер 3 (10пкМ/мкл) - 2,5 мкл, праймер 4 (10пкМ/мкл) - 2,5 мкл, проба ДНК - 2 мкл кожної). Для проведення полімеразної ланцюгової реакції з запропонованими праймерами використовували проби ДНК окремих культур та бактеріальних препаратів на їх основі. ПЛР 4 аліквоти проводили у апараті "GenеAmp PCR System 9600" з відповідним програмним забезпеченням. Параметри: початкова стадія денатурації ДНК при 95 °C впродовж 2 хвилини; 30 циклів, які складаються з: етапу при 95 °C впродовж 30 секунд, етапу при 56 °C впродовж 30 секунд, етапу при 72 °C впродовж 60 секунд; кінцева стадія елонгації при 72 °C впродовж 5 хвилин. Результати полімеразної ланцюгової реакції наведені в таблиці 2 "Якісне визначення ДНК за праймерами" та Фотографія 2. У кожній реакції використовували праймери з різними пробами ДНК: Маркер, реакції № 1, № 4, ампліфікація ДНК чистих культур Lactococcus lactis subsp. I act is, реакція № 3ампліфікація ДНК з бакпрепарату суміші культур Lactococcus lactis subsp. lactis , Lactococcus lactis ssp. с re mo r is. та реакція № 2 - негативний контроль ДНK з бакпрепарату суміші культур Streptococcus salivarius ssp. thermophilus та Lactobacillus delbruecki ssp. bulgaricus. З кожної реакції відбирали по 10 мкл кінцевого продукту ПЛР і аналізували шляхом електрофорезу в агарозному гелі. Для розділення фрагментів ДНК отриманих під час проведення ПЛР використовується 1 % агарозний гель, який містить бромистий етидій для фарбування ампліконів і подальшої візуалізації результатів під дією ультрафіолетових променів. Продукти ПЛР являють собою амплікони фрагментів вибраних генів характерної довжини, що дозволяє розрізнити їх після електрофоретичного розділення в агарозному гелі: 352 bp специфічного фрагмента ДНК гену recN DNA repair protein культури Lactococcus lactis subsp. lactis ; 529 bp специфічного фрагмента ДНК гену recN DNA repair protein культури Lactococcus lactis subsp. cremoris . Результати електрофорезу наведені на Фотографії 1 "Амплікони після електрофоретичного розділення в агарозному гелі фарбовані бромистим етидієм при опроміненні ультрафіолетом". Результатом підбору праймерів є ампліфікація фрагментів 352 bp специфічного фрагмента ДНК гену recN DNA repair protein культури Lactococcus lactis subsp. lactis , 529 bp специфічного фрагмента ДНК гену recN DNA repair protein культури Lactococcus lactis subsp. cremoris , що дозволяє визначити присутність геномної ДНК культури Lactococcus lactis subsp. lactis або Lactococcus lactis subsp. cremoris , або засвідчити присутність ДНК обох культур одночасно. Бажаного результату досягають внаслідок використання для ПЛР розроблених пар праймерів для ампліфікації фрагментів цільових генів. Запропонованіпраймери: прямий праймер 9 UA 107547 C2 та зворотній праймер - для ампліфікації 790 bp фрагмента ДНК культур Lactococcus lactis subsp. lactis та Lactococcus lactis subsp. cremoris ; 5 прямий праймер та зворотній праймер - для ампліфікації 352 bp фрагмента ДНК культури Lactococcus lactis subsp. lactis ; прямий праймер 10 15 20 25 30 35 40 45 та зворотній праймер - для ампліфікації 529 bp фрагмента ДНК культури Lactococcus lactis subsp. cremoris . При цьому прямий праймер підібраний так, що є уніфікованим для визначення кожного фрагмента ДНК гену recN repair protein культури Lactococcus lactis subsp. lactis та/або Lactococcus lactis subsp. cremoris , а зворотні - визначають свій фрагмент ДНК характерної довжини для культури Lactococcus lactis subsp. lactis та Lactococcus lactis subsp. cremoris . Запропоновані праймери для визначення культур Lactococcus lactis subsp. lactis та Lactococcus lactis subsp. cremoris методом полімеразної ланцюгової реакції застосовують для дослідних та технологічних робіт у промисловості, кількісного визначення ДНК технологічних штамів молочнокислих бактерій для виробництва бактеріальних препаратів та ферментованих харчових продуктів, ідентифікації ДНК культур Lactococcus lactis subsp. lactis та/бо Lactococcus lactis subsp. cremoris . Рекомендовано використовувати праймери для визначення ДНК культур Lactococcus lactis subsp. lactic та Lactococcus lactis subsp. cremoris при проведенні моніторингу зразків бактеріальних препаратів, перевірки відповідності вмісту Lactococcus lactis subsp. lactis та/або Lactococcus lactis subsp. cremoris нормативним та супровідним документам на виробництві та у торгівельній мережі. Запропоновані праймери для визначення присутності культур Lactococcus lactis subsp. lactis та/або Lactococcus lactis subsp. cremoris у бактеріальних препаратах методом полімеразної ланцюгової реакції мають переваги у значній економії витрат часу та точності ідентифікації ДІІК культур Lactococcus lactis subs p. lactis та Lactococcus lactis subsp. cremoris методом полімеразної ланцюгової реакції. Джерела інформації: [1] Enzymatic amplification of beta-globin genomic sequences and restriction site analysis for diagnosis of sickle cell anemia/RK Saiki, S Scharf, F Faloona, KB Mullis, GT Horn, HA Erlich, and N Arnheim // Science, !985, v. 230, p. 1350-1354. [2] Primer-directed enzymatic amplification of DNA with a thermostable DNA polymerase / Saiki RK, Gelfand DH, Stoffel S, Scharf SJ, Higuchi R, Horn GT, Mullis KB, Erlich HA.// Science, 1988, v. 239, p. 487-491. [3] Species-Specific Identification of Penicillium Linked to Patulin Contamination/ DombrinkKurtzman MA, McGovern AE.// J Food Prot., 2007, v. 70(11), p. 2646-2650. [4] PGR Protocols/ Innis ct al.// Academic Press. Inc., 1990, p.219-227. [5] Quantitative PGR for DNA identification based on genome-specific interspersed repetitive elements/ Walker JA, Hughes DA, Hedges DJ, Anders BA, Laborde ME, Shewale J, Sinha SK, Batzer MA.// Genomics, 2004, v. 83 (3) p. 518-527. [6] Молекулярно-биологические подходы к отбору бактериальных культур при создании заквасок для биотехнологии. Ботина Светлана Геннадиевна, 14.03.2011. [7] Rapid PCR-based method which can determine both phenotypc and genotype of Lactococcus lactis subspecies./ Masaru Nomura, Miho Kobayashi, and Takashi Okamoto/ Appl Environ Microbiol. 2002 May; 68(5): 2209-2213. 50 10 UA 107547 C2 Таблиця 1 № реакції Проба ДНК Праймери * - не виявлено, не ідентифіковано. 11 Ампліфікат* UA 107547 C2 Таблиця 2 Якісне визначення ДНК за праймерами № реакції Проба ДНК Праймери Ампліфікат* * - не виявлено, не ідентифіковано. ФОРМУЛА ВИНАХОДУ 5 Спосіб визначення ДНК культури Lactococcus lactis subsp. cremoris у бактеріальних препаратах та ферментованих харчових продуктах методом полімеразної ланцюгової реакції, який відрізняється тим, що для визначення ДНК культур Lactococcus lactis subsp. cremoris, застосовують пару олігонуклеотидних праймерів до гену recN DNA repair protein: 10 прямий праймер та зворотній праймер - для ампліфікації 529 bp фрагмента ДНК культури Lactococcus lactis subsp. cremoris. 12 UA 107547 C2 Фіг.1 13 UA 107547 C2 продовження 1 Фігури 1 14 UA 107547 C2 продовження 2 Фігури 1 15 UA 107547 C2 продовження 3 Фігури 1 Продовження 3 Фігури 1 16 UA 107547 C2 Фігура 2 17 UA 107547 C2 продовження 1 Фігури 2 продовження 2 Фігури 2 18 UA 107547 C2 "Амплікони після електрофоретичного розділення в агарозному гелі фарбовані бромистим етидієм при опроміненні ультрафіолетом". Фотографія 1 19 UA 107547 C2 "Амплікони після електрофоретичного розділення в агарозному гелі фарбовані бромистим етидієм при опроміненні ультрафіолетом". Фотографія 2 Комп’ютерна верстка А. Крижанівський Державна служба інтелектуальної власності України, вул. Урицького, 45, м. Київ, МСП, 03680, Україна ДП “Український інститут промислової власності”, вул. Глазунова, 1, м. Київ – 42, 01601 20
ДивитисяДодаткова інформація
Автори англійськоюSemenivska Olena Anatoliivna
Автори російськоюСеменовская Елена Анатолиевна
МПК / Мітки
МПК: C12N 15/09
Мітки: cremoris, визначення, днк, subsp, культури, полімеразної, спосіб, ланцюгової, lactis, реакції, lactococcus, методом
Код посилання
<a href="https://ua.patents.su/22-107547-sposib-viznachennya-dnk-kulturi-lactococcus-lactis-subsp-cremoris-metodom-polimerazno-lancyugovo-reakci.html" target="_blank" rel="follow" title="База патентів України">Спосіб визначення днк культури lactococcus lactis subsp. cremoris методом полімеразної ланцюгової реакції</a>
Попередній патент: Спосіб визначення днк культури lactococcus lactis subsp. lactis методом полімеразної ланцюгової реакції
Наступний патент: Осьовий вентилятор
Випадковий патент: Автоматичний гранатомет